「RDKitによるコンフォマーの生成」という記事では,RDKitを用いたコンフォマーの生成方法について説明しました.
我々が興味のある分子は大抵いくつかの回転可能な結合を持っていることから,一連の配座異性体に関する情報が重要になります.
このように様々なコンフォマーを取得する試みを「コンフォメーション探索」と呼びます.計算化学における重要なトピックです.
コンフォメーション探索とは
コンフォメーション探索とは,多数存在する分子の安定配座のリストを取得し,最安定構造を中心としたコンフォマー間のエネルギー差を計算する試みと言えます.
具体的には下記のような手順を踏むことが多いです.
- 興味のある分子の構造をコンピュータ上で作成する
- 何らかの方法で分子のコンフォマーを大量に生成させる
- 全てのコンフォマーを分子力学法や半経験的分子軌道法などの低レベルの計算手法で構造最適化する
- 最適化構造を吟味し,同一コンフォマーを除外する
- 得られたコンフォマーに対して,より高精度の量子化学計算を用いて構造最適化・エネルギー計算を行う
場合によってはステップ3の前後で,ある閾値エネルギー以上の構造を存在比が低いと想定して削除することもあります.いずれにせよ,いかにして構造的に多様なコンフォマーを効率よく得るかが焦点になります.
コンフォメーション探索の大切さ
我々が興味のある分子は大抵いくつかの回転可能な結合を持っていることから,最安定構造以外にも複数の安定配座のアンサンブルとして存在しています.その存在比率は各配座異性体間のエネルギー差から,ボルツマン分布によって決まります.
そのため,計算化学による実験結果の考察を行う場合,ほぼ全ての場合において各コンフォマーの存在比を加味した上で考察を行うことが大切になります.
- NMRのケミカルシフト・カップリング定数を見積もる
- 化学反応の活性化エネルギーを見積もる
- ドッキング構造中に見られる配座とフリー体の安定配座の違いを考察する
など,全ての場合において分子のコンフォメーション探索が重要です.
コンフォメーション探索の難しさ
それではどのようにして分子のコンフォマーを求めていけばよいでしょうか?
例えば「QSARにおける立体因子の記述:Sterimolパラメータを用いた線形モデル」という記事で紹介したwSterimolスクリプトでは,二面角をスキャンしていくことでコンフォマーを生成させています.小さい分子では,このような網羅的な方法によるコンフォマーの探索が可能ですが,分子が大きくなるに連れて不可能になります.
例えば直鎖アルカン,CH3-(CH2)n+1-CH3,の配座異性体を考えてみましょう.対称性を加味しないとブタン(n=1)では1つのアンチ配座と2つのゴーシュ配座,合計3個のコンフォマーが考えられます.コンフォマーの数は回転可能なC–C結合が1つ増えるごとに3個増えますので,ヘキサン(n=3)では27個,オクタン(n=5)では243個と,3n で指数関数的に増加していきます.
このように,多くの分子において,全てのコンフォマーを調べ上げて最安定構造を見つけることは極めて困難です.
そのため,最安定構造を探したり,多数の安定構造を探し出す,「コンフォメーション探索」は計算化学における重要な研究分野になります.実際にコンフォメーション探索が行える計算ソフトはたくさん存在します.
コンフォメーション探索に用いられる手法
類似の初期構造から構造最適化を開始してしまうと同じ安定構造に収束してしまいますので,先にも述べたように「いかにしてばらけた初期構造を発生させるか」が重要になります.
例えば,
- モンテカルロ法
- 分子動力学(MD)法
- シミュレーティド・アニーリング法(焼きなまし法)
- 遺伝的アルゴリズム
- ディスタンス・ジオメトリー法
などの方法が挙げられます.本記事ではRDKitに実装されている改良ディスタンス・ジオメトリー法(ETKDG)を用いてコンフォメーション探索を行ってみます.
RDKitのディスタンス・ジオメトリー法を用いた探索
今回は「Platinum dataset」と呼ばれるハンブルク大の研究チームが高精度のX線構造から得た構造データセット中の1分子を用います.
なおPlatinum datasetに関しての詳細は以下の論文を参照してください.
「Benchmarking Commercial Conformer Ensemble Generators」(J. Chem. Inf. Model. 57, 11, 2719-2728.)
必要なライブラリのインポート
「RDKitによる3次元構造の生成」という記事ではAllChemモジュールのメソッドを用いて立体配座を作成しました.
この記事ではディスタンス・ジオメトリー法に特化したChem.rdDistGeomモジュールを利用してコンフォマー生成を行ってみたいと思います.(メソッドの中身は同じです)
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, rdDistGeom
from rdkit.Chem.Draw import IPythonConsole
import py3Dmol
import numpy as np
import pandas as pd
from sklearn.preprocessing import RobustScaler
from sklearn.cluster import DBSCAN
import matplotlib as mpl
import matplotlib.pyplot as plt
import seaborn as sns
sns.set()
%matplotlib inline
print('rdkit version: {}'.format(rdBase.rdkitVersion))
# rdkit version: 2019.09.3
suppl = Chem.SDMolSupplier('./platinum_dataset_2017_01.sdf', removeHs=False)
mols = [mol for mol in suppl if mol is not None]
print(len(mols)) # 4548
mol = mols[7]
以下のコードではデータセット中7番目の分子を用います.構造を眺めるとビアリールの二面角などに多数のコンフォマーが考えられそうです.
配座の発生
以下のコードでは
- 1000個のコンフォマーを発生させる
- 各コンフォマーをMMFFで最適化し,エネルギーを計算
- コンフォマー間のRMS行列を計算
- 重原子の座標をnumpy配列に格納,クラスタリング用に標準化
という順番で処理を行っています.
その際,AllChem.GetConformerRMSMatrixは下三角行列の中身を1次元のリストとして返すので,後のコンフォマー除去を容易にするように1000×1000のnumpy配列に格納しています.
またクラスタリング用の座標を格納する前にAllChem.AlignMolConformersを用いて,最初のコンフォマーを鋳型として整列しています.
## 1. コンフォマー発生
pm = rdDistGeom.ETKDGv2()
m_h = Chem.AddHs(m)
cids = rdDistGeom.EmbedMultipleConfs(m_h, 1000, pm)
print(m_h.GetNumConformers()) # 1000
## 2. MMFFによる最適化とエネルギー計算
energy = []
prop = AllChem.MMFFGetMoleculeProperties(m_h)
for cid in cids:
mmff = AllChem.MMFFGetMoleculeForceField(m_h, prop, confId=cid)
mmff.Minimize()
energy.append(mmff.CalcEnergy())
energy = np.array(energy)
## 3. RMS行列を計算
m = Chem.RemoveHs(m_h)
rms_mat = AllChem.GetConformerRMSMatrix(m)
rms_mat.append(0)
rms = np.zeros((1000,1000))
idx = 0
for i in range(1,1000):
rms[i][:i+1] = rms_mat[idx:i+idx+1]
idx += i
## 4. 重原子の座標をnumpy配列に格納
def genConfCoord(cid):
conf = m.GetConformer(cid)
coord = []
for atom in m.GetAtoms():
atom_idx = atom.GetIdx()
x,y,z = conf.GetAtomPosition(atom_idx)
coord.extend([x,y,z])
return np.array(coord)
AllChem.AlignMolConformers(m)
coord_array = np.zeros((len(cids), 3*m.GetNumAtoms()))
for i, cid in enumerate(cids):
coord_array[i] = genConfCoord(cid)
### クラスタリング用に標準化
scaler = RobustScaler()
scaler.fit(coord_array)
scaled_coord = scaler.transform(coord_array)
同一コンフォマーの除去
続いて最適化後に同一構造と見なせそうなコンフォマーを削除します.今回は
- 各原子平均でRMSが0.05Å未満
- エネルギー差が0.5 kcal/mol未満
を満たす構造を同一コンフォマーとします.絞り込みにはpandas.queryを使い,削除するコンフォマーのindexをdel_indexという集合で管理しています.
del_index = set()
for i in range(1000):
d = pd.DataFrame({'rms': rms[:,i], 'energy': energy})
d.energy = d.energy - d.energy[i]
del_index = del_index | set(d[i:].query('rms < 0.05 and
-0.5 < energy and
energy < 0.5').index)
d = d.drop(del_index)
scaled_coord = scaled_coord[d.index]
print(len(d)) # 737
263個のコンフォマーが除かれました.
クラスタリング
クラスタリング手法にも色々ありますが,今回はいくつのクラスタに分けるかを決め打ちせずにアルゴリズムに決めさせることとし,scikit-learnに実装されているDBSCANを使ってみます.
まずはepsとmin_samplesの値を変えていくことで,どれくらいのクラスターができるかを見てみます.
num_clusters = []
for e in np.arange(2.0,4.1,0.4):
for s in range(2,20,2):
db = DBSCAN(eps=e, min_samples=s)
classes = db.fit_predict(scaled_coord)
num_clusters.append([e,s,len(set(classes))])
ddf = pd.DataFrame(num_clusters)
ddf.columns = ['eps', 'min_samples', 'num_clusters']
ddf = pd.pivot_table(data=ddf, index=['eps'], columns=['min_samples'], values=['num_clusters'])
fig = plt.figure()
ax = fig.add_subplot(111)
sns.heatmap(ddf, vmin=3, vmax=80, annot=True, cmap='Reds')
ax.set_xlabel('min_samples')
ax.set_xticklabels(range(2,20,2), rotation=0)
ax.set_yticklabels(['{:.1f}'.format(i) for i in np.arange(2,4.1,0.4)])
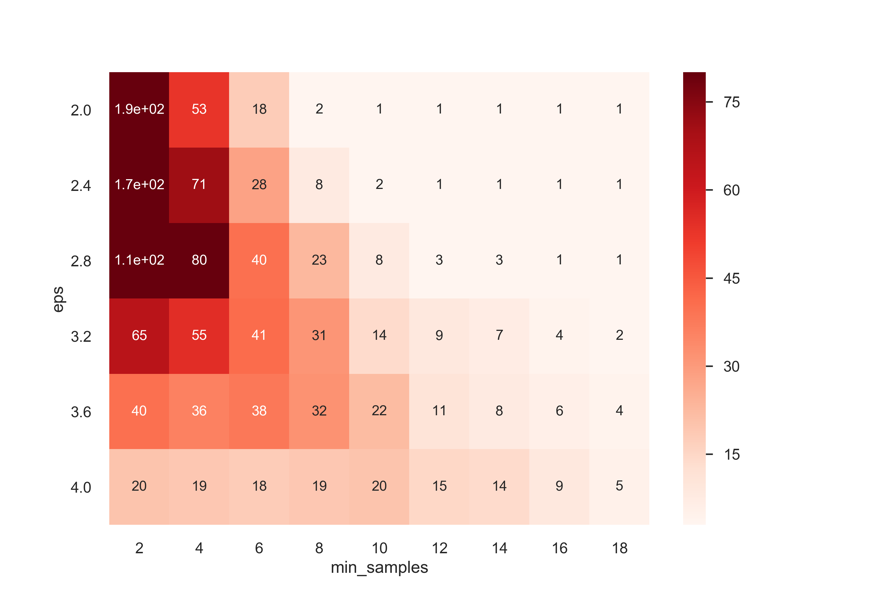
本来はChem.Lipinski.NumRotatableBondsなどで回転可能結合数を求め,その値に応じてクラスタの数を決めていくのがよいですが,ここでは可視化しやすいように18個のクラスターに分けられるeps=3.6, min_samples=4でやってみましょう.
dbscan = DBSCAN(eps=4.0, min_samples=6) clusters = dbscan.fit_predict(scaled_coord) pd.value_counts(clusters, sort=False)
DBSCANのクラスタ「-1」はどのクラスタにも属さないデータポイントになります.必然的に構造のばらつきが大きくなりますので,この後の高次計算を行う際にはこのクラスタからはサンプリング数を多めにしたほうがよいでしょう.
今回の場合はクラスター0番に分類される分子が多くなっています.
| class | count |
|---|---|
| 0 | 341 |
| 1 | 55 |
| 2 | 90 |
| 3 | 9 |
| 4 | 14 |
| 5 | 26 |
| 6 | 9 |
| 7 | 30 |
| 8 | 6 |
| 9 | 30 |
| 10 | 35 |
| 11 | 19 |
| 12 | 18 |
| 13 | 6 |
| 14 | 13 |
| 15 | 5 |
| 16 | 6 |
| -1 | 25 |
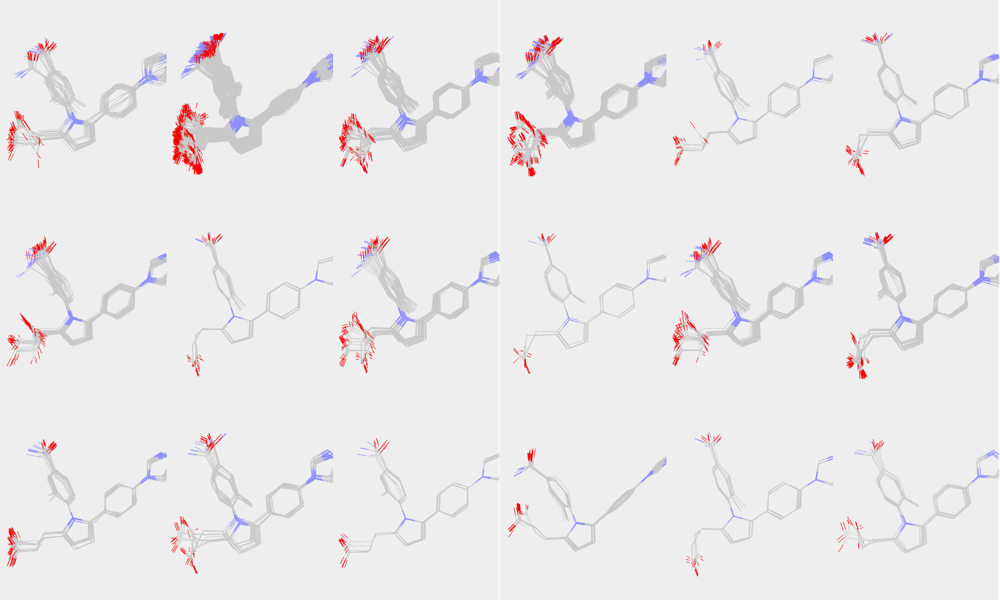
py3Dmolを用いた可視化
最後にpy3Dmolを用いて各クラスターを可視化してみましょう.
下のコードでは辞書型オブジェクトを用いて各クラスターに属するコンフォマーIDを管理しています.
d['group'] = clusters
db_classes = {}
for i in range(-1,17):
db_classes[i] = d[ d.group == i ].index
view = py3Dmol.view(width=1000, height=600, viewergrid=(3,6), linked=False)
for i, (j,k) in enumerate((m,n) for m in range(3) for n in range(6)):
for cid in db_classes[i-1]:
mb = Chem.MolToMolBlock(m, confId=cid)
view.addModel(mb, 'sdf', viewer=(j,k))
view.setStyle({'line': {}})
view.setBackgroundColor('0xeeeeee')
view.zoomTo()
view.show()
カルボニル周りがやや微妙ですが,クラスター数を絞った割にはまずまず揃っていそうでしょうか?
終わりに
今回の記事では「RDKitを用いたコンフォメーション探索:MMFFによる配座異性体生成とクラスタリング」という話題について,
- コンフォメーション探索がなぜ重要なのか
- コンフォメーション探索を行う方法
- RDKitのディスタンス・ジオメトリー法を用いたコンフォメーション探索
などについて説明してきました.
今回のコードをそのまま実戦投入するわけにはいかないでしょうが,本文中でも述べたように例えばk-平均法を用いてクラスタリングする際に,回転可能結合数に応じてクラスター数を変えるなどの処理を行うことで,サンプリング精度を上げていくことが可能です.
今回は構造最適化やエネルギー評価をRDKitに実装されているMMFFで行いました.実はMMFFで得られるポテンシャルエネルギー曲面(エネルギー値)はコンフォマー間でエネルギー差の大小を決められる精度にはないという報告もあります(PM7やB3LYP-D3によるエネルギー計算を推奨).
Ilana Y. Kanal John A. Keith Geoffrey R. Hutchison
Int. J. Quant. Chem., 2018, 118, e25512. (DOI: 10.1002/qua.25512)
次回は計算コストが高くなりますが,計算レベルを変えることによるエネルギーや最適化構造に与える影響について見ていきたいと思います.
コメント