pythonの代表的なケモインフォマティクスライブラリであるRDKitを用いて,これまで本ブログでは化合物の性質を表現するための分子記述子について学んできました.
- 「RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ」
- 「RDKitで立体的な分子の形を記述する」
- 「RDKitで化合物の芳香族度合を示す分子記述子を計算する」
- 「RDKitで薬らしさを定量的に評価する」
分子記述子は
- 分子内の芳香環の数
- 回転可能な結合の総数
といった分子の部分構造に関連するものから,
- 分子の脂溶性を表すLogP
- もっと抽象的な「薬らしさ」を表すQED
など化合物の物性や性質に関連するものまで色々あります.
これらの記述子は,基本的には分子の特徴の一部を切り出して表現しているに過ぎません.とは言っても例えば構造に関する記述子を多数まとめれば,分子全体の構造的特徴を網羅できそうなことは想像できると思います.
今回扱う分子フィンガープリントもそういったある一定のルールに基づいて分子全体を1つの表現型で表したものになります.人間1人1人の指紋が異なるように,異なる化合物間ではフィンガープリントは異なりますが,類似した化合物では似たものになるように設計されています.
「類似した化合物」の定義が場合によって異なりますので,データセットや目的によって最適なフィンガープリントは異なります.そのため様々なフィンガープリントが開発されています.
今回はRDKitに実装されているフィンガープリントについて簡単に見ていきながら,どのようにして類似度を評価するか,類似度をどのように可視化するかについて学んでいきます.

ライブラリと分子の準備
まずはいつものように必要なライブラリのimportを行っておきましょう.今回はある程度類似した化合物のセットが欲しいので,SDFにはfluorochemで検索したサリチル酸誘導体を使います.
import pandas as pd
from rdkit import rdBase, Chem, DataStructs
from rdkit.Avalon import pyAvalonTools
from rdkit.Chem import AllChem, Draw
from rdkit.Chem.Fingerprints import FingerprintMols
from rdkit.Chem.AtomPairs import Pairs, Torsions
print(rdBase.rdkitVersion) # 2020.09.3
suppl = Chem.SDMolSupplier('./salicylate_jan_2021.sdf')
mols = [mol for mol in suppl if mol is not None]
len(mols) # 431
分子類似性:なぜ似た化合物に興味があるのか?
我々がなぜ類似化合物に興味があるかというと,「類似した化合物は似た性質を持つ」というsimilar property principleというものに由来します.ある生理活性を持つ化合物と似た構造の化合物はやはり同じような生理活性を持つというわけです.この「法則」自体はこれまでの数多くの検証によって正当性が確かめられています.
問題は先ほども述べたように,何をもって似ているとするかは場合によって異なるので,万能な方法がないということになります.
フィンガープリントの作成方法
フィンガープリント作成には色々なルールがあると述べました.その一つとして,部分構造のを数え上げていくタイプを考えてみます.この場合,
- 「芳香環の数」=3
- 「窒素原子の数」=4
といった形である部分構造とその数をペアとした辞書型のデータができあがると考えられます.しかしこのままではコンピュータで処理をしにくく,類似度の評価などが困難です.
そこで得られた辞書型のデータにある処理を施すことで,長さ一定(1024や2048が多いです)のビット配列に0か1を埋め込んだ形式に変換を行うことが多いです.この作業のことをハッシュ化と呼びます.
これから実際にRDKitで実装されているメソッド名を紹介していきます.メソッド名に「GetHashedFingerprintAs’XXX’」といった記載があるものはHashed化されたフィンガープリントを‘XXX’の形で取得といった意味になります.大抵のメソッドは対象とするMolオブジェクトの他に,nBitsというビットの長さを指定する引数を取ることができます.
類似度の評価
ここまでの説明から,化合物の類似度をコンピュータを使って考えるにあたって,化合物のフィンガープリントが必要なことはわかっていただけたかと思います.それでは2つの化合物のフィンガープリントが与えられたときにどのようにして類似度を評価すればよいでしょうか?
困ったことにフィンガープリント同様に,様々な評価方法が提唱されていますが,最もよく使われている「タニモト係数」と呼ばれるものです.2つの分子AとBのビット配列フィンガープリントからタニモト係数を計算するに下記の式を使います.
$$ S_{AB} = \frac{c}{a + b – c} $$
ここで,
- aは分子Aのビット配列で1が立っている数
- bは分子Bのビット配列で1が立っている数
- cは分子AとBで共通に1が立っている数
を示します.タニモト係数はその定義から0から1の値を取ることになります.
下に示した仮想のフィンガープリントを使ってタニモト係数は計算してみましょう.
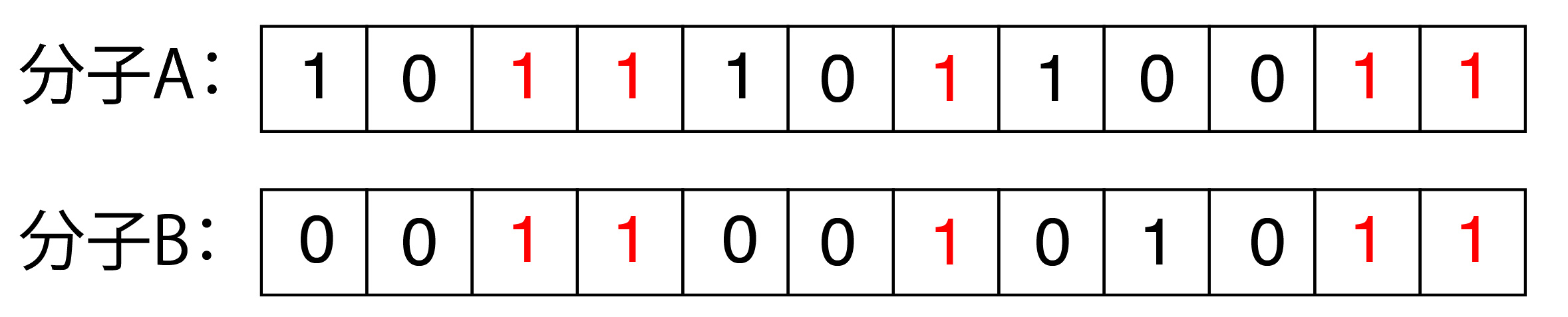
この場合,a=8,b=6,c=5(赤色の数字の数)となりますから,タニモト係数は
$$ S_{AB} = \frac{5}{8 + 6 – 5} = 0.56 $$
と求まります.
他によく使われる評価方法としてDice係数があって,こちらの定義式は
$$ S_{Dice} = \frac{2c}{a + b} $$
で表されます.a,b,cの定義はタニモト係数と同じです.
より一般的なTverskyインデックスというものがあり,こちらは
$$ S_{Tversky} = \frac{c}{\alpha (a-c) + \beta (b-c) + c} $$
になります.つまりタニモト係数は「α=β=1」の場合,Dice係数は「α=β=0.5」の場合に相当します.
タニモト係数やDice係数は例えばZINCデータベースで類似度構造検索する際に使われています.
RDKitでのフィンガープリントを用いた類似度評価の行い方
DataStructs.FingerprintSimilarity(fp1, fp2, metric)
DataStructs.TanimotoSimilarity(fp1, fp2)
DataStructs.DiceSimilarity(fp1, fp2)
・まとめて計算
DataStructs.BulkTanimotoSimilarity(refFP, FP_list)
DataStructs.BulkDiceSimilarity(refFP, FP_list)
フィンガープリントの比較にはrdkit.DataStructsモジュールにあるメソッドを使います.
分子1つずつを比較する場合にはFingerprintSimilarityを用います.その際にmetricを指定することでどの基準で比較するかを選択できます.デフォルトではタニモト係数で比較されますが,他にもいくつかの方法を選択可能です.
また各metricごとに専用のメソッドも用意されていて,
- タニモト係数で計算したい場合にはTanimotoSimilarity
- Dice係数を計算したい場合にはDiceSimilarity
を使います.
ある分子のフィンガープリントに対する,一連の分子に対する類似度を一挙に計算したい場合が多々あります.こういった場合にはBulk‘XXX’メソッドを使うことで計算可能です.具体的には後ほどコードを使って見ていきます.
RDKitに実装されているフィンガープリントのまとめ
MACCS Keys
AllChem.GetMACCSKeysFingerprint(mol)
ケモインフォマティクスでは非常に有名なMDL社の開発した化学構造データベースに由来するフィンガープリントです.
全部で166の部分構造についての有無を調べ上げたもので,RDKit内の情報保持のために1ビット使っているため全部で167ビットのフィンガープリントになります.部分構造を有する場合は1が,ない場合は0が格納されています.
maccs_fps = [AllChem.GetMACCSKeysFingerprint(mol) for mol in mols] maccs = DataStructs.BulkTanimotoSimilarity(maccs_fps[0], maccs_fps[1:])
Topologicalフィンガープリント (RDKitフィンガープリント)
ハッシュ化フィンガープリントの中では有名なDaylightフィンガープリントに類似したものと考えてよいです.RDKitフィンガープリントとも呼ばれています.
一定の結合数に相当する原子と結合種類を格納する方法で,事前に部分構造を用意する必要がないことから,より柔軟に分子構造を表現することが可能です.
rdkit_fps = [Chem.Fingerprints.FingerprintMols.FingerprintMol(mol) for mol in mols] rdkit = DataStructs.BulkTanimotoSimilarity(rdkit_fps[0], rdkit_fps[1:])
Morganフィンガープリント (Circularフィンガープリント)
AllChem.GetMorganFingerprint(mol, radius)
AllChem.GetMorganFingerprintAsBitVect(mol, radius, nBits)
・FCFP相当
AllChem.GetMorganFingerprint(mol, radius, useFeatures=True)
原子からある距離にある部分構造を数え上げていく,「circular substructures」と呼ばれるタイプのフィンガープリントです.radiusの値で距離を設定します.いわゆるECFP(Extended Connectivity Fingerprint)フィンガープリントに相当するものですが,探索距離の定義が異なる点に注意が必要です.
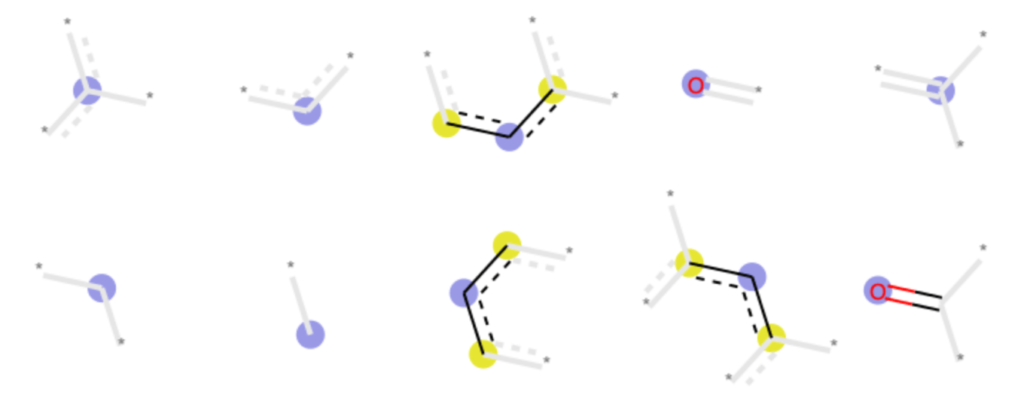
RDKitでは対象とする原子からの一定結合距離を「半径(radius)」として探索しますが,ECFPでは「直径(diameter)」で距離を考慮しています.例えば下のアセトフェノンの芳香環上の置換基を有する炭素(青色)に着目した場合,そこから結合1つ,結合2つの場合の「部分構造」を示したものが下の図になります.

よく使われるECFP4はradius=2の設定(デフォルト)にほぼ相当します.FCFP(Functional Connectivity Fingerprint)様のフィンガープリントを用いたい場合にはuseFeatures=Trueを設定します.
“When comparing the ECFP/FCFP fingerprints and the Morgan fingerprints generated by the RDKit, remember that the 4 in ECFP4 corresponds to the diameter of the atom environments considered, while the Morgan fingerprints take a radius parameter. So the examples above, with radius=2, are roughly equivalent to ECFP4 and FCFP4.”
また標準のGetMorganFingerprintでは非常に長いフィンガープリントが得られてきますので,ビット長を指定したGetMorganFingerprintAsBitVectを用いる方が一般的です.あまりに小さいビット長ですと衝突が問題になると予想されるため,1024以上の長さが使われることが多いです.
Morganフィンガープリントはこれから最も頻繁に使っていくフィンガープリントになると思います.
morgan_fps = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048) for mol in mols] morgan = DataStructs.BulkTanimotoSimilarity(morgan_fps[0], morgan_fps[1:])
MinHashフィンガープリント
MHFPEncoder.EncodeMol(mol)
MHFPEncoder.EncodeMolsBulk(mols)
2020.03のアップデータから利用可能になったフィンガープリントです.pipでインストール可能なMHFPライブラリと同様の構成で,smilesではなく,RDKitのMOLオブジェクトからフィンガープリントが生成できるように実装されています.
「A probabilistic molecular fingerprint for big data settings」という論文で報告されている,ECFPと同様にCircular型のフィンガープリントになります.Chem.rdMHFPFingerprintモジュールに実装されています.
import numpy as np from rdkit.Chem import rdMHFPFingerprint encoder = rdMHFPFingerprint.MHFPEncoder() m = mols[0] mhfp = encoder.EncodeMol(m) len(np.array(mhfp)) # 2048
Avalonフィンガープリント
rdkit.Avalon.pyAvalonTools.GetAvalonCountFP(mol)
RDKitから用いることのできるAvalon Chemoinformatics TookKitにもフィンガープリントが実装されています.
avalon_fps = [pyAvalonTools.GetAvalonFP(mol) for mol in mols] avalon = DataStructs.BulkTanimotoSimilarity(avalon_fps[0], avalon_fps[1:])
アトムペアフィンガープリントとトポロジカル二面角フィンガープリント
Chem.AtomPairs.Torsions.GetTopologicalTorsionFingerprint(mol)
アトムペアを用いるフィンガープリントでは
のように,全ての原子同士について原子のアトムタイプとその最小結合距離を記録していきます.
アトムタイプには
- 原子の種類
- 結合する重原子の数
- π電子数
の情報が記録されます.アトムペアフィンガープリントは分子全体の情報を効率的に取り込むことができるフィンガープリントになります.
トポロジカル二面角フィンガープリントは,二面角を形成する4連続原子について,上記アトムタイプの情報を用いて表記していくフィンガープリントです.
これらフィンガープリントについては「アトムペアやドナーアクセプターペアフィンガープリントは分子の形状を捉えた化学表現」という記事でさらに詳しく解説しています.参照してみてください.

ドナーアクセプターペアフィンガープリント
Chem.AtomPairs.Sheridan.GetBTFingerprint(mol)
ドナーアクセプターペアフィンガープリントでは「アトムタイプの情報」について
- 水素結合供与体
- 水素結合受容体
といった一般性のある「化学的性質」を情報として用いているフィンガープリントです.
原子を化学的性質の異なる7種類に分類した後,アトムペアフィンガープリントやトポロジカル二面角フィンガープリントと同様にして原子の情報をコードしていきます.
各フィンガープリントの比較
ここではRDKit,MACCS,Morgan,Avalonの各フィンガープリントについて相関を見てみましょう.先に求めた最初の分子のフィンガープリントに対するタニモト係数を使って比べてみます.
df = pd.DataFrame({'RDKit': rdkit,
'Avalon': avalon,
'MACCS': maccs,
'Morgan': morgan})
df.corr().round(2)
| RDKit | Avalon | MACCS | Morgan | |
|---|---|---|---|---|
| RDKit | 1.00 | 0.59 | 0.52 | 0.36 |
| Avalon | 0.59 | 1.00 | 0.75 | 0.68 |
| MACCS | 0.52 | 0.75 | 1.00 | 0.41 |
| Morgan | 0.36 | 0.68 | 0.41 | 1.00 |
同じ分子を同じ方法で類似度を比較しても,用いるフィンガープリントによって随分と値がことなることが見て取れると思います.
フィンガープリントの扱い方
BitVectオブジェクト(ExplicitBitVect)の扱い方
BitVect.GetNumOffBits()
BitVect.GetNumOnBits()
BitVect.GetOnBits()
BitVect.GetBit(idx)
フィンガープリントのビット配列はExplicitBitVectオブジェクトと呼ばれ,
- ビット配列の長さ:GetNumBits
- 1が立っているビットの数:GetNumOnBits
- 1が立っているビットの位置:GetOnBits
などの情報が取得可能です.
下の例ではMorganフィンガープリントを用いてデフォルトの長さと,長さ2048のビット配列へと短縮した場合について長さを求めています.デフォルトのMorganフィンガープリントはExplicitBitVectオブジェクトではないので,メソッド名(GetLength)が若干異なりますが意味は通じると思います.
morgan_default = AllChem.GetMorganFingerprint(mols[0], 2) morgan_bitvect = AllChem.GetMorganFingerprintAsBitVect(mols[0], 2, 2048) print(morgan_default.GetLength(), morgan_bitvect.GetNumBits())
(4294967295, 2048)
フィンガープリントのnumpy配列への変換
先に述べたように,フィンガープリントはExplicitBitVectオブジェクトとして扱われており,このままでは機械学習モデルの入力ベクトルとしては扱いにくい形式になっています.
DataStructs.ConvertToNumpyArrayメソッドを用いることで,フィンガープリントオブジェクトをnumpy配列へと変換することが可能です.このメソッドでは
- フィンガープリントを格納するnumpy配列を用意する
- フィンガープリントを用意した配列に書き込む
という2段階を経て変換作業を行います.
import numpy as np ar = np.zeros(2048) DataStructs.ConvertToNumpyArray(morgan_bitvect, ar) print(np.nonzero(ar))
(array([ 25, 80, 94, 114, 145, 213, 322, 329, 487, 500, 650,
695, 718, 807, 809, 841, 875, 906, 936, 980, 1057, 1121,
1152, 1380, 1452, 1453, 1501, 1744, 1750, 1873, 1917, 1928, 1970,
1991]),)
化合物の類似性を可視化する
各ビットの構造情報を可視化
RDKitの2018.09のアップデートから,RDKitフィンガープリントやMorganフィンガープリントのビット配列を可視化するコードが追加されました.このコードを用いると下のように,各ビットがどのような化学的な意味を表しているかを掴むことが可能になります.
この可視化方法については,「RDKitでフィンガープリントの可視化」という記事で具体的に解説していますので,参照してみてください.
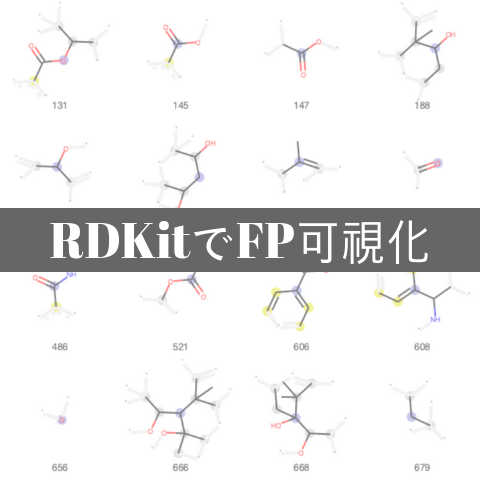
原子ごとの寄与率を用いた可視化方法
SimilarityMaps.GetSimilarityMapForFingerprint(refMol, prbMol, fp_F)
・ウェイトの絶対値を利用
SimilarityMaps.GetAtomicWeightsForFingerprint(refMol, prbMol, fp_F)
SimilarityMaps.GetSimilarityMapFromWeights(mol, weight)
アトムペア,二面角,そしてMorganフィンガープリントを用いた場合には,原子ごとに類似度の寄与率を可視化することが可能です.寄与度を規格化する場合と,しない場合の2通りの方法が選択可能です.
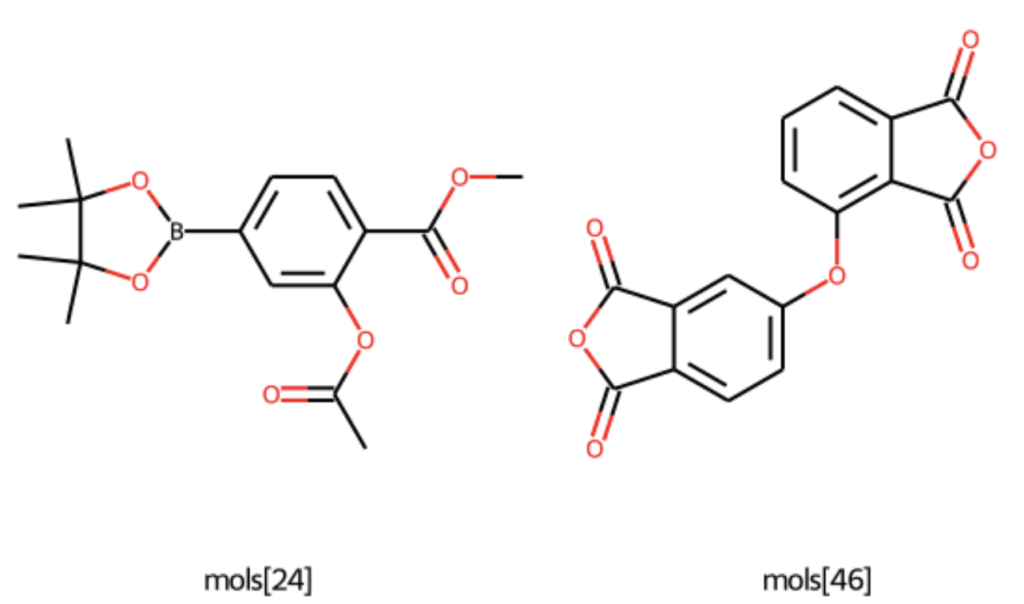
下のコードでは,ランダムに選んだmols[24]とmols[46]を用いて,Morganフィンガープリントへの各原子の寄与度を可視化してみます.
Draw.MolsToGridImage([mols[24], mols[46]], subImgSize=(300,400), legends=['mols[24]', 'mols[46]']) from rdkit.Chem.Draw import SimilarityMaps weight = SimilarityMaps.GetAtomicWeightsForFingerprint(mols[24], mols[46], SimilarityMaps.GetMorganFingerprint) fig = SimilarityMaps.GetSimilarityMapFromWeights(mols[24], weight, size=(400,400))
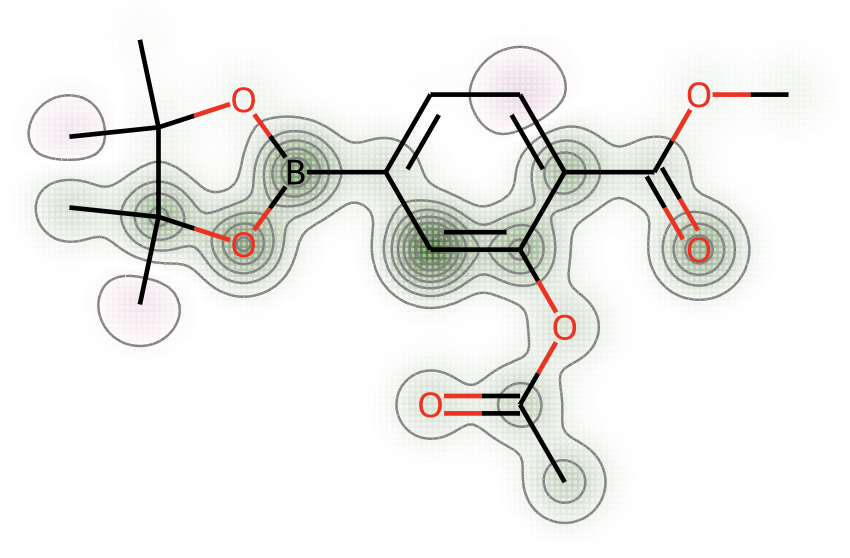
当然ですがサリチル酸部位の寄与が大きくなっています.
終わりに
今回は分子の個々の性質を表現する記述子の考え方をさらに発展させることで,分子全体の特徴を表すフィンガープリントを扱いました.さまざまなフィンガープリントがあることを学び,ビット文字列で表されたフィンガープリントを比較することで,分子の類似度を評価しました.さらに類似分子では実際にどの構造が類似しているかを知るために,フィンガープリントにおける原子毎の寄与率を求め可視化しました.
なお今回は扱いませんでしたが,RDKitには2Dファーマコフォアによるフィンガープリントも実装されています.こちらはファーマコフォアを扱う際に触れる予定です.
今回求めた分子同士の類似度は,分子全体の類似になります.次回は分子同士の部分構造に着目し,共通構造を探し出す手法,Maximum Common Subgraph (Substructure)を扱います.次々と新しいコンセプトが出てきますが,その分できることがどんどん増えていっています.焦らずに,地に足をつけて学習していきましょう.
>>次の記事:「RDKitを用いた部分構造検索とMCSアルゴリズム」

コメント