「類似した化合物は似た性質を持つ(similar property principle)」ことから,我々は類似化合物に興味があります.そのためケモインフォマティクスでは化合物の類似性を評価する様々な手法が開発されてきました.
もっともよく使われる手法は,「RDKitでフィンガープリントを使った分子類似性の判定」という記事で説明したフィンガープリントを用いる方法です.また「RDKitでFraggleを用いた化合物の類似度評価」という記事では,Fraggleという異なる手法の解説を行いました.
フィンガープリントにしろFraggleにしろ,分子のグラフ構造を基準とした2次元の結合情報に基づく評価方法です.一方で,化合物が生理活性などの機能を発揮する際には3次元構造が重要となることから,立体構造に基づいた類似の評価方法も重要となると考えられます.
今回取り上げるESP-Simは分子の電子的性質の類似性を評価する手法になります.
ESP-Sim自体のコードはgithubにあります.また2022年6月に行われた開発者のEsther Heid博士によるワークショップの動画(2時間程度)がYouTubeに上がっており,理解を深めることができます.
分子の形の類似度
官能基などの分子のグラフ構造に基づいた部分構造ではなく,立体的な分子の形の類似度を求めたい場合,対象分子のVan der Waals表面の重なりに基づいて評価する方法が考えられます.RDKitにはShape Tanimoto距離を評価するShapeTanimotoDistが実装されています.
距離ではなく類似度を求めたい場合は「1 – 距離」とすればよいです.
注意点としては,このメソッドでは与えられた座標に基づいて重なりを判定します.すなわち,比較する分子を予め共通構造などで揃えておかないと正しい距離を見積もることができません.
ESP-Simの使い方
Shape Tanimoto類似度は分子の形のみに着目した手法ですので,電子的性質は全く考慮されていません.ESP-Simではクーロンポテンシャルの重なりに基づいた類似度評価を行いますので,より電子的性質の近い分子が高い類似度になると期待できます.
ESP-Simのインストール
現時点ではgithubにあるyamlファイルを用いて新しいconda環境を作成する方法が推奨されています.
conda env create -f environment.yml
espsimの依存性は
- rdkit
- numpy
- scipy
- scikit-learn
などです.他にも電荷の計算方法次第ではpsi4などが必要となってきます.
今回はGoogle Colab上にgithubからインストールしていきます.
!pip install git+https://github.com/hesther/espsim
espsim以外にもpsi4,rdkit,py3dmolなどをインストールしてあります.今回用いた環境は以下の通りです.
import sys
import numpy as np
import rdkit
from rdkit import Chem
from rdkit.Chem import AllChem, Draw, rdMolAlign, rdDetermineBonds
from rdkit.Chem.Draw import IPythonConsole
import psi4
import py3Dmol
from espsim import GetEspSim, GetShapeSim
print(f'python version:\n{sys.version}')
print(f'numpy version: {np.__version__}')
print(f'rdkit version: {rdkit.__version__}')
print(f'psi4 version: {psi4.__version__}')
python version: 3.8.16 (default, Dec 7 2022, 01:12:13) [GCC 7.5.0] numpy version: 1.21.6 rdkit version: 2022.09.3 psi4 version: 1.7

ESP-Sim計算の具体例
espsim.GetShapeSim(prbMol, refMol)
espsimにはいくつかの方法が実装されていますが,もっとも直接的なメソッドがGetEspSimです.このメソッドでは立体的に重ね合わせ済みの2つのRDKitのMolオブジェクト(prbMolとrefMol)を引数として取り,その電子的類似度を返します.
なおGetShapeSimは「1 – ShapeTanimoto距離」を返すメソッドです.
ここでは5種類の一置換ベンゼン化合物に対して,クロロベンゼンに対するShape Tanimoto及びESP-Simの類似度を求めてみます.
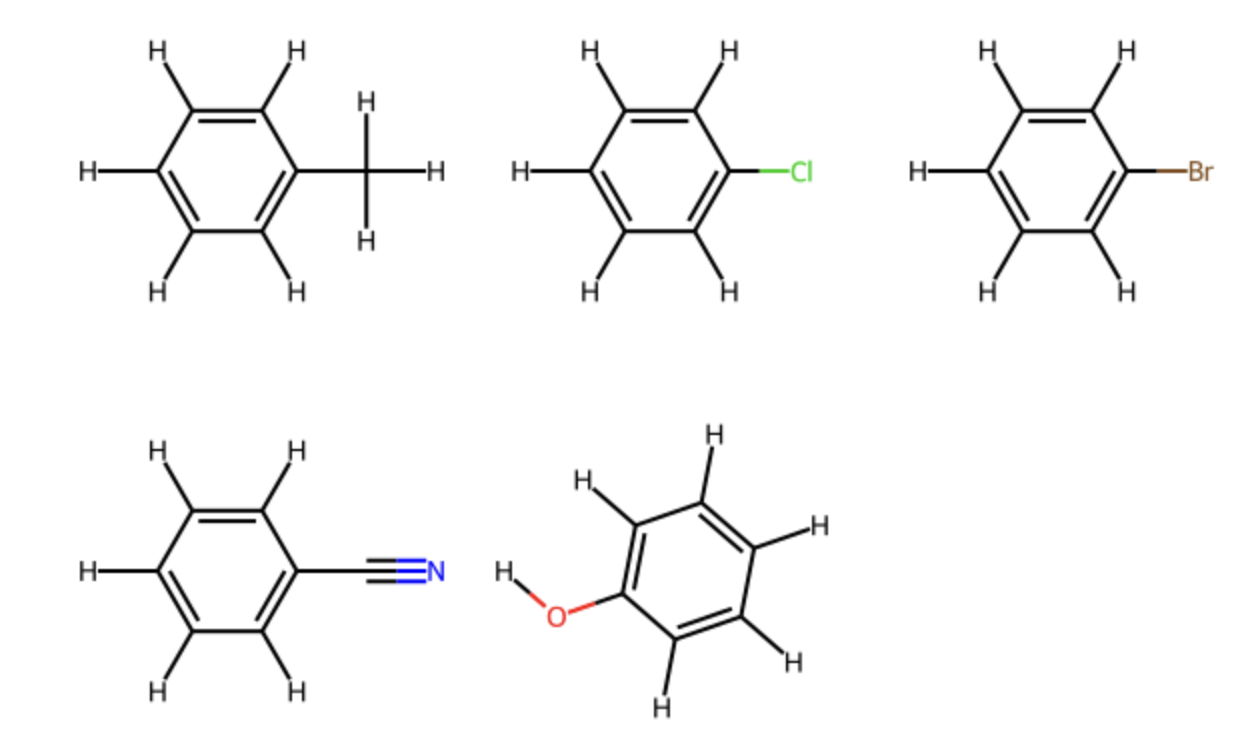
# 分子の準備
methyl = Chem.AddHs(Chem.MolFromSmiles('Cc1ccccc1'))
chloro = Chem.AddHs(Chem.MolFromSmiles('Clc1ccccc1'))
bromo = Chem.AddHs(Chem.MolFromSmiles('Brc1ccccc1'))
cyano = Chem.AddHs(Chem.MolFromSmiles('N#Cc1ccccc1'))
oh = Chem.AddHs(Chem.MolFromSmiles('Oc1ccccc1'))
# ベンゼンを鋳型に設定
core = Chem.MolFromSmiles('c1ccccc1')
refMatch = chloro.GetSubstructMatch(core)
AllChem.EmbedMolecule(chloro, AllChem.ETKDGv3())
# クロロベンゼンを基準に4化合物を比較対象として設定
prbMols = [methyl, bromo, cyano, oh]
prbNames = ['methyl', 'bromo', 'cyano', 'phenol']
print('Similarities with chlorobenzene')
print('prbMol\tShape-Sim\t Esp-Sim')
for prb, prb_name in zip(prbMols, prbNames):
#立体構造準備し,クロロベンゼンと重ね合わせ
AllChem.EmbedMolecule(prb, AllChem.ETKDGv3())
prbMatch = prb.GetSubstructMatch(core)
AllChem.AlignMol(prbMol=prb,
refMol=chloro,
atomMap=list(zip(prbMatch, refMatch)))
# ShapeSimとESPSimの計算
shape_sim = GetShapeSim(prb, chloro)
esp_sim = GetEspSim(prb, chloro)
print(f'{prb_name}\t{shape_sim:.2f}\t\t{esp_sim:.2f}')
得られた結果は以下のようになりました.
予想通りShapeSimはいずれも高い類似性を示していますが,置換基の性質によってEspSimは0.52から0.97まで差が出ています.電子吸引性置換基(Br, CN)の方が高い値が出ていることから,正しく評価できそうです.
Similarities with chlorobenzene prbMol Shape-Sim Esp-Sim methyl 0.93 0.52 bromo 0.94 0.97 cyano 0.91 0.79 phenol 0.91 0.68
ESP-Simの詳細
前項ではESP-Simの簡単な使い方を見ましたので,本項ではESP-Simの詳細についてもう少し掘り下げてみます.
ShapeTanimotoではVan der Waals表面の重なりに基づいて距離・類似度を算出しました.名前の通り距離関数はTanimotoです.
ESP-Simでは各原子上の電荷から形成されるクーロンポテンシャルの重なりを評価します.その際,
- 各原子の電荷計算方法
- クーロンポテンシャルの計算方法
- ポテンシャルの重なりの評価方法
などの点で設定が可能です.
以下の5つの二置換ベンゼンを用いて見ていきましょう.
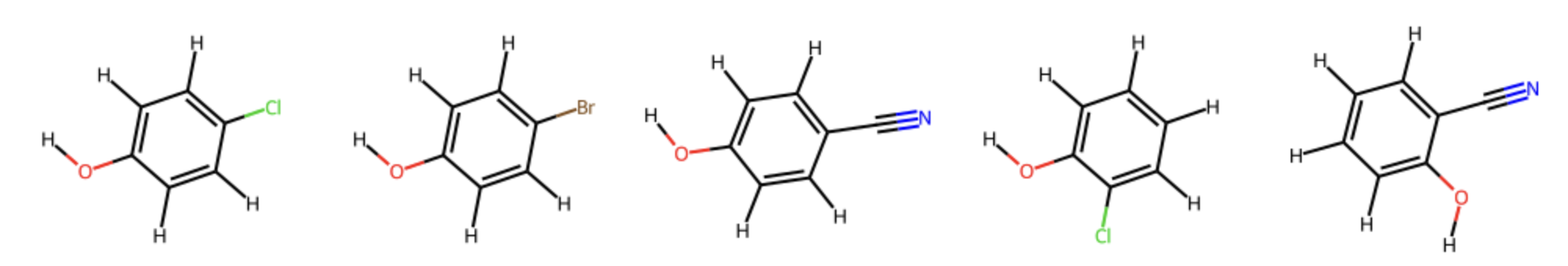
# 分子の準備
p_OH_Cl = Chem.AddHs(Chem.MolFromSmiles('Oc1ccc(Cl)cc1'))
p_OH_Br = Chem.AddHs(Chem.MolFromSmiles('Oc1ccc(Br)cc1'))
p_OH_CN = Chem.AddHs(Chem.MolFromSmiles('Oc1ccc(C#N)cc1'))
o_OH_Cl = Chem.AddHs(Chem.MolFromSmiles('Oc1ccccc1Cl'))
o_OH_CN = Chem.AddHs(Chem.MolFromSmiles('Oc1ccccc1(C#N)'))
# 立体構造を生成
for mol in [p_OH_Cl, p_OH_Br, p_OH_CN, o_OH_Cl, o_OH_CN]:
AllChem.EmbedMolecule(mol, AllChem.ETKDGv3())
# フェノール部位を鋳型とし,クロロフェノール類を基に重ね合わせ
core2 = Chem.MolFromSmiles('Oc1ccccc1')
AllChem.AlignMol(prbMol=p_OH_Br, refMol=p_OH_Cl,
atomMap=list(zip(p_OH_Br.GetSubstructMatch(core2),
p_OH_Cl.GetSubstructMatch(core2))))
AllChem.AlignMol(prbMol=p_OH_CN, refMol=p_OH_Cl,
atomMap=list(zip(p_OH_CN.GetSubstructMatch(core2),
p_OH_Cl.GetSubstructMatch(core2))))
AllChem.AlignMol(prbMol=o_OH_CN, refMol=o_OH_Cl,
atomMap=list(zip(o_OH_CN.GetSubstructMatch(core2),
o_OH_Cl.GetSubstructMatch(core2))))
原子の電荷計算法
Esp-Simを求める際にpartialChargesオプションを指定することで電荷の計算方法を変更できます.
実装としては
- gasteiger
- mmff
- ml
- resp
の4種類が選択可能で,デフォルトはRDKitで計算可能なGasteiger電荷です.mlとresp電荷のオプションではespsim以外のライブラリーのインストールが必要です.
RESP電荷は量子化学計算による静電ポテンシャルマップを再現するように決められた電荷ですので,最も精度が高いと考えられます.論文中でも高精度のMP2計算で得た値をベンチマークに設定しています.定性的にはどの手法でも同じ傾向を示しますが,論文中ではB3LYP/6-31G(d,p)で計算したRESP電荷の利用が推奨されています.
Gasteiger・MMFFの使い方
GasteigerやMMFFではpartialChargesオプションを変えるだけです.
print(f'gasteiger charge: {GetEspSim(p_OH_Cl, p_OH_Br, partialCharges="gasteiger"):.2f}')
# gasteiger charge: 0.52
print(f'MMFF charge: {GetEspSim(p_OH_Cl, p_OH_Br, partialCharges="mmff"):.2f}')
# MMFF charge: 0.49
RESPの使い方
一方でRESP電荷を用いる場合にはまずrespパッケージのインストールが必要です.
!conda install resp -c psi4 import resp
さらに私の環境ではpartialCharges=’resp’とするだけではエラーが出てしまったため,別途計算したChargeを与えています.
prbChargeやrefChargeを与えることで,GetEspSim内での電荷計算は省略され,与えられた電荷が計算に使われます.今回はこの方法でpartialCharges=’resp’と同等の結果を得ています.
具体的にはEspSim内で使われているpsi4Chargesという関数を利用してRESP電荷を計算します.今回は臭素原子を含む分子があるため,臭素の原子半径を論文推奨の1.8として設定しています.
# EspSimのhelpers関数から取得,一部改変
def psi4Charges(xyz,
basisPsi4 = '3-21G',
methodPsi4 = 'scf',
gridPsi4 = 1,):
"""
Calculates RESP charges via Psi4.
:param xyz: String of xyz file of an embedded molecule.
:param basisPsi4: (optional) Basis set.
:param methodPsi4: (optional) Method.
:param gridPsi4: (optional) Integer grid point density for ESP evaluation.
:return: Array of RESP partial charges.
"""
mol = psi4.core.Molecule.from_string(xyz, dtype='xyz')
mol.update_geometry()
options = {'VDW_SCALE_FACTORS' : [1.4, 1.6, 1.8, 2.0],
'VDW_POINT_DENSITY' : int(gridPsi4),
'RESP_A' : 0.0005,
'RESP_B' : 0.1,
'BASIS_ESP' : basisPsi4,
'METHOD_ESP' : methodPsi4,
'VDW_RADII': {'BR': 1.8} # 臭素原子の半径を与える
}
charge = resp.resp([mol], options)[1]
return charge
# RDKitのMOLオブジェクトからxyzブロックを作成して,psi4に渡す
def getPsi4Charge(mol, basisPsi4='3-21g', methodPsi4='scf') -> np.array:
xyz: string = Chem.MolToXYZBlock(mol)
return psi4Charges(xyz, basisPsi4, methodPsi4)
getPsi4Chargeを使って事前に電荷を計算しておくことで,GetEspSimメソッドに反映させます.今回は2つの計算レベルで実行しました.
# HF/3-21Gで計算
charges = GetEspSim(p_OH_Cl,
p_OH_Br,
prbCharge=getPsi4Charge(p_OH_Cl),
refCharge=getPsi4Charge(p_OH_Br))
print(f'resp charge at HF/3-21G: {charges:.2f}')
# B3LYP/6-31G(d,p)で計算
charges2 = GetEspSim(p_OH_Cl,
p_OH_Br,
prbCharge=getPsi4Charge(p_OH_Cl, basisPsi4='6-31g(d,p)', methodPsi4='b3lyp'),
refCharge=getPsi4Charge(p_OH_Br, basisPsi4='6-31g(d,p)', methodPsi4='b3lyp'))
print(f'resp charge at b3lyp/6-31g(d,p): {charges2:.2f}')
デフォルトのHF/3-21Gと推奨のB3LYP/6-31G(d,p)で計算してみましたが,先に得たGasteigerやMMFFも含めて,電荷計算方法の違いによる差はそこまでなさそうでした.
resp charge at HF/3-21G: 0.52 resp charge at b3lyp/6-31g(d,p): 0.50
クーロンポテンシャルの計算方法
クーロンポテンシャルの積分方法として
- gauss
- mc
の2つのオプションが選択可能です.デフォルトはgaussです.
以下の式であらわされるクーロンポテンシャルの積分を簡単に実行するため,複数のガウス関数でポテンシャルを近似する方法がgaussオプションです.
$$ V(r) = \frac{1}{4 \pi \varepsilon _0} \sum _i \frac{q_i}{| r – r_i |} $$
この場合0に近い部分では近似がうまくいきませんが,積分範囲はVan der Waals半径の外の部分で実行するため問題となりません.解析的に積分が実行可能なため高速に計算可能です.
一方でモンテカルロ法による数値積分を実行するのがmcオプションになります.
計算時間がかなり異なりますので,特にこだわりがない場合はgaussオプションを用いるのがいいでしょう.
下記のコードでは%timeitコマンドを用いて実行時間を測定しています.予想通りgaussオプションの方が圧倒的に速いことがわかります.
%timeit GetEspSim(p_OH_Cl, p_OH_Br, integrate='gauss') # 921 µs ± 40.7 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each) %timeit GetEspSim(p_OH_Cl, p_OH_Br, integrate='mc') # 641 ms ± 14.4 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
クーロンポテンシャルの重なりの評価方法
積分を実行したクーロンポテンシャルの重なりを評価する方法をmetricオプションで指定できます.
- carbo
- tanimoto
の2つが設定可能です.Carbo類似度は-1から1,Tanimoto類似度は-1/3から1の範囲を取ります.そのため異なる指標で計算した類似度の比較を容易とするためrenormalizeオプションが用意されています.
Carbo類似度は
- 電荷の位置には感度が高いが,その絶対値には受容性が高い
という特徴を有しています.そのため,例えば分子内の正電荷と負電荷の分布を大局的に捉えたい場合に適しています.
一方でTanimoto類似度では,
- 電荷の絶対値の差にも敏感
という特徴があるため,例えば双極子モーメントの向きだけでなく,その絶対値も気にかけたい場合に適していると言えます.
これら特徴を踏まえて,自身の目的に合わせた指標を適切に選択することが大切となります.
以下の例では
- p-クロロフェノールとp-シアノフェノール
- o-クロロフェノールとo-シアノフェノール
というペアについて,CarboとTanimoto類似度を求めています.両方を比較しやすいようにrenormalizeオプションを指定しています.
print('### Carbo ###')
print(f'ESP-Sim of para-substituted compounds using Carbo metric: {GetEspSim(p_OH_Cl, p_OH_CN, metric="carbo", renormalize=True):.2f}')
print(f'ESP-Sim of ortho-substituted compounds using Carbo metric: {GetEspSim(o_OH_Cl, o_OH_CN, metric="carbo", renormalize=True):.2f}')
print('### Tanimoto ###')
print(f'ESP-Sim of para-substituted compounds using Tanimoto metric: {GetEspSim(p_OH_Cl, p_OH_CN, metric="tanimoto", renormalize=True):.2f}')
print(f'ESP-Sim of ortho-substituted compounds using Tanimoto metric: {GetEspSim(o_OH_Cl, o_OH_CN, metric="tanimoto", renormalize=True):.2f}')
塩素原子とシアノ基では電子吸引能が相当異なりますが,置換基の位置は同じためCarbo類似度ではパラ,オルト置換体ともに高い類似度が得られていることが確認できます.一方でTanimoto類似度では電子吸引力の差が類似度に反映され,低い数値が得られていると考えられます.
### Carbo ### ESP-Sim of para-substituted compounds using Carbo metric: 0.64 ESP-Sim of ortho-substituted compounds using Carbo metric: 0.81 ### Tanimoto ### ESP-Sim of para-substituted compounds using Tanimoto metric: 0.37 ESP-Sim of ortho-substituted compounds using Tanimoto metric: 0.58
具体例:ペプチド結合等価体の評価
最後にペプチド結合等価体として用いられるフルオロアルケンを題材に,ESP-Simを用いて類似性を評価してみます.具体的には次の化合物を扱います.
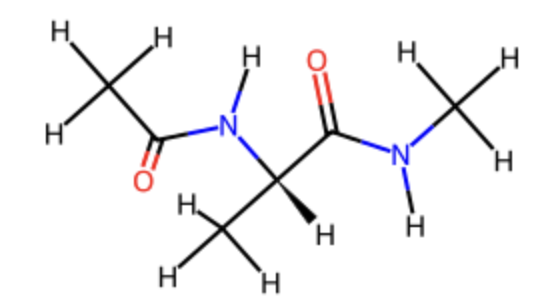
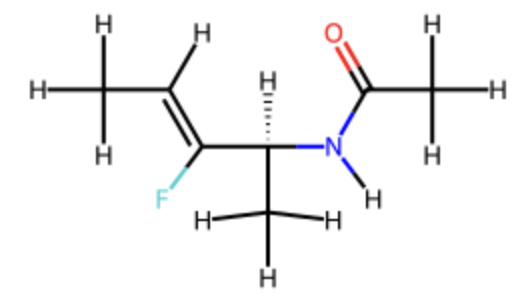
これら化合物に対して,
- psi4を用いてB3LYP/6-31G(d,p)レベルで構造最適化と振動数計算を行う
- RDKitのxyz2molを用いて最適化構造をMOLオブジェクトに変換
- RDKitのO3Aを用いて二つの構造を重ね合わせ
- ESP-Simを用いて類似度の評価
という順番で処理をしていきます.
まずはアミドとオレフィンの初期構造を定義します.また与えられた初期構造を基にpsi4で構造最適化と振動数計算を行い,最適化構造をXYZ形式で返す関数を作成します.
# アミドの初期構造
amide_xyz = '''
0 1
C -0.85007050 0.59890023 -0.11291408
O -1.23585306 -0.50245815 -0.58381969
N 0.40298549 0.67267127 0.65214050
H 0.81204273 1.57767795 0.53537116
C 1.33593477 -0.35336229 0.16453331
H 1.54062317 -0.18554318 -0.87221131
H 2.24802314 -0.29966500 0.72140977
H 0.89824352 -1.32171944 0.28947650
C -1.69068431 1.86943248 -0.33811674
H -1.37665406 2.34940815 -1.24139320
C -1.49559544 2.83109420 0.84878977
H -0.46195359 3.09646503 0.92665409
H -2.07965827 3.71386661 0.69231779
H -1.80962569 2.35111853 1.75206622
N -3.11073432 1.50485760 -0.44508923
C -3.91313841 2.71763839 -0.66005541
H -3.40422054 1.05628221 0.39909438
O -3.41603555 3.70162001 -1.26688279
C -5.36209487 2.78399732 -0.14263714
H -5.93887066 3.42296210 -0.77821067
H -5.78671712 1.80186014 -0.14421982
H -5.36743933 3.17327626 0.85402395
'''
# オレフィンの初期構造
olefin_xyz = '''
0 1
C -1.10461862 0.27078483 0.38719779
H 0.65650931 1.33953336 0.82048296
C 0.83504163 -0.80037242 1.56427693
H 1.38135686 -1.35562125 0.83069776
H 1.50706389 -0.44939333 2.31932644
H 0.09494166 -1.43110271 2.01074231
C -1.79292148 1.47255941 -0.28628348
H -1.56543094 1.47397396 -1.33181973
C -1.28458018 2.77889198 0.35143460
H -0.22565507 2.85295396 0.21692647
H -1.76281658 3.61389121 -0.11650369
H -1.51207072 2.77747743 1.39697085
N -3.24770644 1.37081072 -0.10149194
C -3.90472280 2.51795919 -0.74436043
H -3.46031442 1.36948871 0.87564475
O -3.38359616 3.05604525 -1.75552855
C -5.23076804 3.06123685 -0.18039818
H -5.81074785 3.48170685 -0.97521162
H -5.77762727 2.26310136 0.27657525
H -5.02527210 3.81637448 0.54928571
F -1.74779050 -0.91283617 0.47591800
C 0.14673716 0.40140332 0.89079937
'''
# 初期構造からpsi4のMoleculeを作成し,構造最適化と振動数計算を行いXYZ形式を返す
def opt_freq(xyz: str) -> str:
psi4_mol: psi4.Molecule = psi4.geometry(xyz)
print('start optimization')
e, wfn = psi4.optimize('b3lyp/6-31g(d,p)',
molecule=psi4_mol,
return_wfn=True)
print(f'optimized structure:\n{psi4_mol.save_string_xyz()}')
print('\nstart freq computation')
e, wfn = psi4.frequency('b3lyp/6-31g(d,p)',
molecule=psi4_mol,
ref_gradient=wfn.gradient(),
return_wfn=True)
return psi4_mol.save_string_xyz_file()
続いてXYZ形式の最適化構造からxyz2molを用いてRDKitのMOLオブジェクトを作成します.今回は共通部分構造が少ないため,O3Aを用いて重ね合わせを試みています.最後にESP-Simを求めます.
# xyz2molを用いてMOLオブジェクトの作成
amide = Chem.MolFromXYZBlock(opt_freq(amide_xyz))
rdDetermineBonds.DetermineBonds(amide)
olefin = Chem.MolFromXYZBlock(opt_freq(olefin_xyz))
rdDetermineBonds.DetermineBonds(olefin)
# O3Aを用いて構造の重ね合わせ
from rdkit.Chem import rdMolDescriptors
crippen_contribs_amide = rdMolDescriptors._CalcCrippenContribs(amide)
crippen_contribs_olefin = rdMolDescriptors._CalcCrippenContribs(olefin)
o3a = AllChem.GetCrippenO3A(prbMol=olefin,
refMol=amide,
prbCrippenContribs=crippen_contribs_olefin,
refCrippenContribs=crippen_contribs_amide)
o3a.Align()
# ESP-Simの計算
score = GetEspSim(prbMol=olefin, refMol=amide)
print(f'ESP-Sim between amide and fluoroalkene: {score:.2f}')
得られたESP-Simは0.48でした.数字だけ見ますとあまり似ていないように思われます.
ESP-Sim between amide and fluoroalkene: 0.48
終わりに
今回は「ESP-Simで分子の電子的性質に着目した類似性を評価する」という話題について,
- 分子の形の類似度
- ESP-Simとは何か
- ESP-Simの使い方
- ESP-Simを用いたペプチド結合等価体の評価
などについて触れてきました.
なお今回は分子の立体構造はRDKitのETKDGメソッドやpsi4を用いて量子化学計算により最適化した構造を用意した上でESP-Simの計算を行いました.espsimにはRDKitのMOLオブジェクトと鋳型の構造を用意するだけで立体構造の発生から重ね合わせ,Esp-Simの計算までを自動で行ってくれるEmbedAlignConstrainedScoreメソッドも用意されています.興味のある方はgithubのnotebookなどを見てみてください.
コメント