これまで「RDKitとk平均法による化合物の非階層型クラスタリング」と「RDKitとウォード法による化合物の階層型クラスタリング」という2つの記事で化合物のクラスタリングについて解説してきました.
これは多様性の高いライブラリーを構築するための4つの方法,
- クラスタリング法
- 距離ベースの方法
- 区分けをベースとした方法
- 最適化手法を用いる方法
のうちの1つめにあたります.
今回は「2. 距離ベースの方法」としてRDKitに実装されているMaxMin法を使って多様なライブラリーを構築していきたいと思います.
– RDKitブログ:Revisting the MaxMinPicker
– Jupyter Notebook:MaxMinPicker

分子の準備
今回もナミキ商事の運営するChemCupidからダウンロードした,カタログの新規追加分の分子を収めたSDFを使い,フリー体の化合物66005個を扱います.
最後にクラスタリングの際と同じ分子を選ぶようにランダムシードを合わせた上で,分子の順番をシャッフルし,Morganフィンガープリントを2048ビット長で作成します.このうち最初の5000分子を使っていきます.
### ライブラリのインポート
import bumpy as np
from rdkit import Chem, DataStructs
from rdkit.Chem import AllChem
from rdkit.SimDivFilters.rdSimDivPickers import MaxMinPicker
### 分子の読み込み
suppl = Chem.SDMolSupplier('./Namiki_NEW_201807_0001.sdf')
mols = [x for x in suppl if x is not None if x.GetProp('NScode').endswith('0000')]
len(mols) ### 66005
### シャッフル
np.random.seed(1234)
np.random.shuffle(mols)
### フィンガープリントの作成
morgan_fp = [AllChem.GetMorganFingerprintAsBitVect(x, 2, 2048) for x in mols]
距離ベースの方法の概略
クラスタリング法では
- データセットを指定したクラスター数に分割
- 各クラスターから分子を選択
という2段階で分子を選んでいきました.一方で今回扱う距離ベースのMaxMin法では1段階で分子が選択されます.
RDKitにおけるMaxMin法の実装
RDKitでは多様な化合物を選択するためのクラスタリング法やMaxMin法などはSimDivFilters.rdSimDivPickersに実装されています.今回はMaxMinPickerを使っていきます.
MaxMinPickerはいくつかの使い方があります.いずれもMaxMinPickerインスタンスを作成してから選択します.
LazyPick:距離も計算させる方法
LazyPickは
- 距離を返す関数
- もとの集団の大きさ
- 選ぶ化合物の数
の3つを引数としてとり,選んだ化合物IDのリストを返します.
下のコードでは「1 – タニモト係数」を返す関数を自分で定義しています.また後の結果と比べるためにランダムシードを設定しています.
### 距離を返す関数を定義
def dis_fps(i,j):
return 1 - DataStructs.TanimotoSimilarity(morgan_fp[i],morgan_fp[j])
### LazyPickの利用
pick = MaxMinPicker()
lazy = pick.LazyPick(dis_fps, 5000, 6, seed=1234)
list(lazy)
6個の分子が選ばれました.
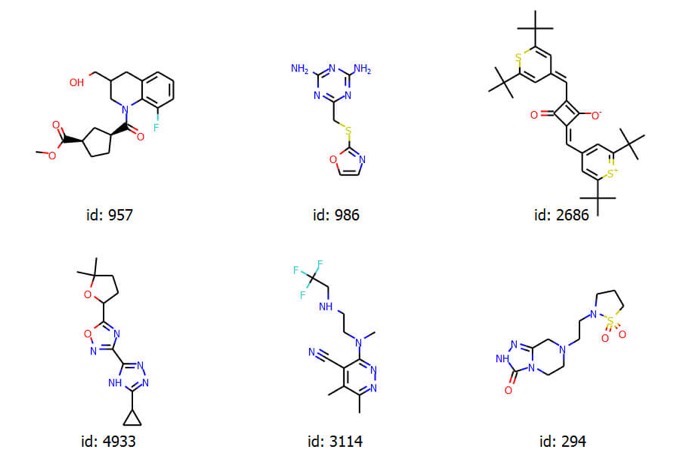
[957, 986, 2686, 4933, 3114, 294]
Pick:距離行列を渡して選択だけさせる方法
Pickは
- numpy配列型の距離行列
- もとの集団の大きさ
- 選ぶ化合物の数
の3つを引数としてり,選んだ化合物IDのリストを返します.
LazyPickは距離の計算方法を与えて,メソッド側で距離を計算させた上で化合物を選択させる方法でした.Pickは距離行列を引数にとり,それをもとに化合物の選択を行うメソッドです.距離の計算をメソッドにさせるか,事前にこちらで計算しておくかが違いになります.
距離行列としては,全行列のうち下半分を1つのリストにしたものを与えます.つまり,下の図の灰色で囲んである部分を赤い数字の順番に1つのリストにします.ここを間違えると結果が変わってきてしまいますので注意してください.
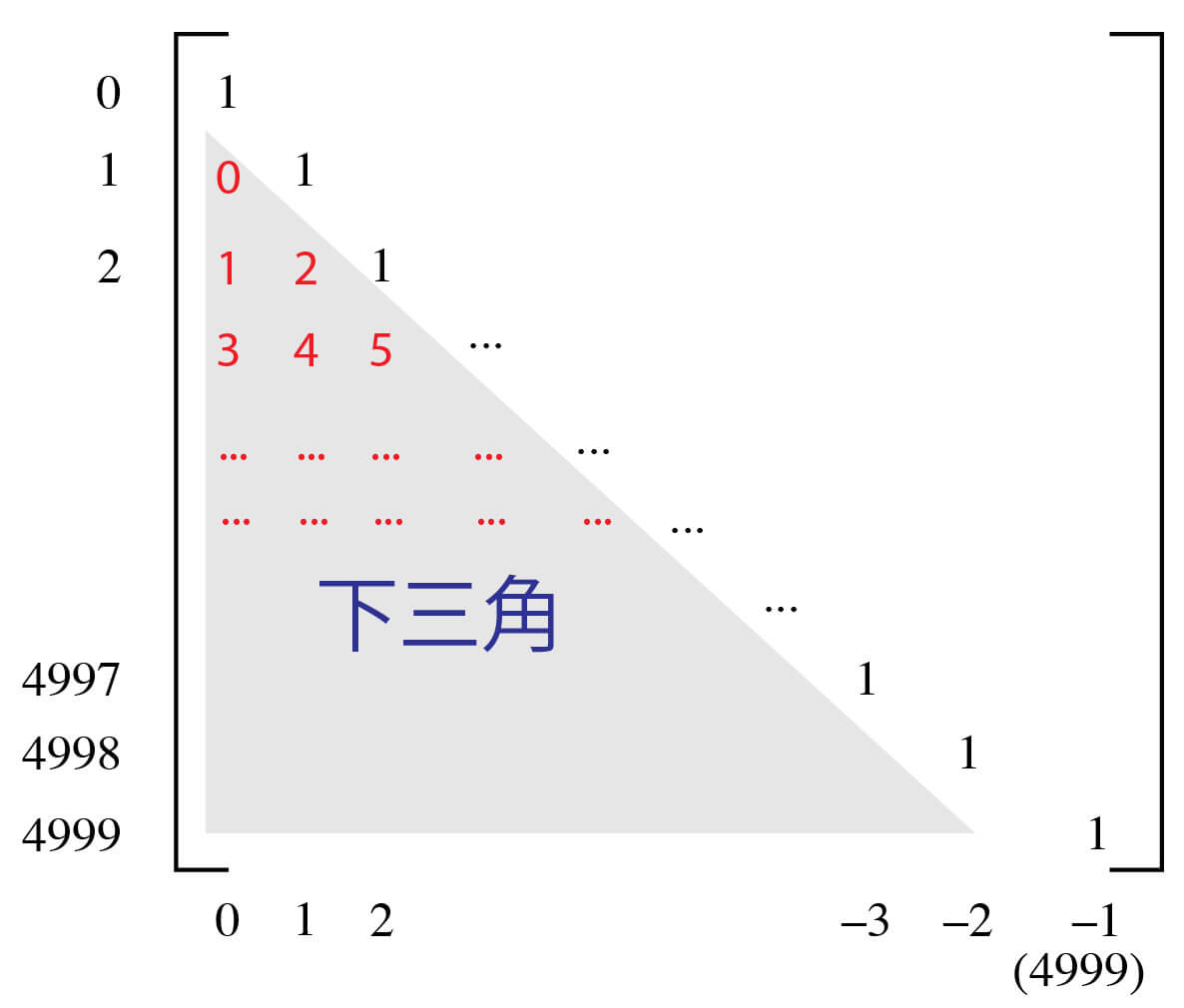
BulkTanimotoSimilarityはreturnDistance=Trueオプションを設定することでの出力が「1 – タニモト係数」によって計算した距離に変わります.下のコードではこれを利用して距離行列を作成しています.今回もランダムシードを設定しています.
### 距離行列の作成
dis_mat = []
for i in range(1,5000):
dis_mat.extend(DataStructs.BulkTanimotoSimilarity(morgan_fp[i], morgan_fp[:i], returnDistance=True))
### Pickの利用
ids = pick.Pick(np.array(dis_mat), 5000, 6, seed=1234)
list(ids)
6個の分子が選ばれました.
[957, 986, 2686, 4933, 3114, 294]
LazyPickの場合と同じランダムシードを設定したため,同じ分子が選ばれています.すなわちこれらのメソッドは引数が異なるだけで,化合物選択に用いるアルゴリズムは同じです.
LazyBitVectorPick:フィンガープリントを与えて距離を計算させる
LazyBitVectorPickはLazyPickと同様にメソッドに距離を計算させて,化合物を選択します.2つのメソッドの違いはフィンガープリントそのものを与えるか,距離を計算する関数を与えるかになります.
lazy_bit = pick.LazyBitVectorPick(morgan_fp[:5000], 5000, 6, seed=1234) list(lazy) == list(lazy_bit) ### True
いずれの場合も同じ分子が選択されます.なお選ばれた分子の構造は以下のようなものでした.
終わりに
今回は「RDKitのMaxMin法を用いた多様性の高いライブラリの選定」という話題について,RDKitに実装されているMaxMin法を用いて多様な化合物を選択してみました.
特にMaxMinPickerはいくつかの使い方がありますが,そのうち
- LazyPick:距離も計算させる方法
- Pick:距離行列を渡して選択だけさせる方法
- LazyBitVectorPick:フィンガープリントを与えて距離を計算させる
という3つの方法を説明しました.
次回は,多様性の高いライブラリーを構築するための4つの方法,
- クラスタリング法
- 距離ベースの方法
- 区分けをベースとした方法
- 最適化手法を用いる方法
のうち,まだ説明してこなかった「区分けをベースとした方法」「最適化手法を用いる方法」について説明しながら,このトピックの復習と総括をしていきたいと思います.
>>次の記事:「ケモインフォマティクスで多様なライブラリーを構築する」
コメント