本ブログでは「有機合成化学者のためのケモインフォマティクス入門」を掲げて,特にpythonを用いてケモインフォマティクスを行う際に必要となる環境構築方法から解説してきました.例えば「RDKitでケモインフォマティクスに入門」という記事では,pythonのケモインフォマティクス用ライブラリであるRDKitのインストール方法から簡単な使い方まで解説しています.
一方で化学系の多くの人にとってはメール,ウェブブラウジング,オフィス・ChemDrawを用いた文書作成,NMR等の実験データの解析以外にコンピュータを使うことはほとんどなく,pythonの環境構築が大きな課題となることも事実だと思います.
今回はGoogleが提供するブラウザ上でpythonの実行環境が無料で利用可能な,Google Colabの紹介と,Colab上へのRDKitのインストール方法を解説していきます.
Google Colabとは
Google ColaboratoryとはGoogleが提供しているクラウド上のpython実行環境です.ChromeやSafariなどのブラウザ上で,Jupyter Notebookを基盤とした実行環境でコードが実行できるようになっています.
Googleアカウントさえ持っていれば「無料」でGPUを利用可能ということで,注目を集めているサービスです.このように無料で使えるGoogle Colabですが,実行時間に制限があります.覚えておくべき制限は以下の通りです.
- 新しいインスタンス(実行環境)を作成して最大12時間経過するとリセットされる(12時間ルール)
- セッションが切れて90分経過するとリセットされる(90分ルール)
その他メモリやディスク容量にも制限がありますので,あまりに大きなデータセットを扱って分析を行うのは現実的でありません.インスタンスが消えると初期状態にリセットされてしまうので,ライブラリのインストールなどを始めから行う必要がある点に注意が必要です.
このような制限を緩和し,より高機能なGPUが優先的に割り当てられる有料サブスクリプションプランColab ProやColab Pro+といったプランも登場しています.
また2022年10月からはコンピューティングユニットという,90日間有効な高性能マシンの利用権にあたるものを都度買い(従量課金)することも可能となりました.
Google Colabの使い方
Google Colabにアクセスすると以下のようなウィンドウが登場します.
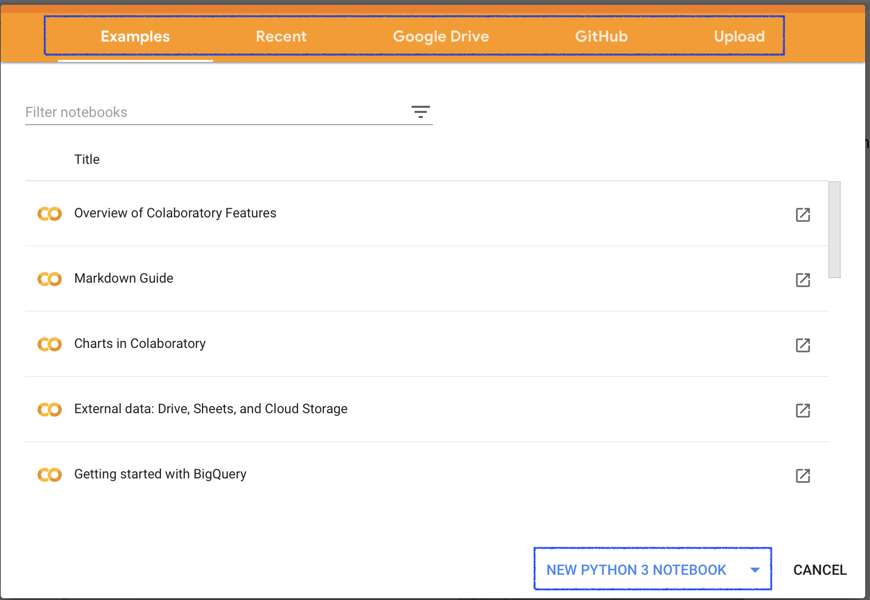
上のタブには
- Googleが最初から用意しているデモ用ノートブック
- 最近Colab上で使ったノートブック
- Googleドライブ上のノートブック
- GitHub上のノートブック
- ローカルからアップロード
の選択肢があります.また右下から新規ノートブックの作成が可能です.作成したノートブックはGoogleドライブ上の「Colab Notebooks」というフォルダに保存されます.
技術書ではコードの例がgithubにアップロードされていて,コードを実行しながら読み進めたい場合も多いと思います.そういった場合には「github」タブから,ノートブックにアクセス可能です.
例として,Aurélien Geron著の「Hands-on Machine Learning with Scikit-Learn, Keras and TensorFlow」を見てみます.URLの部分にgithubのページを打ち込むとrepositoryのデータが取得され,ノートブックの一覧が表示されます.あとは希望のファイル名を選択するだけで,Colab上でノートブックが開かれます.
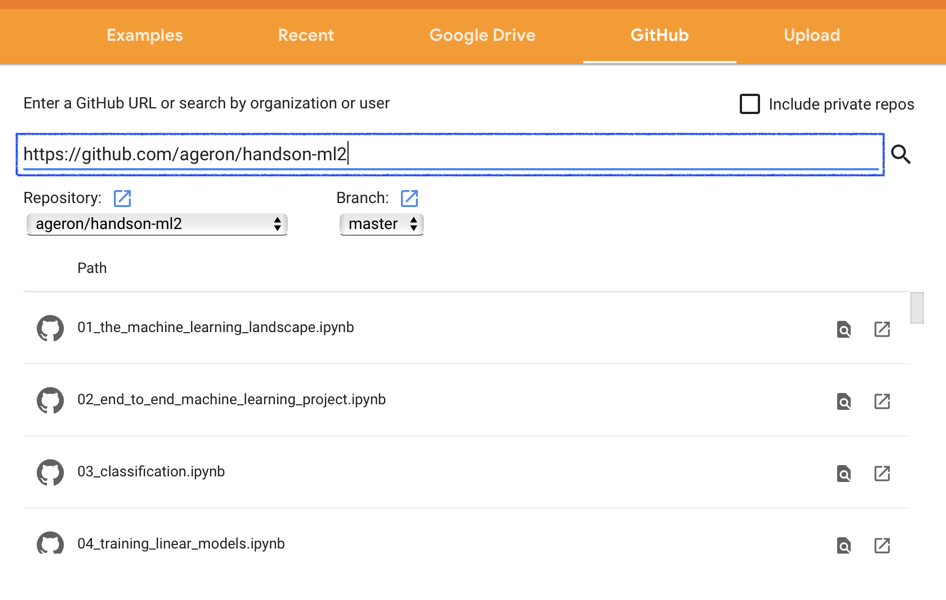
ノートブックの設定方法
GPUの利用設定
Google Colabは「無料でGPUが利用可能」ということで話題を集めているサービスですが,新しくノートブックを作成した段階ではGPUを利用する設定になっていません.
まず「Edit-> Notebook settings」に進みます.
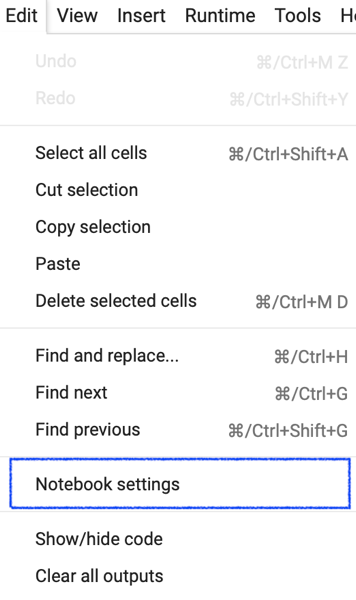
表示されたウィンドウの「Hardware accelerator」からGPUを選択することでGPUの利用が有効になります.ここではGoogleの開発しているTPU(Tensor Processing Unit)も選択可能ですが,TPU利用に最適化したコードを書く必要があるようです.
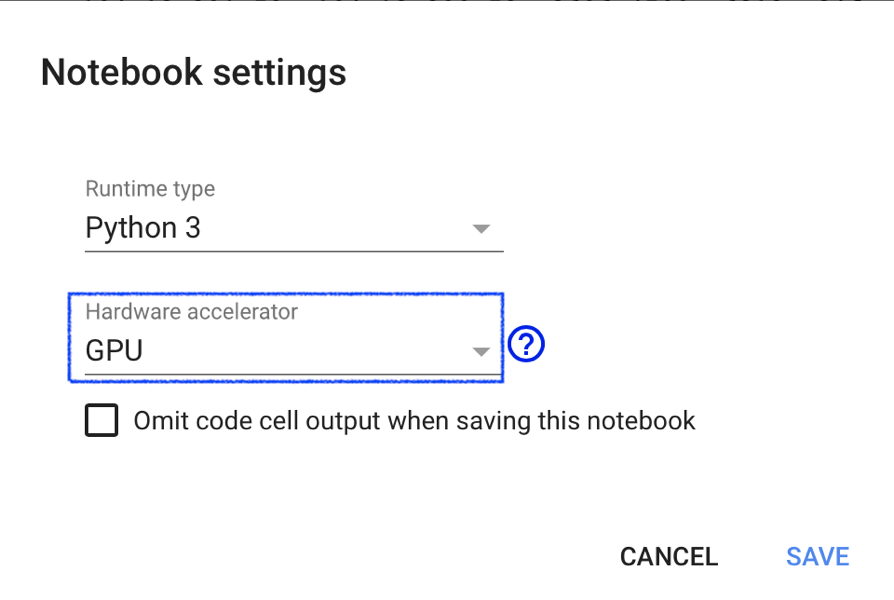
なお有料プランを選択している方は,こちらの設定画面から
- 高性能GPU
- 高メモリマシン
などが選択可能です.
インストール済みライブラリ
Google Colabでは,ノートブック作成後にすぐに作業が開始できるように主要なライブラリーが既にインストールされています.
2023年5月時点でpythonのバージョンは3.10,pipで確認可能なものは405個でした.
!python -V # Python 3.10.11 !pip freeze | wc -l # 405
いくつかデータ分析・可視化・機械学習で使いそうなライブラリーを挙げておきます.
| ライブラリ名 | バージョン |
|---|---|
| altair | 4.2.2 |
| arviz | 0.15.1 |
| beautifulsoup4 | 4.11.2 |
| bokeh | 2.4.3 |
| graphviz | 0.20.1 |
| jax | 0.4.8 |
| Keras | 2.12.0 |
| lightgbm | 3.3.5 |
| matplotlib | 3.7.1 |
| numba | 0.56.4 |
| numpy | 1.22.4 |
| pandas | 1.5.3 |
| plotly | 5.13.1 |
| pymc | 5.1.2 |
| scikit-learn | 1.2.2 |
| scipy | 1.10.1 |
| seaborn | 0.12.2 |
| statsmodels | 0.13.5 |
| sympy | 1.11.1 |
| tensorflow | 2.12.0 |
| torch | 2.0 |
| xgboost | 1.7.5 |
| yellowbrick | 1.5 |
このようにColabでは標準で非常に多くのライブラリが用意されており,すぐに作業に取り掛かることが可能となっています.しかし,ケモインフォマティクス用のツールなど専門性の高いものは準備されていませんので各自でインストールする必要があります.
例えば「化合物データベースPubChemをpythonで使いこなす」という記事で解説しているpubchempyをpipを用いてインストールしてみましょう.
!pip install pubchempy
RDKitのインストールとimport
「RDKitでケモインフォマティクスに入門」という記事でも説明したように,2022年時点ではpip,condaのいずれを用いてもrdkitをインストールすることが可能です.
Colab上にconda環境を準備するのはやや面倒ですので,「計算化学にpythonとpsi4で入門」という記事で説明している量子化学計算用ライブラリpsi4を同時に使いたいなどの理由がない限りpipを用いるのがオススメです.
pipを用いたインストール
pipを用いる場合,通常のinstallコマンドで問題なくインストール可能です.
!pip install rdkit
きちんとインストールできていることを確認してみます.無事にimportできれば問題ありません.
from rdkit import rdBase
print(f'rdkit version from pip: {rdBase.rdkitVersion}')
# rdkit version from pip: 2022.09.3
condaを用いたインストール
初期状態ではcondaコマンドが使えませんので,まずcondaインストールのためのスクリプトファイルをダウンロードして環境構築を行う必要があります.
以下に,長々とColab上へのRDKitのインストール方法を書きましたが,時間がない人やとりあえず動けばいいという人は,以下のコードを貼り付ければColab上にconda環境の構築からrdkitのimportまでができるようになるはずです.
!wget -c https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh
!chmod +x Miniconda3-latest-Linux-x86_64.sh
!bash ./Miniconda3-latest-Linux-x86_64.sh -b -f -p /usr/local
!conda install -q -y -c conda-forge rdkit python=3.10
import sys
sys.path.append('/usr/local/lib/python3.10/site-packages')
コード各部の説明については以下に説明をつけていますので参照してください.
conda環境の構築とRDKitのインストール
Colab上にconda環境を構築するために,
- minicondaのインストールスクリプトのダウンロード
- スクリプトの実行権限の付与
- インストールスクリプトを実行して/use/local下に環境構築
- condaコマンドでRDKitのインストール
という手順でconda環境構築を行い,RDKitをインストールしていきます.pythonのバージョンはシステムと同じ3.10を指定しています.
!wget -c https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh !chmod +x Miniconda3-latest-Linux-x86_64.sh !bash ./Miniconda3-latest-Linux-x86_64.sh -b -f -p /usr/local !conda install -q -y -c conda-forge rdkit python=3.10
RDKitのimport
ここまででRDKitはインストールできていますが,このままではimportできません.pythonがimportするライブラリ・モジュールを探す場所を「モジュール検索パス」といいますが,condaでインストールしたrdkitの入っているディレクトリはこの中に入っていません.
モジュール検索パスの確認はsys.pathで確認できます.
import sys import pprint pprint.pprint(sys.path)
['/content', '/env/python', '/usr/lib/python310.zip', '/usr/lib/python3.10', '/usr/lib/python3.10/lib-dynload', '', '/usr/local/lib/python3.10/dist-packages', '/usr/lib/python3/dist-packages', '/usr/local/lib/python3.10/dist-packages/IPython/extensions', '/root/.ipython']
インストールできてもimportできないということは,RDKitは上記パス中には存在していないということになります.それではRDKitはどこにインストールされているのでしょうか?
下のコードのようにsystemで使われているpythonは3.10.11です.
print(sys.version)
3.10.11 (main, Apr 5 2023, 14:15:10) [GCC 9.4.0]
一方でcondaで使われているpythonは3.10.10であり,同じ3.10でも実態は異なることがわかります.
# condaインストール後 !python -V # Python 3.10.10
以下のコマンドを用いることで,conda環境のpython3.10.10に付随してインストールしたパッケージの場所を確認可能です.
!python -c "import site; print (site.getsitepackages())
['/usr/local/lib/python3.10/site-packages']
後はこの場所をモジュール検索パスに追加してあげればimportできるようになります.
sys.path.append('/usr/local/lib/python3.10/site-packages')
from rdkit import rdBase
print(f'rdkit version from conda: {rdBase.rdkitVersion}')
rdkit version from conda: 2022.09.1
conda環境構築を自動化するライブラリの利用
上述の工程を自動化するスクリプトを自分で書いて毎回読み込んでもよいのですが,pipでインストール可能なライブラリとして用意されているものも存在します.
condacolab
condacolabは2020年12月に登場したライブラリで,colab上のpython3.10に合わせてpython3.10のconda環境を構築します.
!pip install -q condacolab import condacolab condacolab.install()
パスの通ったconda環境が構築されてしまえば,あとは通常通りにインストールすればRDKitが利用可能になります.
!conda install -q -y -c conda-forge rdkit
import rdkit
print(f'rdkit version from condacolab: {rdkit.__version__}')
# rdkit version from condacolab: 2023.03.1
Google Colabへのファイルのアップロード方法
作業するにあたってファイルからデータを読み込む作業は必須ですので,どのようにGoogle Colabにファイルをアップロードするかを学んでおきましょう.
大きくわけて
- ローカルから直接ファイルを選択してアップロード
- Googleドライブ上のファイルへアクセス
の2つの方法があります.
ローカルからファイルのアップロード
ローカルからファイルを選択して,Colab上にアップロードすることが可能です.
例えばコードブロックの左側に存在するファイルエクスプローラー部位に所望のファイルをドラッグ&ドロップすることでファイルのアップロードが可能になっています.

またコードブロックにて以下のコードを実行することでもファイルのアップロードが可能です.
from google.colab import files uploaded = files.upload()
コマンドを入力すると以下のようにファイル選択画面が表示されますので,アップロードしたいファイルを選択します.

無事にアップロードされれば,ファイルが確認できます.今回は「線形モデルを用いた化合物の溶解度予測:通常最小二乗法,Ridge回帰,Lasso回帰」などの記事で使っている溶解度のデータをアップロードしてみました.
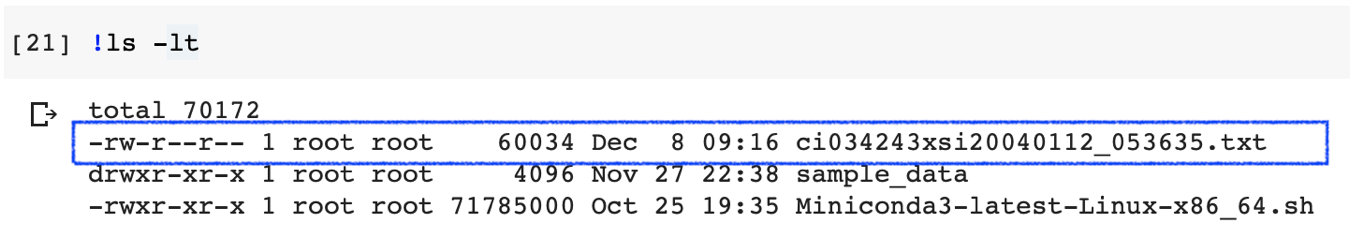
この方法は1つ1つのファイルを扱う必要があるため,大量のファイルを扱いたい場合などには不便です.また容量の大きいファイルをアップロードしようとすると失敗することが多い気がしています.こういった場合には次項のGoogleドライブ上のファイルにアクセスする方法を使いましょう.
Googleドライブのファイルにアクセス
colab.drive.mountを用いることで,Colab上にGoogleドライブをマウントすることができます.
以下のコードを実行するとまず
- Googleドライブへのアクセスを許可する認証
が行われ,その後
- drive以下にGoogleドライブをマウント
します.なお次のコードは直接コードブロックに書いてもいいですし,左側のファイルエクスプローラー部位の「Google Drive」のアイコンをクリックすることでも登場します.
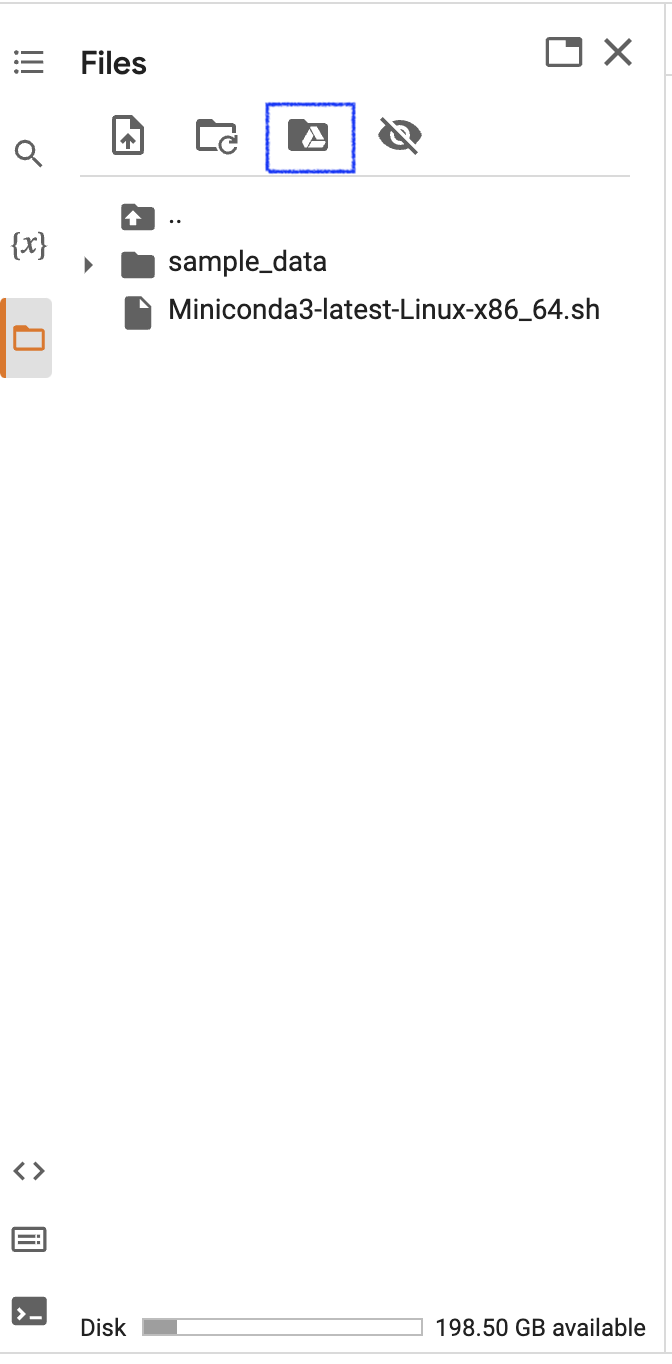
from google.colab import drive
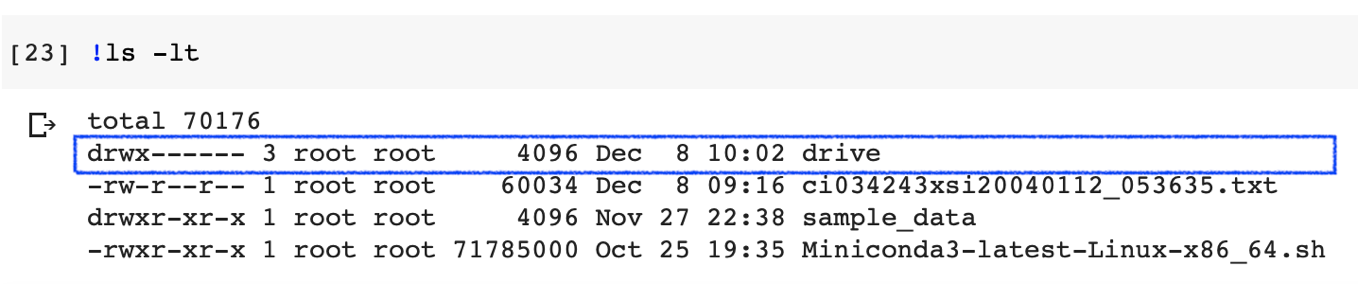
drive.mount('/content/drive')
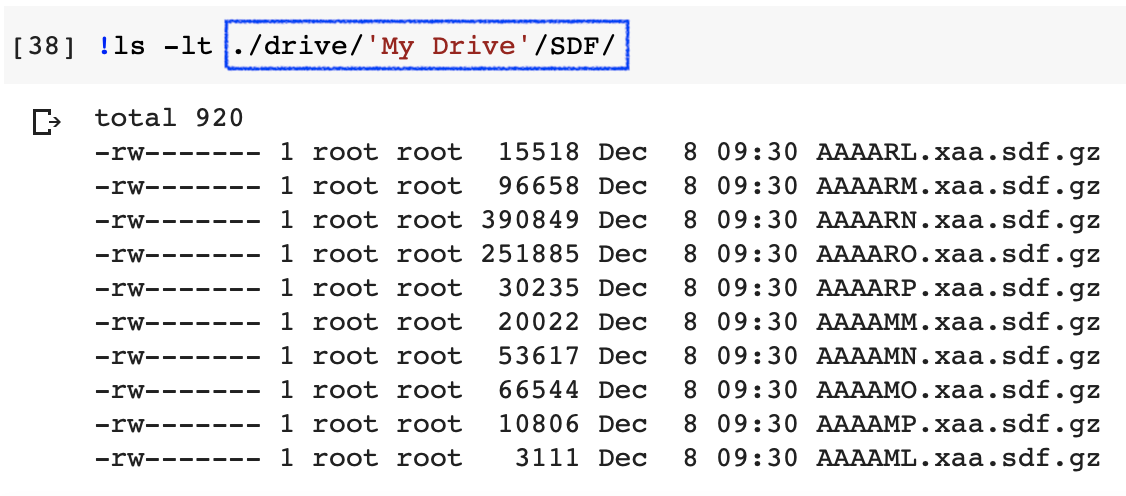
Googleドライブのトップディレクトリ(My Drive)にSDFというフォルダがありその中にいくつかZINC15からダウンロードしたSDFが入っているとします.
ここでは
- gzファイルで圧縮されたファイルのリストを取得
- RDKitのChem.ForwardSDMolSupplierを用いて読み込み
- 分子の読み込み
という作業でGoogleドライブ上のファイルからRDKitのMOLオブジェクトを作成します.
import glob
files = glob.glob('./drive/My Drive/SDF/*.sdf.gz')
import gzip
from rdkit import Chem
from rdkit.Chem import AllChem
mols = []
for file in files:
with gzip.open(file, 'rb') as f:
suppl = Chem.ForwardSDMolSupplier(f)
for mol in suppl:
if mol is not None:
mols.append(mol)
print(len(mols)) ## 4060
ファイルのエクスポート
pickle.dump
Google Colabでは利用時間に制限がありますので,作業結果を次回利用可能な形できちんと保存することが大切になります.
pythonオブジェクトをpickleモジュールを利用することで保存したり,pandasのデータフレームをcsv形式として保存することが可能です.なお作業ログとしてのノートブックはGoogleドライブ上のColab Notebooksフォルダに保存されます.
RDKitのMOLオブジェクトはSDFやSMILESなどの形で保存することも可能です.以下のコードでは先ほど作成したMOLオブジェクトのリストを1つのSDFとして保存しています.
with open('./drive/My Drive/SDF/zinc_sample.sdf', 'w+') as f:
writer = Chem.SDWriter(f)
for mol in mols:
writer.write(mol)
終わりに
今回は「Google ColabでRDKit:ケモインフォマティクス用のpython環境を手軽に構築」という話題について,
- Google Colabとは何か
- Google Colabの使い方
- Google ColabへのRDKitのインストールの仕方
などについて説明してきました.初心者にとってpythonの環境構築は挫折ポイントの1つですので,Google Colabを使うことで気軽に分析が始められる点は大きな利点だと思います.
本ブログではpythonのケモインフォマティクス用ライブラリであるRDKitについて基本から解説しています.是非学習を継続していきましょう.
>>次の記事:「RDKitでケモインフォマティクスに入門」

コメント