「RDKitでRECAPを用いた分子のフラグメント化」,及び「RDKitでBRICSを用いた仮想ライブラリーの構築」という記事では化合物のde novoデザインの方法論の一つとして,ライブラリ中の化合物のフラグメント化を行うRECAPとBRICSを扱いました.
これらの方法は医薬品様の化合物を,逆合成の観点を取り入れながらフラグメント化を行いますので,得られたフラグメントを用いて仮想のバーチャルライブラリーを構築した場合にもある程度の合成容易性が担保されているとも言えます.
とはいえ,リード候補化合物の合成しやすさを評価することは,その化合物がどのような経緯で得られたかに関わらず重要です.特にin silicoでどれだけ有望そうに見える化合物でも,実際に合成・評価されないことには「絵に描いた餅」です.
今回は化合物の合成しやすさを評価する方法として,SAスコア(Synthetic Accessibility score)を紹介します.
創薬研究において合成難易度の評価が大切な理由
創薬研究においてリード候補化合物は
- ドラッグライクネス
- 予測される活性値
- 構造の新規性・特許の取りやすさ
など複数の観点から順位付けが行われます.
化合物が市販品やインハウスのライブラリーなどから取得された場合には,「合成可能性」は担保されていると言えます.一方で複雑な天然物由来の化合物やde novoデザインされた化合物は実際に合成できるかわかりません.
いずれにせよ,研究のある段階において化合物の「合成しやすさ」を考慮する必要があります.この際に,活性などの他の指標を優先して,最後に「合成しやすさ」を考慮してしまうと,どうしても似たようなケモタイプ・骨格の化合物が選ばれがちになります.こういった事態を防ぐためにはスクリーニングの初期段階から「合成可能性」も同等に扱って化合物評価を行っていくことが望ましいと言えます.
すなわち,大量の化合物の「合成しやすさ」を評価する簡便な方法が重要となります.
経験のある合成化学者ならば,化合物の構造を見ることで合成難易度を見極めることができますが,それでは何百万といった化合物のスクリーニングには対処できません.そこでコンピュータを用いて「合成しやすさ」を評価することが必要になります.
コンピュータを用いた合成難易度の評価方法
コンピュータに合成難易度を評価させる方法として,
- 分子構造の複雑さを基準とした方法
- 逆合成解析に基づいた方法
が考えられます.
前者は中員環・スピロ骨格・多数の不斉中心など,合成が難しい部分構造がある場合に難易度が高いと判断する方法,後者は実際に逆合成解析を行って組み立てた合成スキームを基に合成難易度を判断する手法です.
当然,後者の方が信頼性の高い指標と言えますが,計算負荷が大きく,また新しい合成方法論に対応するなど反応データベースのメンテナンスも大変です.
今回紹介する「SAスコア」も前者の方法論になります.
SAスコアとは
SAスコアは膨大な数の化合物を迅速に評価するために簡潔なルールに基づいて設計されました.基本的には分子の「複雑さ」を基準とした方法ですが,複雑な構造を瞬時に構築可能な試薬や反応の効果を取り込むために,「よく現れる部分構造は合成しやすい」という仮定を置いています.
具体的には,PubChemから取得した100万個の化合物のECFP4フィンガープリントの出現頻度をもとに重み付けを行っています.部分構造を出現頻度で重み付けした上で和を取ったものが,下式の「fragmentScore」になります.
$$ SAscore = fragmentScore – complexityPenalty $$
「complexityPenalty」は単純に環状構造,立体,大員環,分子量などの要素を考慮したものになります.詳細は論文を参照してください.
最後に1(易)から10(難)までの値に標準化を行います.
RDKitを使って化合物のSAスコアを評価する
必要なスクリプトファイルの入手
RDKitのコアモジュールには含まれていませんので,GiHubのContrib/SA_Scoreから必要なスクリプトとデータベースを入手してimportする必要があります.
下の写真で青枠で囲われている2つのファイルをダウンロードします.また「data」フォルダの中に,テスト計算用の分子構造(SMILES)と計算したSA Scoreが格納されたテキストファイル「zim.100.txt」があります.今回はそちらを使って,きちんと計算できているかどうかをチェックしましょう.
なおソースコードを眺めるとわかりますが,マクロサイクルや分子の対称性の扱い方が論文記載の方法から改良されています.
RDKitを使って必要な分子の準備
まずは必要なライブラリのimportと分子の準備を行います.今回は上記の「zim.100.txt」というファイルに保存されているSMILESから分子を構築していきます.このテキストファイルはタブ区切りになっていますのでpandasのデータフレームとして読み込んでいきます.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, PandasTools
from rdkit.Chem.Draw import IPythonConsole
import sascorer
import pandas as pd
import matplotlib as mpl
import matplotlib.pyplot as plt
%matplotlib inline
print(rdBase.rdkitVersion) ### 2018.09.1
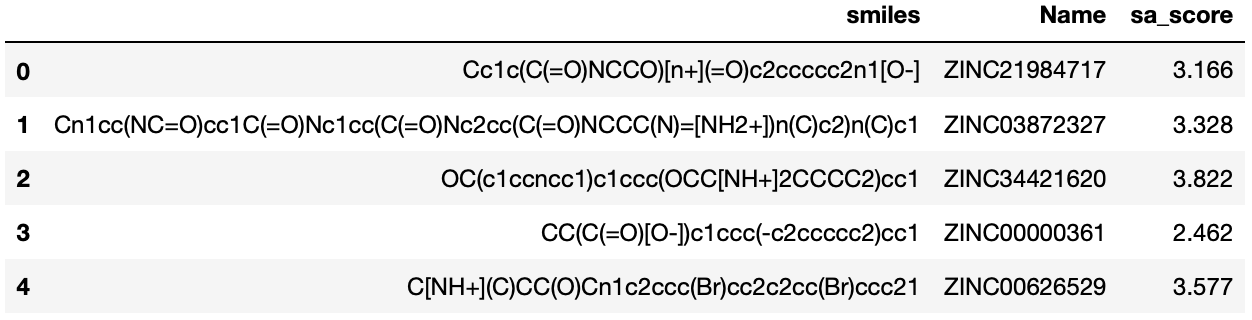
df = pd.read_csv('./zim.100.txt', sep='\t')
df.head()
データフレームは以下のようでした.
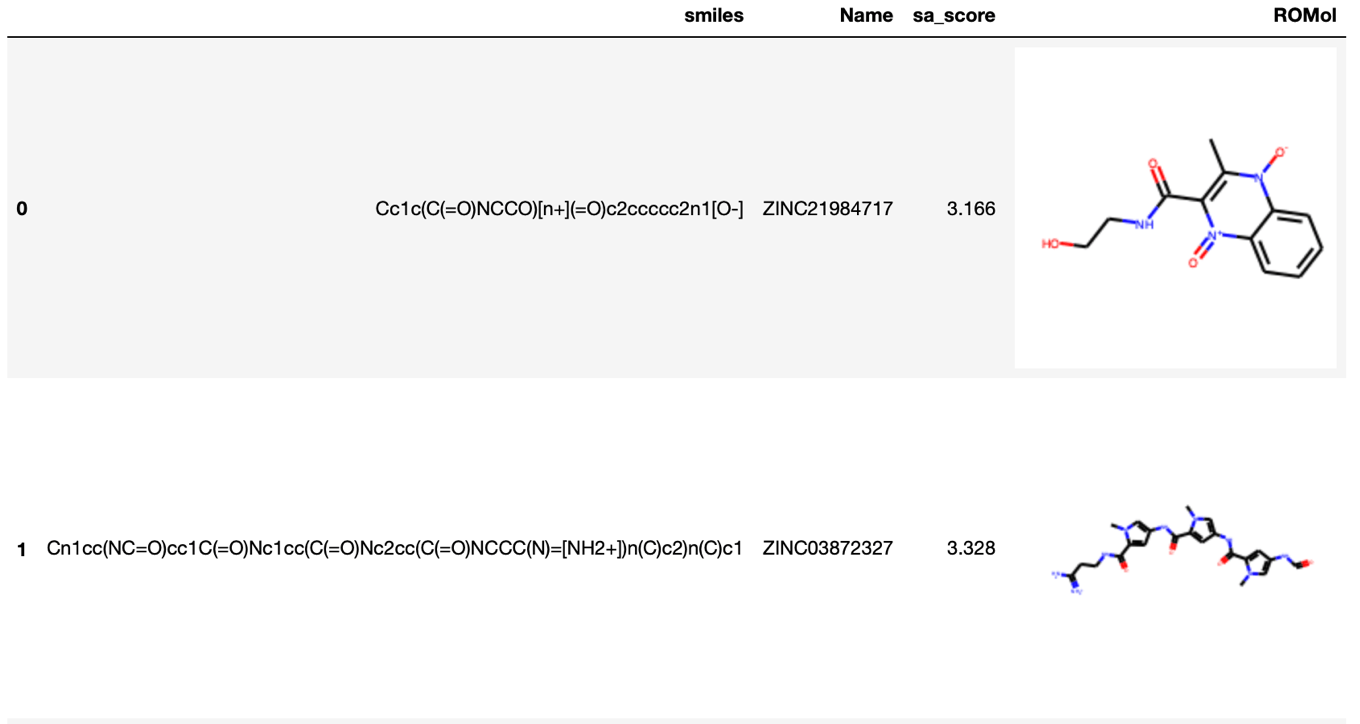
続いてデータフレームにAddMoleculeColumnToFrameメソッドを用いてRDKitのMOLオブジェクトを追加します.
PandasTools.AddMoleculeColumnToFrame(frame=df, smilesCol='smiles') df.head(2)
この段階のデータフレームは以下のようです.
RDKitで合成難易度の評価
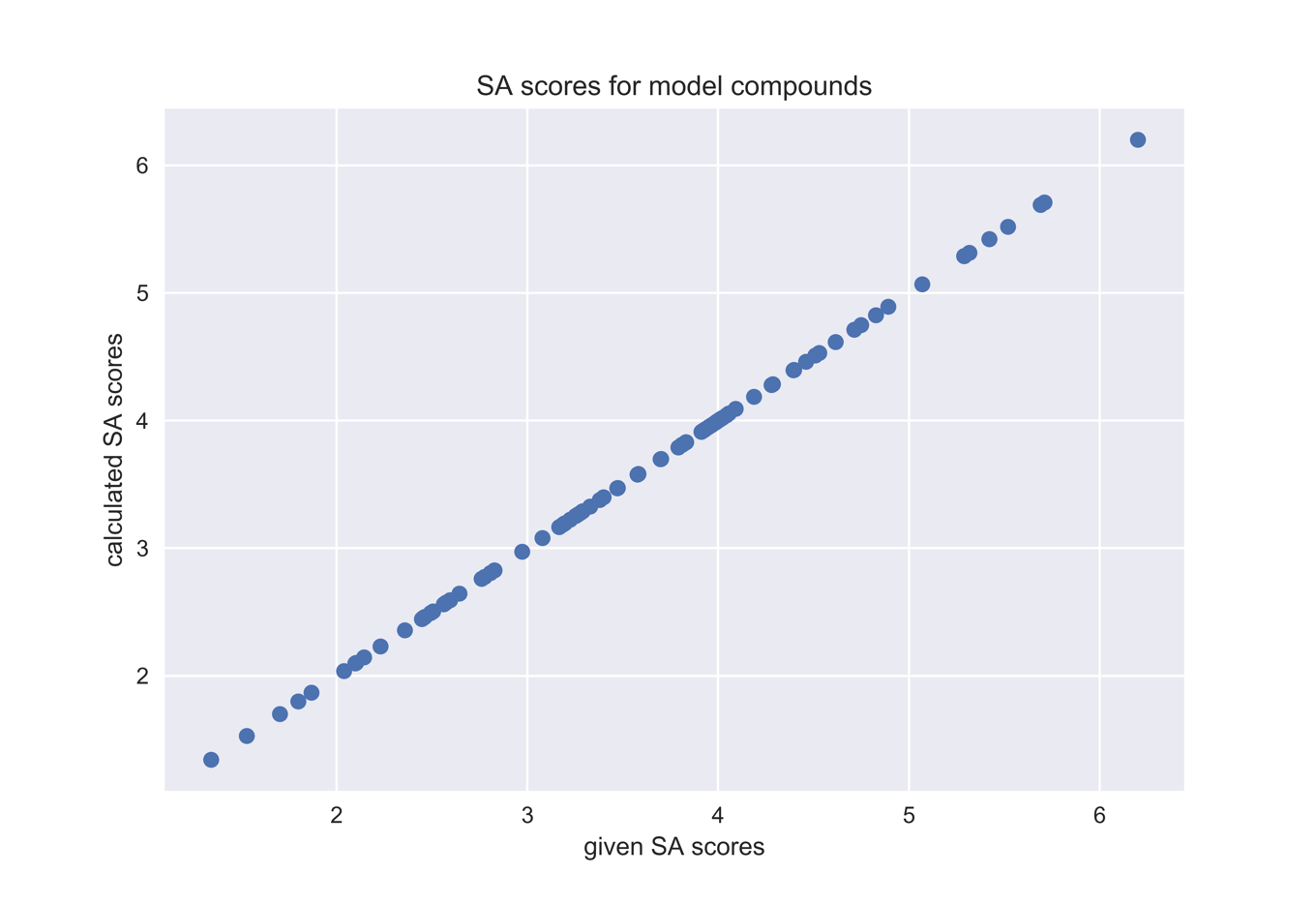
いよいよSAスコアを求めていきます.計算にはcalculateScoreメソッドを用います.今回はデータフレームのカラムに対して一括で計算を行いますので,map関数を利用しています.
下のコードではX軸にもともとファイルに入っていたスコアを,Y軸に今回計算したスコアをとってプロットしています.
df['calc_SA_score'] = df.ROMol.map(sascorer.calculateScore)
x = df.sa_score
y = df.calc_SA_score
with mpl.style.context('seaborn'):
plt.plot(x, y, 'o')
plt.xlabel('given SA scores')
plt.ylabel('calculated SA scores')
plt.title('SA scores for model compounds')
一直線に並んでいますので,きちんと計算できているようです.
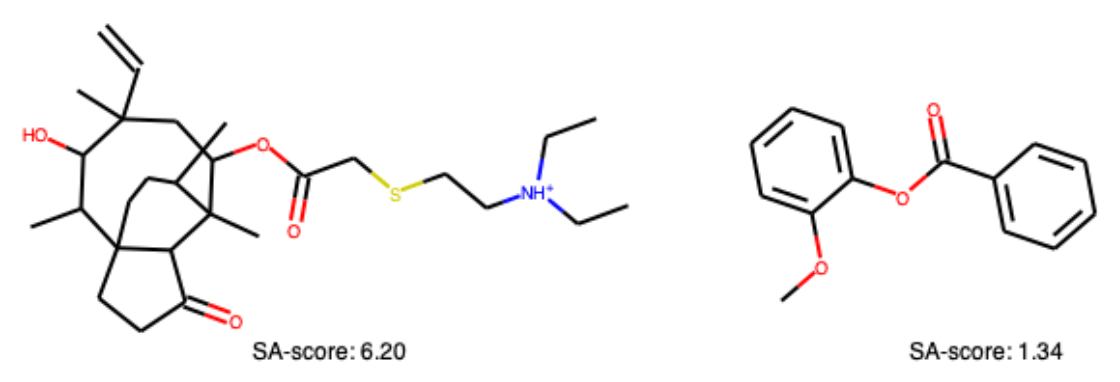
最後にSAスコアの最も高い(合成難易度の高い)化合物と低い化合物の構造を眺めてみます.
(id_max, id_min) = (df.sa_score.idxmax(), df.sa_score.idxmin())
sa_mols = [df.ROMol[id_max], df.ROMol[id_min]]
Draw.MolsToGridImage(sa_mols, subImgSize=(340,200),
legends=['SA-score: {:.2f}'.format(df.sa_score[i]) for i in [id_max, id_min]])
目で見ても構造の複雑さが異なるのがわかりますね.
終わりに
今回は「RDKitで合成難易度を評価して化合物をスクリーニング」という点について,
- なぜ「合成しやすさ」を考慮する必要があるのか
- SAスコアとは何か
- RDKitでのSAスコアの計算方法
という順番で解説してきました.
次回は化合物の「天然物らしさ」というものを考え,それをRDKitを用いてどのように評価するかについて説明していきたいと思います.
>>次の記事:「RDKitで天然物らしさをスコア化して化合物ライブラリーの指標にする」
コメント