「RDKitでフィンガープリントを使った分子類似性の判定」という記事では,ケモインフォマティクス用ライブラリであるRDKitで利用可能な分子フィンガープリントについて説明しました.
ECFP4(Morganフィンガープリント)などのCircularフィンガープリントでは,着目する原子を中心とした部分構造に焦点を当てて構築されており,化合物間の類似性評価や予測モデルにおける入力ベクトルとして高い有用性が示されています.
一方で部分構造に着目しているあまり,例えばペプチドなどの主構造に変化がなく側鎖の一部が変化している場合や,スキャホールドホッピングなど分子全体の形に着目したい場合にはあまり精度が出ないことも知られています.
今回の記事ではこういった場合にも有用であるフィンガープリントとして,分子中の原子ごとのペアに着目したアトムペアフィンガープリント(AP)を取り上げます.
具体的には,
- RDKitにおける実装と使い方
- アトムペアに含まれる情報
- 類似したフィンガープリント
などについて詳しく説明してきます.
アトムペアフィンガープリント
GetAtomPairFingerprintAsBitVect(mol)
IntSparseIntVect.GetNonzeroElements()
アトムペア(AP)フィンガープリントはChem.AtomPairsモジュール中の,Pairs.GetAtomPairFingerprintで求めることが可能です.またフィンガープリントもMorganフィンガープリントなどと同様に,GetAtomPairFingerprintAsBitVectを用いることでBitベクトルとしても取得可能です.
APフィンガープリントでは,
というフォーマットで分子中の全ての重原子の組合せを考えていきます.2つの原子距離は「最短結合数」です.このようにして得られた「原子ペアの情報」を一定のルールに基づいて数字で表していきます(コード化します).
APフィンガープリントはIntSparseIntVectオブジェクトとして得られますが,そのGetNonzeroElementsメソッドを用いることで「数字:個数」から形成される辞書オブジェクトが得られます.
以下に例では「689187」でコードされるアトムペアが分子内に2つあることを示しています.
{
689187: 2,
689188: 2,
689189: 1,
・・・
}
この個数の総和(アトム組合せの数)は,重原子の数をnとすると
$$ \frac{n(n-1)}{2} $$
になります.
局所的な部分構造のみを考えるECFP4などと異なり,APフィンガープリントでは遠く離れた原子との関係も情報として取り込むことができます.APフィンガープリントは広いタイプの分子で異なる出力を与えますが,以下のような分子では全く同じフィンガープリントになることも知られています.
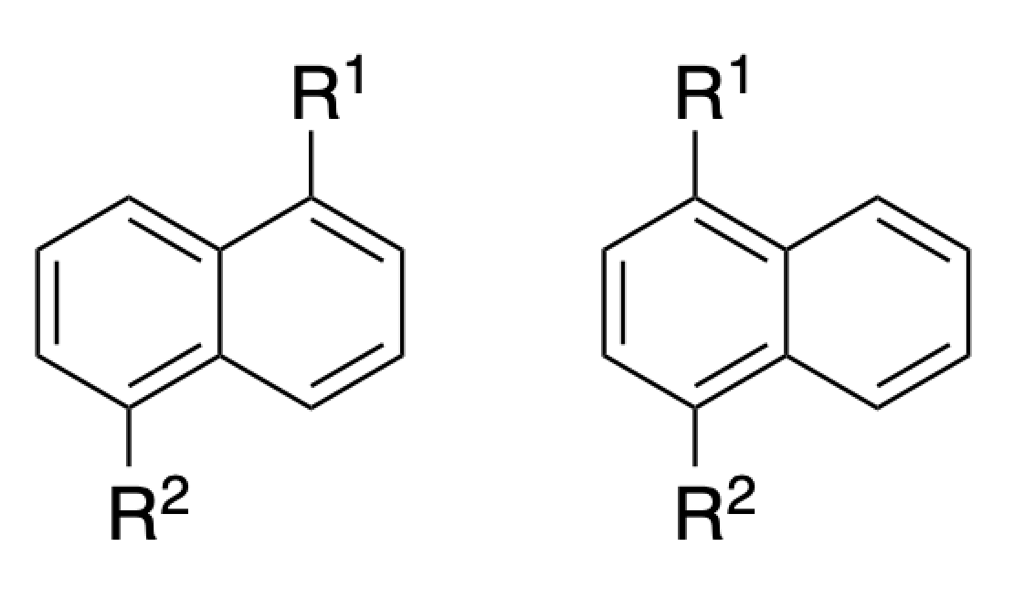
以下では
- アトムタイプの分類
- アトムタイプのコード化
についてさらに詳しく見ていくことで「689187」などのコードの中身を解き明かしていきます.
“Atom pairs as molecular features in structure-activity studies: definition and applications” J. Chem. Inf. Comput. Sci. 1985, 25, 64-73.
アトムタイプの分類とコード化
Chem.AtomPairs.Utils.ExplainAtomCode(code)
アトムタイプはAtomPairs.Utilsモジュールにある関数で行えます.
- GetAtomCode:指定した原子のアトムタイプをコード(数字)で表したもの
- ExplainAtomCode:コードの意味するアトムタイプ
をそれぞれ返す関数です.
アトムタイプは
- 原子の種類(炭素,窒素,酸素など)
- 隣接する重原子の数
- π結合の数
の3つの要素から算出され,これらを組み合わせた(C, 2, 1)などのように表記します.このうちπ結合の数は,
- 二重結合(芳香族結合)が1つなら1
- 三重結合が1つなら2
というようにカウントされます.
以下に1つ例を示します.
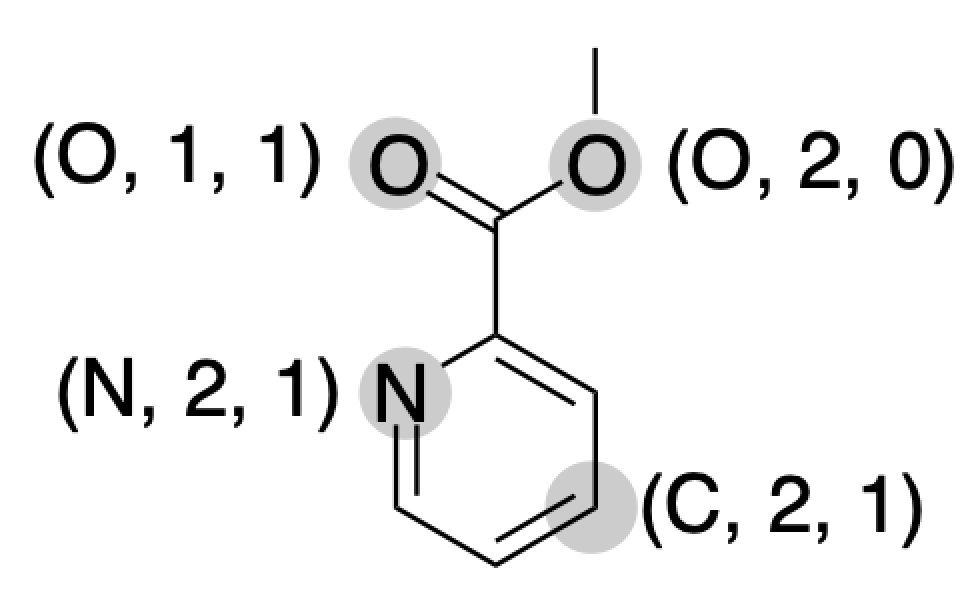
このようにして得たアトムタイプを
$$ atomcode = 32 \times 原子の種類 + 隣接重原子の数 + 8 \times π結合の数 $$
を用いて数字に変換しています.なお原子の種類は
| 原子 | 数字 |
|---|---|
| B | 0 |
| C | 1 |
| N | 2 |
| O | 3 |
| F | 4 |
| Si | 5 |
| P | 6 |
| S | 7 |
| Cl | 8 |
| As | 9 |
| Se | 10 |
| Br | 11 |
| Sb | 12 |
| Te | 13 |
| Tc | 14 |
| その他 | 15 |
というルールでコード化されているようです.
先ほどの分子の場合以下のようになります.
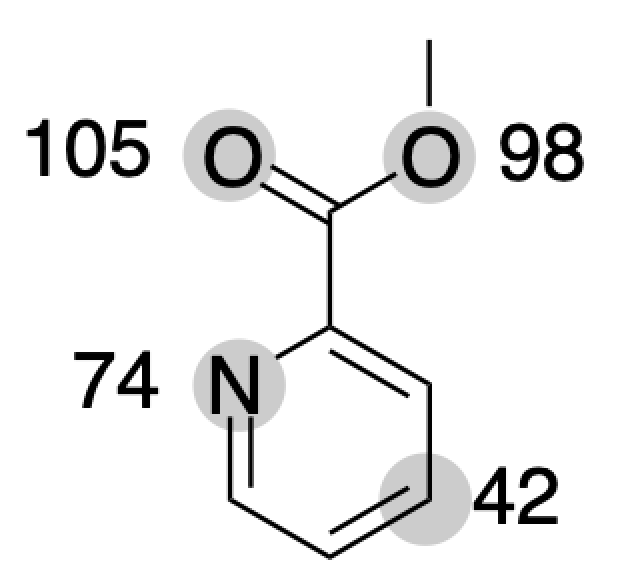
下記のサンプルコードでは,
- 元素記号
- コード
- アトムタイプの情報
を分子内の全原子について出力しています.
from rdkit import Chem
from rdkit.Chem.AtomPairs import Pairs, Utils
mol = Chem.MolFromSmiles('n1c(C(=O)OC)cccc1')
for i,atom in enumerate(mol.GetAtoms()):
symbol = atom.GetSymbol()
code = Utils.GetAtomCode(mol.GetAtomWithIdx(i))
explained = Utils.ExplainAtomCode(code)
print(symbol, code, explained)
N 74 ('N', 2, 1)
C 43 ('C', 3, 1)
C 43 ('C', 3, 1)
O 105 ('O', 1, 1)
O 98 ('O', 2, 0)
C 33 ('C', 1, 0)
C 42 ('C', 2, 1)
C 42 ('C', 2, 1)
C 42 ('C', 2, 1)
C 42 ('C', 2, 1)
アトムペア表記とスコア化
Chem.AtomPairs.Pairs.ExplainPairScore(score)
前述のようにして得たアトムタイプの情報を用いて,2つの原子のペア情報を取得していきます.AtomPairs.Pairsモジュールにある関数を用いて行います.
- pyScorePair:指定した2つの原子に対して,指定した距離のコードのスコア
- ExplainPairScore:スコアの意味するアトムペアの情報
をそれぞれ返す関数です.
アトムペア情報のスコア化には
$$ score = 16384 \times max(原子{1,2}コード) + 32 \times min(原子{1,2}コード) + 距離 $$
という式が使われています.
下の例ではピリジン窒素(N, 2, 1)とエステル酸素(O, 2, 0)の距離は3であり,スコアは
$$ 16384 \times 98 + 32 \times 74 + 3 = 1608003 $$
と計算できます.
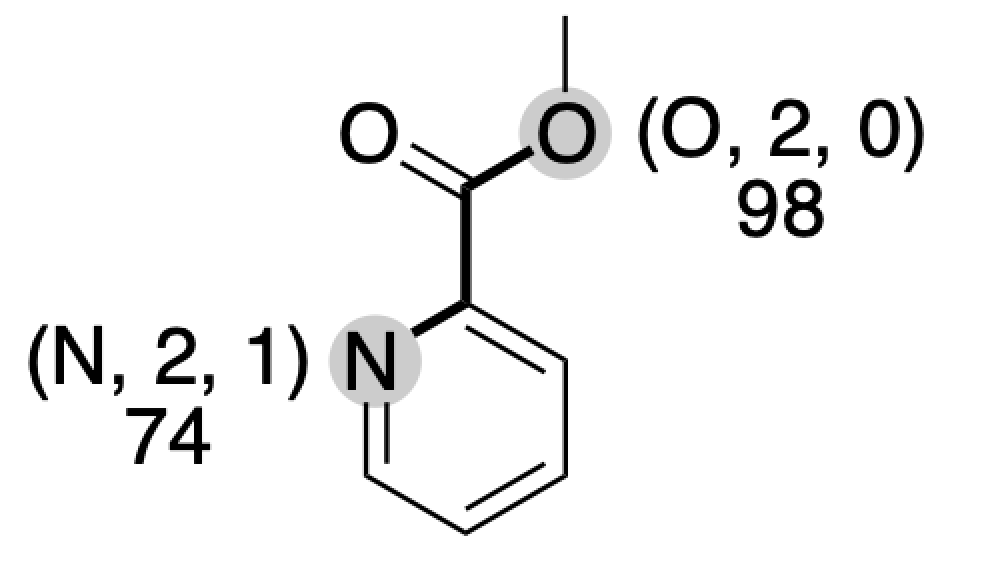
全ての組合せを計算したサンプルコードは以下です.
ap = Pairs.GetAtomPairFingerprint(mol)
d = ap.GetNonzeroElements()
for key in d.keys():
explained = Pairs.ExplainPairScore(key)
print(key, explained)
689188 (('C', 1, 0), 4, ('C', 2, 1))
689189 (('C', 1, 0), 5, ('C', 2, 1))
689190 (('C', 1, 0), 6, ('C', 2, 1))
689473 (('C', 2, 1), 1, ('C', 2, 1))
689474 (('C', 2, 1), 2, ('C', 2, 1))
689475 (('C', 2, 1), 3, ('C', 2, 1))
705570 (('C', 1, 0), 2, ('C', 3, 1))
705571 (('C', 1, 0), 3, ('C', 3, 1))
705857 (('C', 2, 1), 1, ('C', 3, 1))
705858 (('C', 2, 1), 2, ('C', 3, 1))
705859 (('C', 2, 1), 3, ('C', 3, 1))
705860 (('C', 2, 1), 4, ('C', 3, 1))
705889 (('C', 3, 1), 1, ('C', 3, 1))
1213476 (('C', 1, 0), 4, ('N', 2, 1))
1213761 (('C', 2, 1), 1, ('N', 2, 1))
1213762 (('C', 2, 1), 2, ('N', 2, 1))
1213763 (('C', 2, 1), 3, ('N', 2, 1))
1213793 (('C', 3, 1), 1, ('N', 2, 1))
1213794 (('C', 3, 1), 2, ('N', 2, 1))
1606689 (('C', 1, 0), 1, ('O', 2, 0))
1606979 (('C', 2, 1), 3, ('O', 2, 0))
1606980 (('C', 2, 1), 4, ('O', 2, 0))
1606981 (('C', 2, 1), 5, ('O', 2, 0))
1607009 (('C', 3, 1), 1, ('O', 2, 0))
1607010 (('C', 3, 1), 2, ('O', 2, 0))
1608003 (('N', 2, 1), 3, ('O', 2, 0))
1721379 (('C', 1, 0), 3, ('O', 1, 1))
1721667 (('C', 2, 1), 3, ('O', 1, 1))
1721668 (('C', 2, 1), 4, ('O', 1, 1))
1721669 (('C', 2, 1), 5, ('O', 1, 1))
1721697 (('C', 3, 1), 1, ('O', 1, 1))
1721698 (('C', 3, 1), 2, ('O', 1, 1))
1722691 (('N', 2, 1), 3, ('O', 1, 1))
1723458 (('O', 2, 0), 2, ('O', 1, 1))
アトムペアのコードやスコアの意味
ここではアトムタイプのコードやアトムペアのスコアについてもう少し見てみます.
まず少し書き直したアトムコードの変換式を以下に示します.
$$ atomcode = 2^5 \times 原子の種類 + 2^3 \times π結合の数 + 隣接重原子の数 $$
続いて74でコードされる(N, 2, 1)を2進数で表した’0b1001010‘と比べてみましょう.
| 原子の種類 | π結合の数 | 隣接重原子の数 |
|---|---|---|
| N(=2) | 1 | 2 |
| 10 | 010 | 10 |
98でコードされる(O, 2, 0)も見てみます.2進数表記は’0b1100010‘です.
| 原子の種類 | π結合の数 | 隣接重原子の数 |
|---|---|---|
| O(=3) | 0 | 2 |
| 11 | 000 | 10 |
わかったでしょうか?25 や23 を使うことで2進数における桁をずらして3つの情報を表記しているわけです.
アトムペアのスコアで使われている214 (=16384)や25 (=32)も同様です.
$$ score = 2^{14} \times max(原子{1,2}コード) + 2^5 \times min(原子{1,2}コード) + 距離 $$
例えば(O, 2, 0)と(N, 2, 1)の距離は3のスコアである1608003の2進数表記は’0b110001000100101000011‘です.
| 原子1の種類 | π結合の数 | 隣接重原子の数 | 原子2の種類 | π結合の数 | 隣接重原子の数 | 距離 |
|---|---|---|---|---|---|---|
| O(=3) | 0 | 2 | N(=2) | 1 | 2 | 3 |
| 11 | 000 | 10 | 0010 | 010 | 10 | 00011 |
トポロジカル二面角フィンガープリント
Chem.AtomPairs.Torsions.ExplainPathScore(score)
APフィンガープリントで用いられるアトムタイプの情報を使って,分子内の二面角に対して適用したものがトポロジカル二面角(TT)フィンガープリントです.
二面角を形成する4つの原子に対して以下のように与えられます.
APフィンガープリントととは異なり,こちらのフィンガープリントは局所的な構造のみを取り込んだフィンガープリントになります.
RDKitによる使い方はほぼAPフィンガープリントと同じです.Chem.AtomPairs.Torsionsモジュールを用いることで利用可能です.以下のコードでは分子内の全ての二面角について情報を取得しています.
from rdkit.Chem.AtomPairs import Torsions
tt = Torsions.GetTopologicalTorsionFingerprint(mol)
for torsion in tt.GetNonzeroElements().keys():
print(torsion, '\t', Torsions.ExplainPathScore(torsion))
5513433129 (('C', 2, 1), ('C', 2, 1), ('C', 2, 1), ('C', 2, 1))
5521822249 (('C', 2, 1), ('C', 3, 1), ('N', 2, 1), ('C', 2, 1))
5647650857 (('C', 2, 1), ('C', 2, 1), ('C', 2, 1), ('C', 3, 1))
5647913001 (('C', 2, 1), ('C', 2, 1), ('C', 3, 1), ('C', 3, 1))
5647929385 (('C', 2, 1), ('N', 2, 1), ('C', 3, 1), ('C', 3, 1))
5647941664 (('C', 1, 0), ('O', 2, 0), ('C', 3, 1), ('C', 3, 1))
5656039465 (('C', 2, 1), ('C', 2, 1), ('N', 2, 1), ('C', 3, 1))
9808400425 (('C', 2, 1), ('C', 2, 1), ('C', 2, 1), ('N', 2, 1))
9808662569 (('C', 2, 1), ('C', 2, 1), ('C', 3, 1), ('N', 2, 1))
13029888553 (('C', 2, 1), ('C', 3, 1), ('C', 3, 1), ('O', 2, 0))
13029888585 (('N', 2, 1), ('C', 3, 1), ('C', 3, 1), ('O', 2, 0))
13969412649 (('C', 2, 1), ('C', 3, 1), ('C', 3, 1), ('O', 1, 1))
13969412681 (('N', 2, 1), ('C', 3, 1), ('C', 3, 1), ('O', 1, 1))
13969440800 (('C', 1, 0), ('O', 2, 0), ('C', 3, 1), ('O', 1, 1))
“Topological torsion: a new molecular descriptor for SAR applications. Comparison with other descriptors” J. Chem. Inf. Comput. Sci. 1987, 27, 82-85.
ドナーアクセプターペアフィンガープリント
APフィンガープリントを用いることで分子全体の情報を取り入れることができました.一方で「アトムタイプの情報」を主流としていることから,例えば生物学的等価体とされるカルボン酸とテトラゾールのような化学的性質が似ている構造の類似性を評価できません.
ドナーアクセプターペア(DP)フィンガープリントは,アトムペアの情報として
- 水素結合供与体
- 水素結合受容体
といった一般性のある「化学的性質」を情報として用いています.
DPフィンガープリントはAPフィンガープリントを代替するものというよりは,2つを同時に用いることで分子の性質をよりよく表現できると考えるべきです.
具体的には分子中の原子を以下の7種類に分けます.
| 原子の性質 | RDKitの記号 |
|---|---|
| カチオン | CAT |
| アニオン | ANI |
| 中性水素結合性供与体 | DON |
| 中性水素結合受容体 | ACC |
| 極性原子 | POL |
| ハイドロフォービック | HYD |
| その他 | OHT |
“Chemical Similarity Using Physiochemical Property Descriptors” J. Chem. Inf. Comput. Sci. 1996, 36, 118-127.
RDKitにおけるドナーアクセプターフィンガープリントの実装
Chem.AtomPairs.Sheridan.GetBTFingerprint(mol)
Chem.AtomPairs.Sheridan.AssignPattyTypes(mol)
RDKitではDPフィンガープリントはChem.AtomPairs.Sheridanモジュールに実装されています.
- GetBPFingerprint:アトムペアに相当するフィンガープリントを作成
- GetBTFingerprint:トポロジカル二面角に相当するフィンガープリントを作成
各原子のタイプ分けはAssignPattyTypesで確認できます.

from rdkit.Chem.AtomPairs import Sheridan
mol2 = Chem.MolFromSmiles('ONC(=O)C(=O)NCCN')
Sheridan.AssignPattyTypes(mol2)
['POL', 'DON', 'OTH', 'ACC', 'OTH', 'ACC', 'DON', 'HYD', 'HYD', 'CAT']
終わりに
今回は「アトムペアやドナーアクセプターペアフィンガープリントは分子の形状を捉えた化学表現」という話題について,
- アトムタイプの情報に関する詳細
- アトムペアフィンガープリント
- トポロジカル二面角フィンガープリント
- ドナーアクセプターペアフィンガープリント
などについて説明していきました.特にアトムペアフィンガープリントで用いられるアトムタイプについては詳しく触れました.
記述子やフィンガープリントを利用した分子のベクトル化はケモインフォマティクス研究において長い歴史を有しますが,全ての目的に使える表現方法は未だ存在せず,今でも新しい方法が開発・報告されています.それぞれの特徴と限界を理解することで,目的に合わせて適切な手法を選べるようにしていきたいものです.

コメント