当サイトでは「有機合成化学者のためのケモインフォマティクス入門」を掲げています.高価なソフトを使うこと無く学習をはじめられるように,これまでpythonのケモインフォマティクスライブラリである「RDKit」の解説を,「RDKitでケモインフォマティクスに入門」という記事から10回以上に渡って行ってきました.
すでにRDKitの基本的な使い方から,ケモインフォマティクスで用いられる色々なコンセプトを見てきました.その過程で扱った化合物データは,pythonの基本データ型であるリストや辞書型の変数,またはnumpy配列に格納して取り扱うことがほとんどでした.
一方で,pythonを用いたデータサイエンス・探索的なデータ分析ではpandasのSeriesやDataFrameというデータ型を用いるのが主流です.今回はRDKitのMolオブジェクトをpandasのDataFrameでどのように扱うかについて学んでいくことで,このギャップを埋めていきます.
pandasとは
「pandas(読み方:パンダス)」はパネルデータを扱うためのpythonライブラリで,pythonデータサイエンスの中心となるデータ型とそれに付随する様々な操作を提供するライブラリになります.特に数値データと時系列データにおける異常値・欠損値の扱いに長けています.またnumpyでは均一な数値データの配列のみしか扱えないのに対し,pandasは不均一なデータを取り扱える点が特長になります.
とても多機能で巨大なライブラリになりますので,全ての機能を把握するのは困難です.必要なところから徐々に慣れていくのがよいと思います.
RDKitのPandasToolsについて
pandasでは数値データや時系列データの扱いは得意ですが,化合物データを扱うためのライブラリではありませんので,当然ですが分子を扱う際に便利な機能は提供されていません.RDKitの「Chem.PandasTools」は,MolオブジェクトをpandasのDataFrameで扱うための拡張機能を提供するものになります.
まずは必要なライブラリのimportを行い,それから具体的にPandasToolsの使い方を見ていきましょう.
from rdkit import rdBase, Chem, DataStructs
from rdkit.Chem import AllChem, Draw, Descriptors, PandasTools
import pandas as pd
print('rdkit version: ',rdBase.rdkitVersion)
print('pandas version: ', pd.__version__)
### rdkit version: 2018.03.3
### pandas version: 0.23.4
Molオブジェクトのデータフレームへの読み込み
PandasToolsを使ったMolオブジェクトのデータフレームへの読み込み方法には大きく
- SDFからMolオブジェクトを構築
- SMILESからMolオブジェクトを構築
の2つの方法があります.前者はデータフレームを1から作る方法,後者はSMILESを含む既存のデータフレームにMOLオブジェクトを格納するカラムを追加する方法です.
SDFのpandasデータフレームへの読み込み
SDFを直接pandasのデータフレームとして読み込むにはPandasTools.LoadSDFの利用が最も便利です.このメソッドを用いるとSDFに設定されているプロパティがカラムに登録されます.肝心のMolオブジェクトは「ROMol」というカラムに格納されます.
デフォルト設定ではROMolに登録された分子はプロパティを保持していませんが,embedPropsオプションをTrueにすることでプロパティを付加できます.
またMolオブジェクトを作成せずに,SDF中のプロパティだけを取り込んだデータフレームを作成したい場合には,molColName=Noneオプションを設定します.
下の例では東京化成より入手したサリチル酸誘導体のSDFを読み込んでいます.
df = PandasTools.LoadSDF('./sdf_20180716131641.sdf')
df.MOLECULAR_WEIGHT = df.MOLECULAR_WEIGHT.astype(float)
df.columns
CAS番号,分子式,カタログ番号をはじめとしたプロパティがデータフレームに読み込まれているのがわかります.またプロパティは全て「文字列」として認識されていますので,分子量だけはastypeを用いてfloat型へと変換しました.
Index(['CAS_NUMBER', 'FREEZING_POINT', 'ID', 'MDL_NUMBER', 'MELTING_POINT', 'MOLECULAR_FORMULA', 'MOLECULAR_WEIGHT', 'PRODUCT_NAME', 'PRODUCT_NUMBER', 'ROMol'], dtype='object')
SMILESからMolオブジェクトを構築する
既にSMILESを含めて化合物データが色々と登録されているデータフレームにMolオブジェクトを登録したい場合には,PandasTools.AddMoleculeColumnToFrameを使います.このメソッドは名前のとおり,SMILESカラムを指定することでMolオブジェクトを生成し,既存のデータフレームに追加します.
データフレームを用いた分子の操作
それでは実際に分子を使った操作をしてみたいと思います.特にPandasTools特有の機能に限定することなく説明していきます.
mapまたはapplyを用いた記述子の一括計算
DataFrame.apply(function or dictionary, axis)
データフレーム中のMolオブジェクトに対して,一括で記述子を計算する場合などにはpandasのmapまたはapplyメソッドを使うとよいです.mapはSeriesに対して,applyはデータフレームの行または列に対して使います.
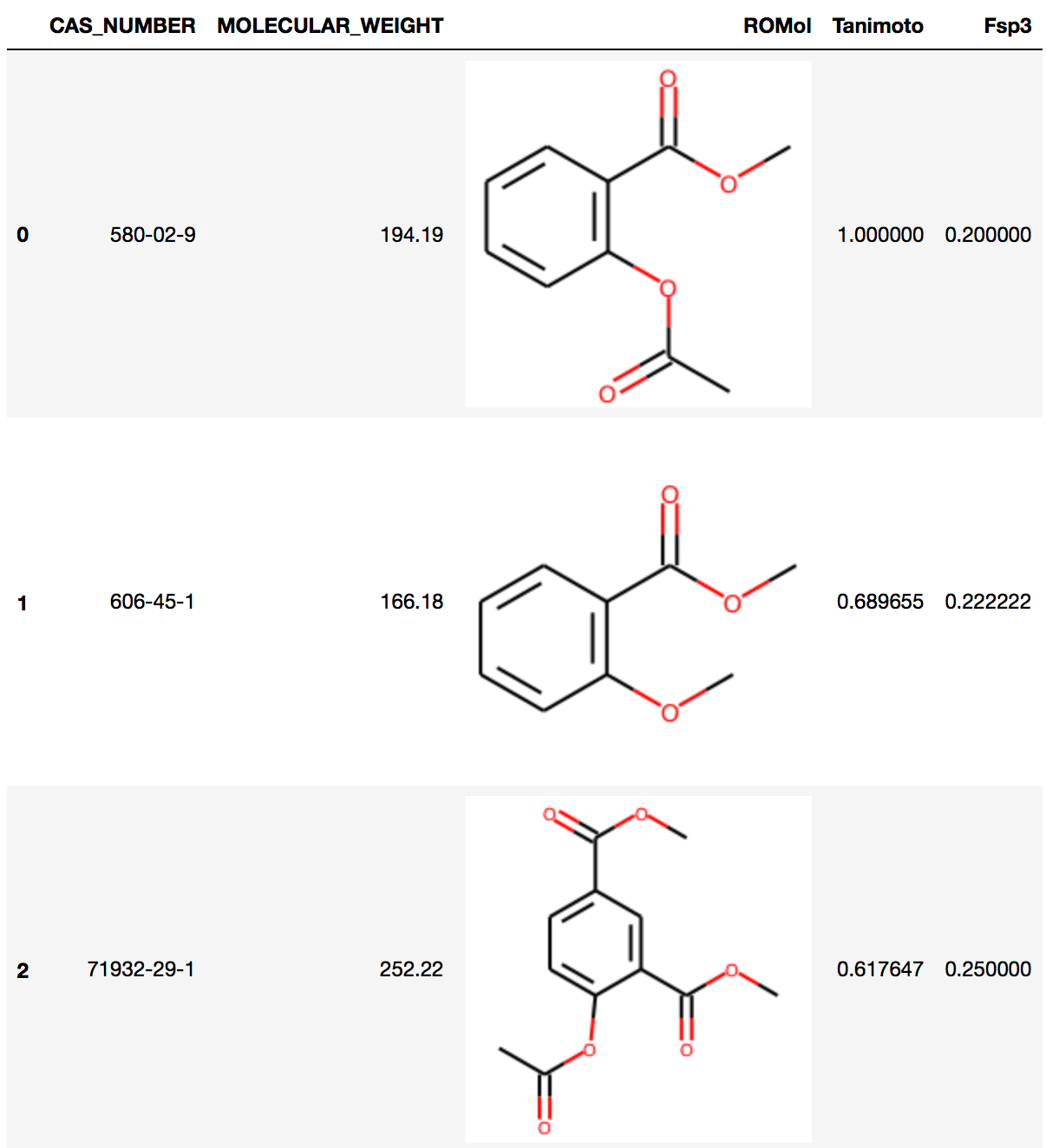
以下の例ではmapを使ってFsp3を,applyを使ってMorganフィンガープリントを計算しています.その後,タニモト係数を計算し,いずれも新しいカラムに保存しています.
df['Fsp3'] = df.ROMol.map(Descriptors.FractionCSP3) df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x['ROMol'], 2, 2048), axis=1) df['Tanimoto'] = DataStructs.BulkTanimotoSimilarity(df.FP[0], df.FP) df[['CAS_NUMBER', 'MOLECULAR_WEIGHT', 'ROMol', 'Tanimoto', 'Fsp3']].head(3)
ここまでのデータフレームの中身は下のようになっています.
ge比較演算子を用いた部分構造検索
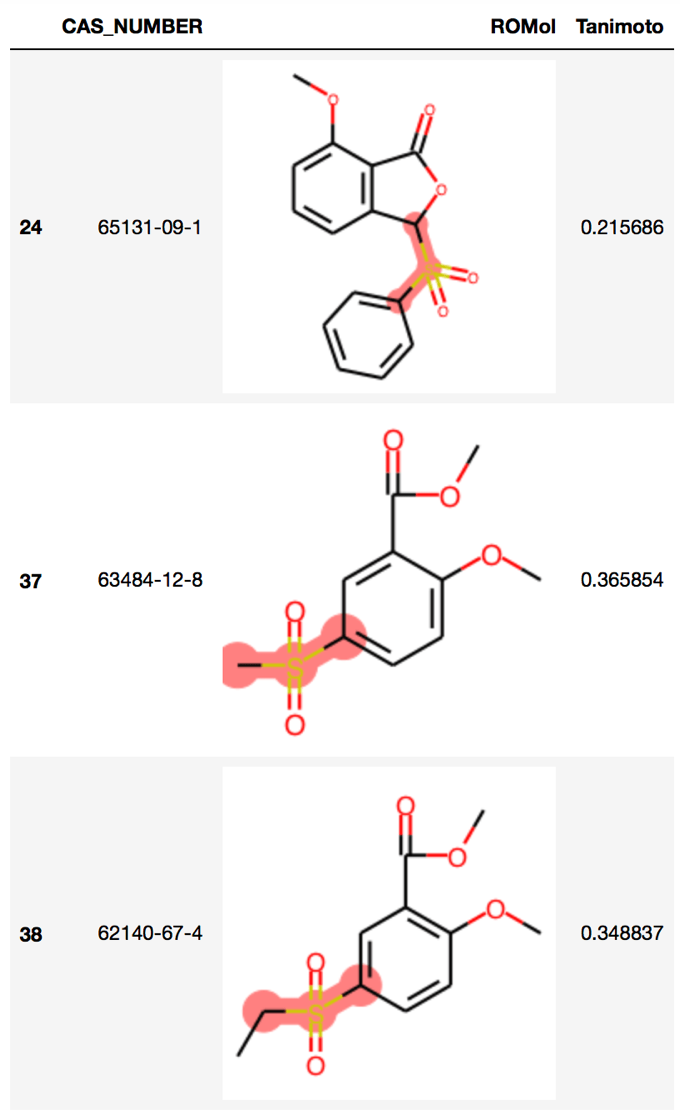
PandasToolsの一番の拡張機能は,「>=(ge比較演算子)」を用いることで部分構造マッチが行える点にあります.Mol.HasSubstructMatchを用いるよりも,ずっと簡単に分子構造のスクリーニングが可能になります.
下の例ではスルホン部位を有する分子を取り出してみました.
CSO2C = Chem.MolFromSmiles('CS(=O)(=O)C')
df[df.ROMol >= CSO2C][['CAS_NUMBER', 'ROMol', 'Tanimoto']]
Bemis・Murcko骨格の追加
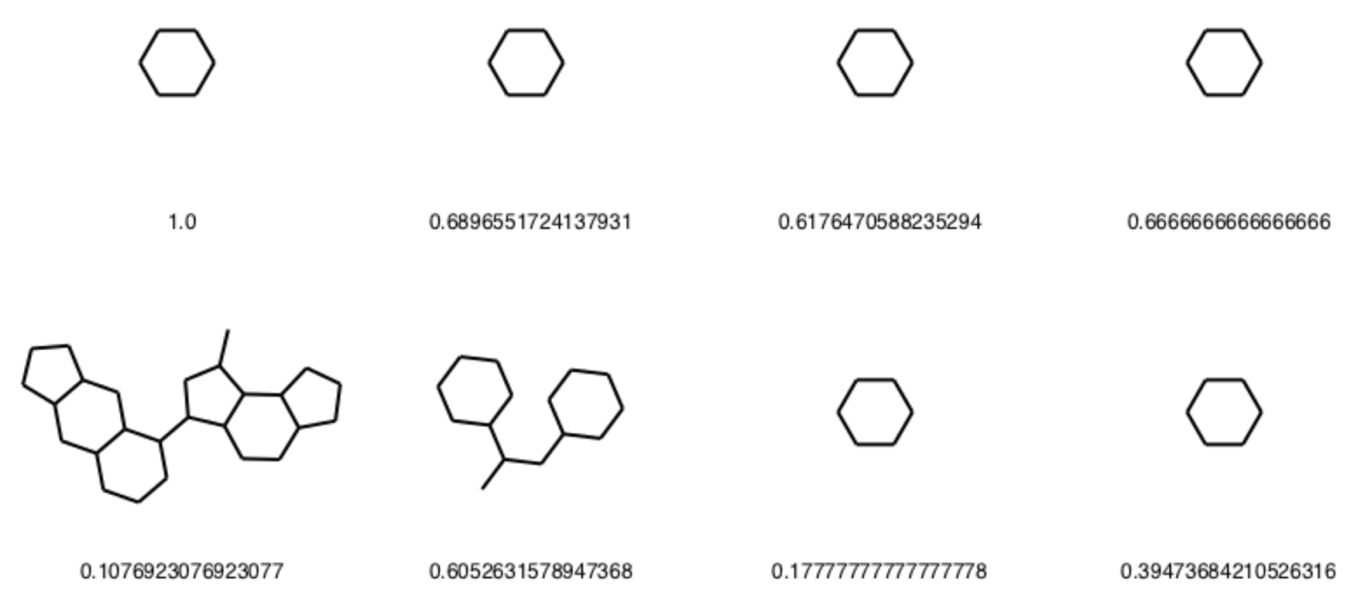
データフレーム中のMolオブジェクトを基にBemis・Murckoらによる方法を使って分子の主骨格をSMILES形式で取り出したカラムを追加します.Generic=Tureオプションを設定することでへテロ元素を炭素に置換した骨格に変更可能です.
下の例では,
- 主骨格のSMILESを格納したカラムをAddMurckoToFrameを用いて作成
- AddMoleculeColumnToFrameによってMolオブジェクトへと変換
- FrameToGridImageメソッドを用いて,データフレーム中のMolオブジェクトの構造を描画
という3ステップを行っています.
PandasTools.AddMurckoToFrame(df, MurckoCol='Murcko_SMILES') PandasTools.AddMurckoToFrame(df, MurckoCol='Murcko_generic_SMILES', Generic=True) PandasTools.AddMoleculeColumnToFrame(df, molCol='Murcko_Mol', smilesCol='Murcko_SMILES') PandasTools.AddMoleculeColumnToFrame(df, molCol='Murcko_generic_Mol', smilesCol='Murcko_generic_SMILES') PandasTools.FrameToGridImage(df[:8], column='Murcko_generic_Mol', legendsCol='Tanimoto', molsPerRow=4)
データフレームからファイルへの書き出し
PandasToolsではデータフレームから分子情報の他のファイルへの書き出しも可能です.
- SDF
- SMILES
- XLS(エクセルファイル)
という3形式に対応しています.
SDF
SDFへの書き出しにはWriteSDFを用います.データフレームと書き出すSDFのファイル名を引数にとり,さらに追加の設定として,
- idNameでカラム名を指定することでSDFのタイトル
- propertiesでSDFに追加するプロパティのリスト
をオプションで設定可能です.
SMILES
SaveSMILESFromFrameを使うことで,データフレーム中のMolオブジェクトカラムからSMILESを生成して保存することが可能です.
エクセル形式
Molオブジェクトの構造を画像ファイルとして埋め込んだ上で,データフレームをエクセル形式で保存します.「xlsxwriter」に依存していますので,利用に先立ってインストールしておきましょう.
下の例ではまずは不要なカラムを削除した後に,主骨格をMolオブジェクトに指定して,画像ファイルをやや小さめにして保存しています.
ddf = df.drop(['FREEZING_POINT', 'ID', 'MELTING_POINT', 'ROMol', 'FP', 'Murcko_generic_Mol'], axis=1) PandasTools.SaveXlsxFromFrame(ddf, './sample.xls', molCol='Murcko_Mol', size=(150,150))
このようなExcelファイルが作成されました.
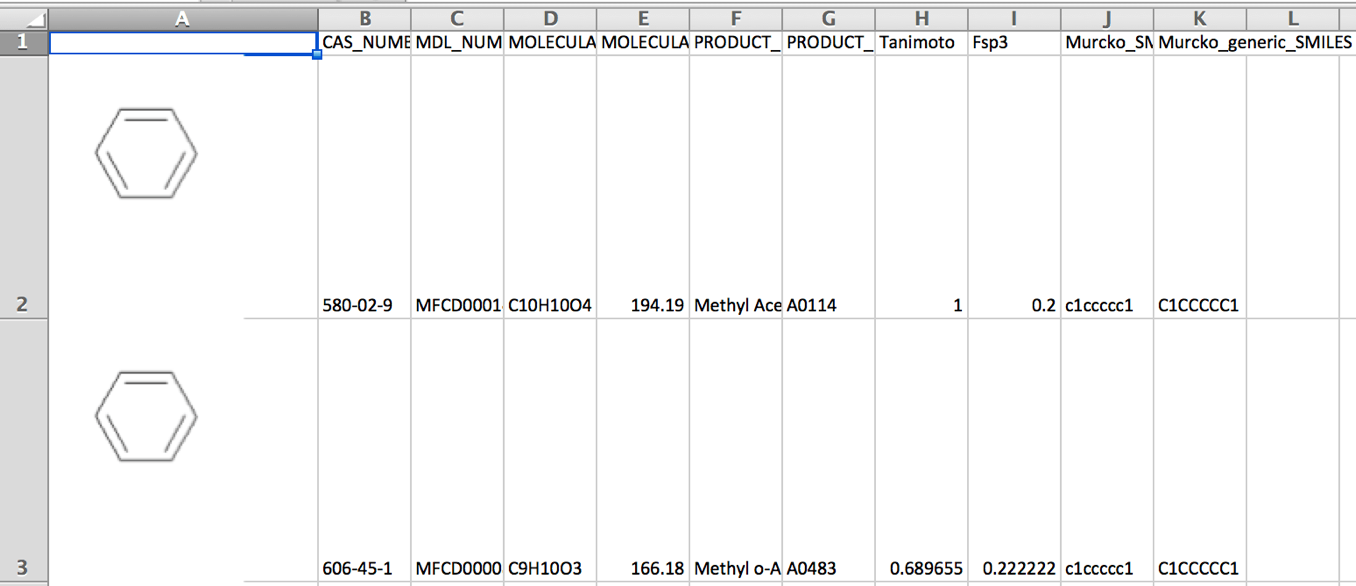
ありがちだと思いますが私の周りでは「Excel最強」ですので,他人との化合物データのやり取りにエクセルを扱うことが多々あります.特にChemDrawで描いた構造をExcelのセルに貼り付ける作業を繰り返して化合物管理をしているようなプロジェクトでは非常に重宝するのではないかと思います.10年前の自分に是非とも教えてあげたい機能です.
終わりに
今回はpythonにおけるデータ分析作業の中心をなすpandasとRDKitとの架け橋となるPandasToolsについて説明してきました.PandasToolsを使うことで,Molオブジェクトが簡単にデータフレームに取り込めること,および部分構造検索が容易に行えることなどを見てきました.
pandas自体は非常に多機能なライブラリですので,今回紹介できなかった役立つ機能がたくさんあります.是非とも公式サイト「Python Data Analysis Library」を読み込んで,理解を深めていってください.
これまで本サイトでpythonの基本データ型を使って行ってきた作業を,pandasのデータフレームに置き換えてみるのもよい練習になるかもしれません.
>>次の記事:「RDKitの分子Molオブジェクトを扱う」
コメント