「RDKitでフィンガープリントを使った分子類似性の判定」という記事では分子の特徴を表現するフィンガープリントについて学び,タニモト係数などを利用して分子同士の類似度を判定する方法を学びました.さらには原子ごとの類似度への寄与率を類似度マップを用いて可視化する方法も扱いました.
今回は,同様の手法を使うことで特定の記述子に対する原子ごとの寄与を可視化してみます.類似度マップ(SimilarityMaps)を用いますが,原子ごとの寄与をもとに可視化するだけで,分子の類似度とは関係ないことに注意してください.
必要なモジュールのimportと分子の準備
まずは必要なライブラリのimportと分子の構築を行いましょう.今回は有機化学では有名なタキソール(パクリタキセル)を扱ってみます.「pythonで化合物データベースPubChemを使いこなす」という記事で説明したようにPubChemからタキソール(パクリタキセル)を検索し,Smiles表記の取得を行います.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, rdMolDescriptors
from rdkit.Chem.Draw import SimilarityMaps
print('rdkit version: ', rdBase.rdkitVersion)
# rdkit version: 2020.03.1
# pubchempyで'Paclitaxel'を検索
import pubchempy as pcp
taxol = pcp.get_compounds('Paclitaxel', 'name')
taxol = taxol[0]
# RDKitでMolオブジェクトの作成
sm = taxol.canonical_smiles
taxol_mol = Chem.MolFromSmiles(sm)
類似度マップと原子寄与率
このメソッドはある分子の性質に対する原子ごとの寄与weightsがわかっていれば,それをもとに可視化することができます.分子と原子の寄与の他にも以下のようなオプションが指定可能です.特にカラーマップを適切に設定すると見やすい図になると思います.
| オプション | 説明 |
|---|---|
| colorMap | 用いるカラーマップ |
| size | 作成するマップの大きさ.デフォルトは(250,250) |
| colors | 等高線の色 |
| contourLines | 描く等高線の数.デフォルトは10本 |
| alpha | 透過度.デフォルトは0.5 |
なおこの類似度マップを保存しようとする際は,Figureオブジェクトのbbox_inchesオプションを設定しないと,分子の一部しか保存されないことが多いので注意してください.下のコードのように設定すればきちんと保存できるはずです.
fig = SimilarityMaps.GetSimilarityMapFromWeights(mol, weights)
fig.savefig('sample.png', bbox_inches='tight')
原子電荷
分子のGasteiger電荷を計算します.計算したされた値は各Atomオブジェクトごとに‘_GasteigerCharge’というプロパティで格納されます.この値を類似度マップへ渡すことで電荷マップの描画が可能になります.
なおプロパティを取り出す際には文字列になるので,float型へと変換してあげる必要があります.
charges = []
hcharges= []
for atom in taxol_mol.GetAtoms():
charge = float(atom.GetProp('_GasteigerCharge'))
hcharge = float(atom.GetProp('_GasteigerHCharge'))
charges.append(charge)
hcharges.append(hcharge)
fig = SimilarityMaps.GetSimilarityMapFromWeights(taxol_mol,
weights=charges,
colorMap='bwr')
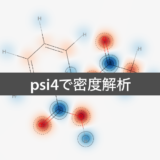
また各原子に結合している表記されていない水素の電荷が‘_GasteigerHCharge’というプロパティに格納されます.水素原子の電荷への寄与を表したものになります.
今回の分子の場合は中性分子ですから,GasteigerChargeとGasteigerHChargeの和はゼロになることが確認できます.また当然ですがChem.AddHsで水素原子を露わにした場合にはGasteigerHChargeはすべて0になります.
print('sum of Gasteiger Charge: {:.2f}'.format(sum(charges)))
print('sum of Gasteiger HCharge: {:.2f}'.format(sum(hcharges)))
sum of Gasteiger Charge: -3.05 sum of Gasteiger HCharge: 3.05
その他の記述子
電荷の他にも,原子ごとの寄与を計算可能な記述子があって,それらはすべて類似度マップによって可視化可能です.ここでは
- MolLogP/MolMR
- LabuteASA
- TPSA
の3つを見ていきましょう.
MolLogP/MolMR
「RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ」という記事で見たように,RDKitで実装されているMolLogPの指標はCrippenらによる原子ごとの寄与から算出したものになります.同様にMolMRについても原子ごとの寄与を求めることができます.
_CalcCrippenContribsでは各原子ごとに(MolLogPへの寄与,MolMRへの寄与)を格納したタプルが得られますので,それぞれを用いることで可視化が可能になります.下のコードで例を見てみましょう.
crippen = rdMolDescriptors._CalcCrippenContribs(taxol_mol)
mol_log = []
mol_mr = []
for x, y in crippen:
mol_log.append(x)
mol_mr.append(y)
fig = SimilarityMaps.GetSimilarityMapFromWeights(taxol_mol,
mol_log,
colorMap='PRGn')
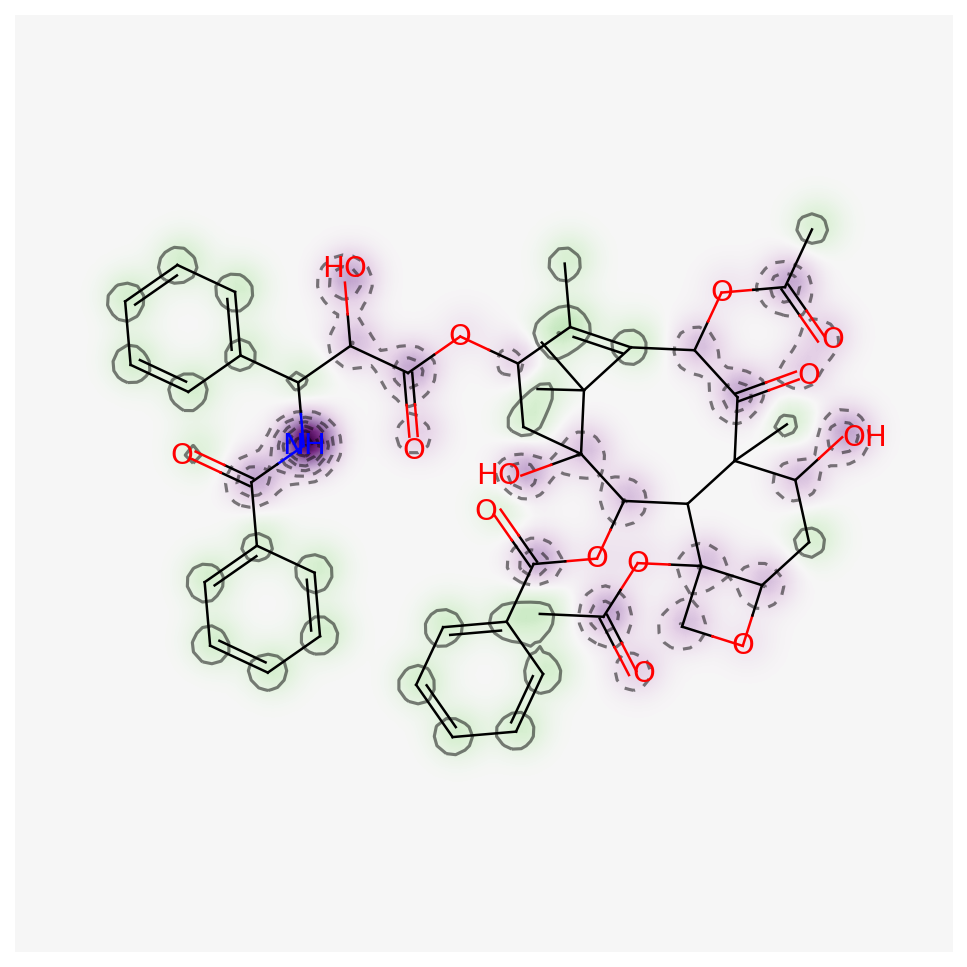
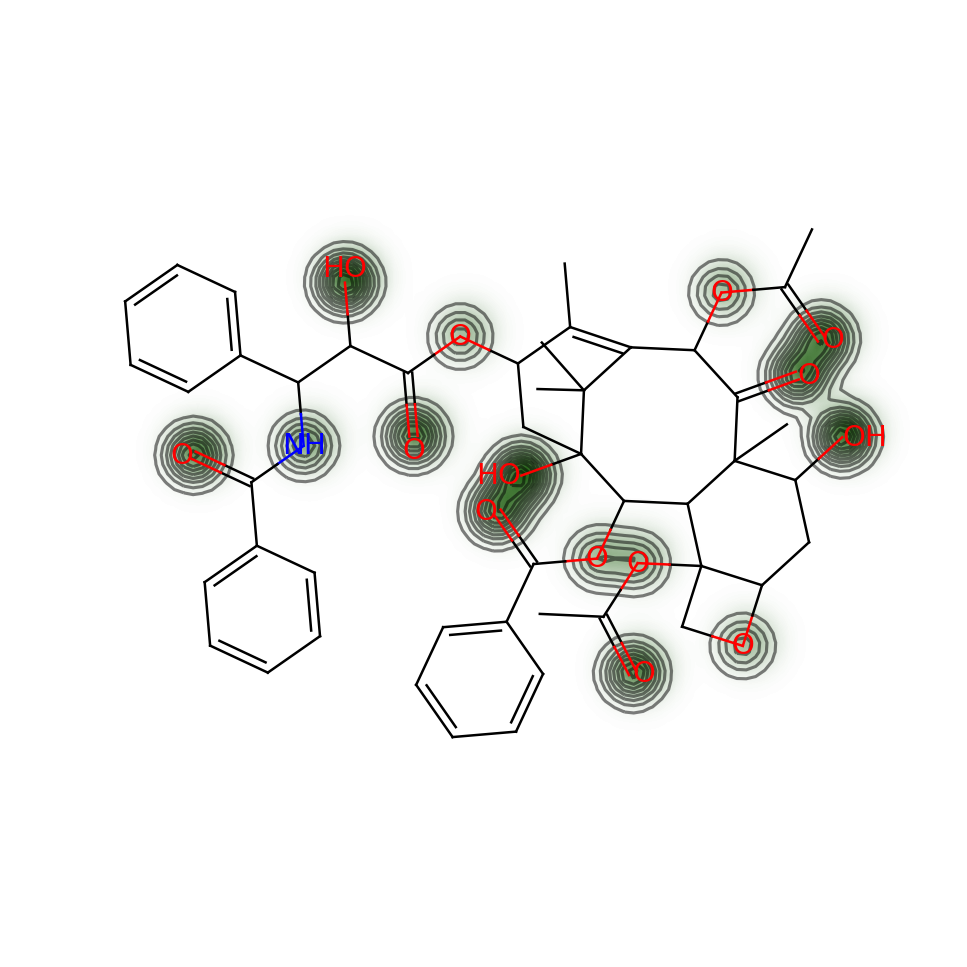
こちらがMolLogPを可視化したものになります.
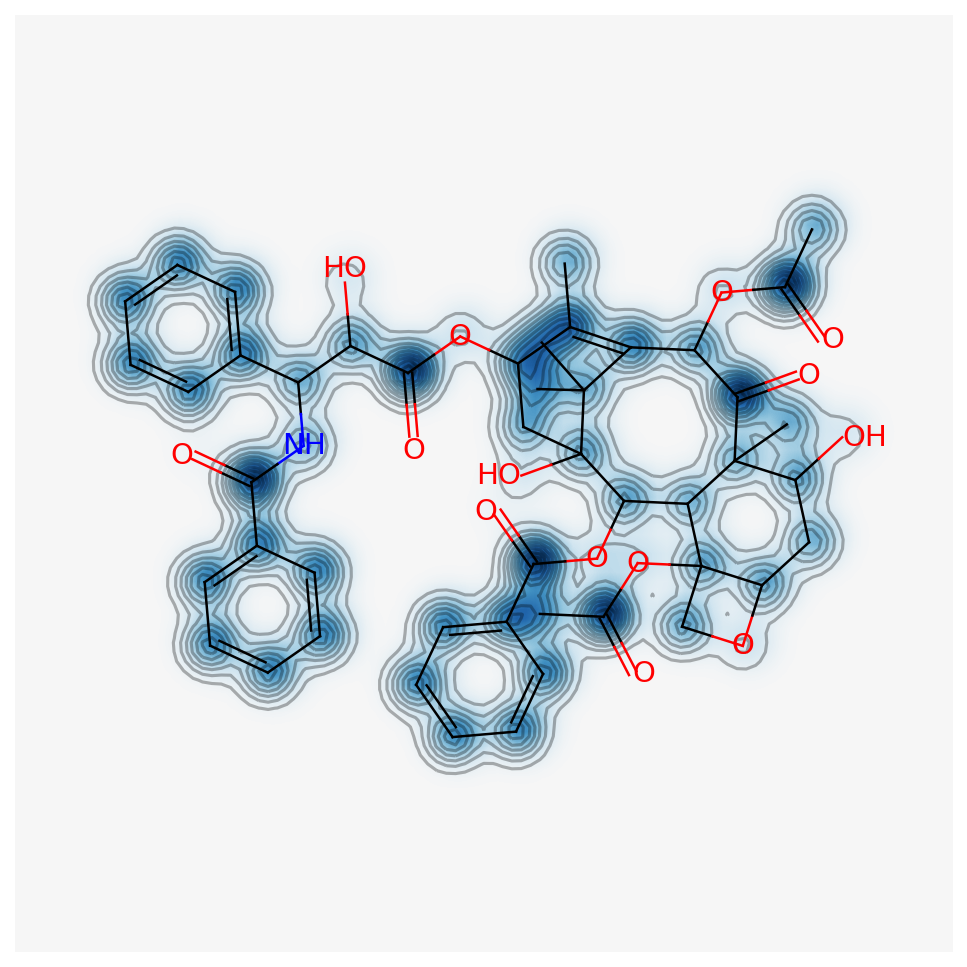
続いてMolMRを可視化したものになります.
LabuteASA
分子の表面積の近似値に対する原子ごとの寄与を計算します._CalcLabuteASAContribsは要素2からなるタプルを返し,1つ目がrdkit.rdBase._vectdというベクトル型のオブジェクトです.この中に原子ごとの寄与が入っていますので,リストに取り出して可視化してみます.タプルの2つの要素はfloatなのですが,分子合計のASA値ではないですし,この値が一体何を示しているのかよくわかっていません.
asa = rdMolDescriptors._CalcLabuteASAContribs(taxol_mol)
fig = SimilarityMaps.GetSimilarityMapFromWeights(taxol_mol,
weights=[x for x in asa[0]],
colorMap='RdBu',
alpha=0.3)
asaの中身は以下の通りです.
(rdkit.rdBase._vectd at 0x11fcc7a50, -1.2087928318874157)

MolMRと同様に分子全体に等高線が広がっていますが,2つの形は微妙に違うことがわかると思います.
TPSA
トポロジカルPSAも原子ごとの寄与を足し合わせたものですから,類似度マップによって可視化が可能になります.
tpsa = rdMolDescriptors._CalcTPSAContribs(taxol_mol)
fig = SimilarityMaps.GetSimilarityMapFromWeights(taxol_mol,
weights=tpsa)
TPSAでは予想通りにヘテロ原子の周りにしか等高線がないことが確認できます.

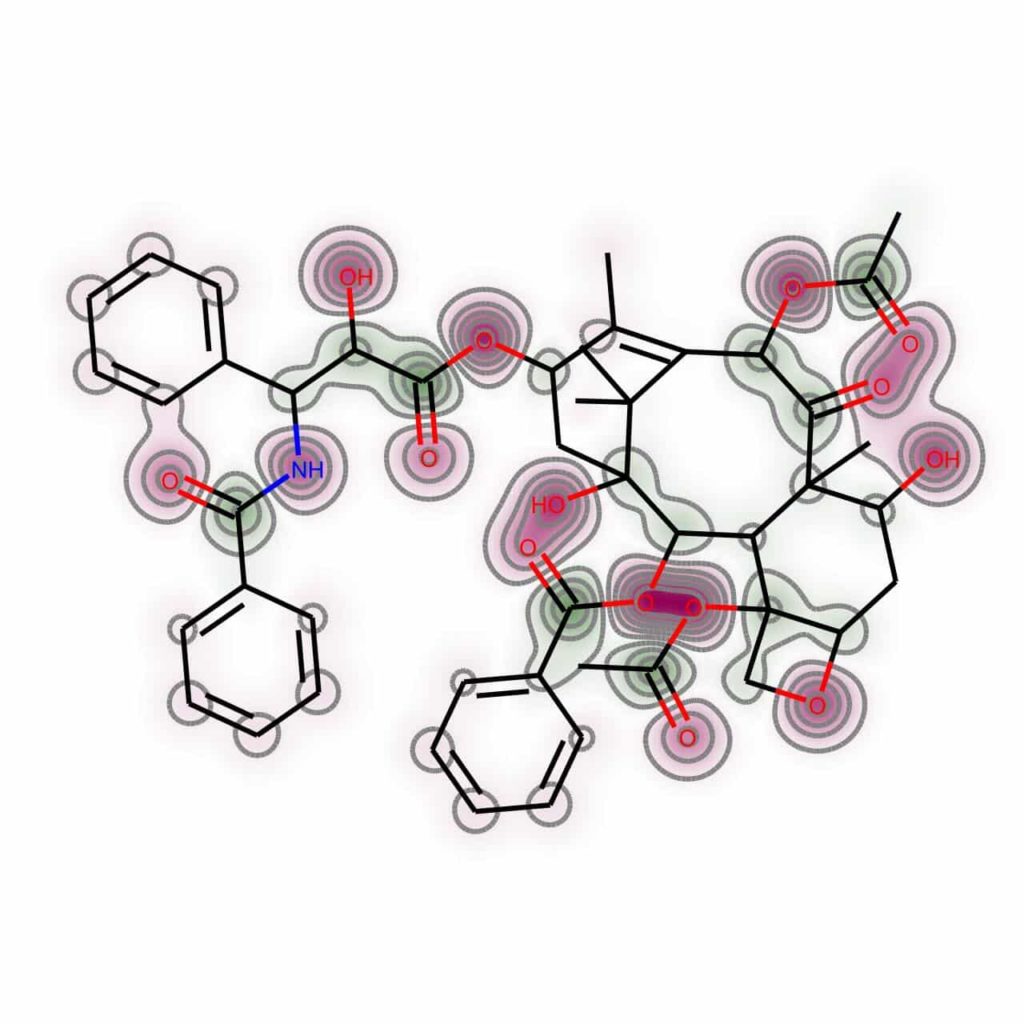
類似度マップをrdMolDraw2Dモジュールで可視化
RDKitでは分子の描画方法を既存のChem.Drawパッケージ(MolToImageなど)から,より速く・美しく描画が可能なrdMolDraw2Dモジュールへの対応が整えられています.今回紹介した類似度マップについても2019.09のアップデートからrdMolDraw2Dモジュールによる描画が可能となっています.
具体的な使い方としては,GetSimilarityMapFromWeightsメソッドのdraw2dオプションに,MolDraw2Dオブジェクトを指定します.以下ではGasteiger電荷について例を示しています.
from rdkit.Chem.Draw import rdMolDraw2D
from IPython.display import SVG
view = rdMolDraw2D.MolDraw2DSVG(400, 400)
fig = SimilarityMaps.GetSimilarityMapFromWeights(taxol_mol,
weights=charges,
colorMap='bwr',
draw2d=view)
view.FinishDrawing()
svg = view.GetDrawingText()
SVG(svg)
終わりに
今回は「RDKitの類似度マップを用いて原子ごとの寄与を可視化する」という話題について,
- 電荷
- MolLogP
- TPSA
などの分子の性質について,原子ごとの寄与を可視化する方法を学びました.フィンガープリントの類似度を可視化する場合にも言えることですが,人間の目は似たものを抽出する能力に非常に長けています.こういった人間の能力を十分に活かすためには,今回のような可視化方法は非常に有用だと言えるでしょう.
RDKitでは2018.09のアップデートから,フィンガープリントのビット配列を化学構造で可視化するコードが追加されました.そちらも併せて学習するとよいと思います.
>>次の記事:「RDKitでフィンガープリントの可視化」
コメント