分子全体の特徴を表現する方法として,「RDKitでフィンガープリントを使った分子類似性の判定」という記事では分子のフィンガープリントを導入し,タニモト係数を用いて類似性の評価などに利用可能であることを述べました.またRDKitで用いることのできるフィンガープリントの種類についても一通りの説明を行いました.
分子のフィンガープリントはケモインフォマティクスにおいてとても有用ですが,具体的な部分構造に基づいたMACCS Keysなどを除くと,フィンガープリントのビット配列から化学的意味を理解するのが困難です.
RDKitでは2018.09のアップデートから,フィンガープリントのビット配列を可視化するコードがrdkit.Chem.Drawに追加されました.今回はこの方法について見ていきたいと思います.
分子と必要なライブラリの準備
まずは必要なライブラリのimportと分子の構築を行いましょう.今回はタキソール(パクリタキセル)を題材としてみます.PubChemでタキソールを検索し,SMILESを取得し,それを使ってRDKitのMolオブジェクトを生成します.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
import pubchempy as pcp
from IPython.display import SVG
print(rdBase.rdkitVersion) ### 2018.09.1
taxol = pcp.get_compounds('taxol', 'name')
taxol = taxol[0]
type(taxol) ### pubchempy.Compound
taxol = Chem.MolFromSmiles(taxol.canonical_smiles)
type(taxol) ### rdkit.Chem.rdchem.Mol
フィンガープリントの準備
Chem.RDKFingerprint(mol, bitInfo)
RDKitのフィンガープリント可視化に関するコードは2018.10月末現在,「Morganフィンガープリント」と「RDKitフィンガープリント」について対応しています.どちらもフィンガープリント作成時に,bitInfoオプションに辞書型の空の変数を指定することで,その後の可視化が可能になります.
### Morganフィンガープリント
bitI_morgan = {}
fp_morgan = AllChem.GetMorganFingerprintAsBitVect(taxol, 2, bitInfo=bitI_morgan)
### RDKitフィンガープリント
bitI_rdkit = {}
fp_rdkit = Chem.RDKFingerprint(taxol, bitInfo=bitI_rdkit)
bitInfoにはビットの立っている位置と,関連する部分構造が辞書形式で格納されています.Morganフィンガープリントでは2048のうち86がON,RDKitフィンガープリントでは2048のうち1444がONのようです.
print(fp_morgan.GetNumBits(),fp_morgan.GetNumOnBits()) ### 2048 86
print(len(bitI_morgan)) ### 86
print(len(fp_rdkit), len(bitI_rdkit.keys())) ### (2048, 1444)
for key in list(bitI_morgan.keys())[:5]:
print(bitI_morgan[key])
((22, 0), (23, 0)) ((30, 1),) ((41, 2),) ((42, 1),) ((20, 1),)
フィンガープリントの可視化
それではいよいよフィンガープリントの可視化について見ていきましょう.
Morganフィンガープリントの可視化
Draw.DrawMorganBit(mol, bit, bitInfo)
Draw.DrawMorganBits(list-of-tuple)
単一のビットの可視化にはDrawMorganBitを,複数のビットの可視化には複数形のDrawMorganBitsを使います.後者ではMolsToGridImageの場合と同様に,molsPerRowやlegendsなどのオプション設定が可能です.
下のコードでは最初の12個の0でないビットについて描画しています.メソッドに渡すタプルは単一ビット描画に必要な「分子」,「描画するビット」,「bitInfoの辞書」の3つをまとめたものになります.
morgan_turples = ((taxol, bit, bitI_morgan) for bit in list(bitI_morgan.keys())[:12]) Draw.DrawMorganBits(morgan_turples, molsPerRow=4, legends=['bit: '+str(x) for x in list(bitI_morgan.keys())[:12]])
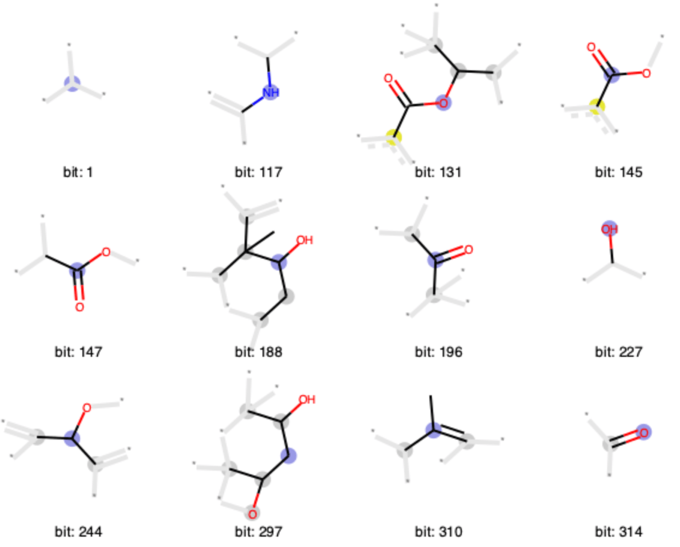
可視化方法としては,
- 原子の位置は分子内の位置と同じように描画
- ビット情報の中心原子は薄い青丸で表現
- 芳香族原子は黄色い丸で表現
- 環状の脂肪族原子は灰色で表現
- 直接にはフィンガープリントに含まれないが,原子の結合タイプ決定に影響する部分を薄い灰色で表現
といったルールに従っているようです.
RDKitフィンガープリントの可視化
Draw.DrawRDKitBit(mol, bit, bitInfo)
Draw.DrawRDKitBits(list-of-tuple)
Morganフィンガープリントの場合とほとんど同じように,単一のビットの可視化にはDrawRDKitBitを,複数のビットの可視化には複数形のDrawRDKitBitsを用います.
下のコードでは最初の0でない12個のビットについて可視化を行っています.RDKitフィンガープリントの方が,
- 0の部分が少ないこと
- 1つのビット情報に含まれる部分構造が大きいこと
が見てとれます.
rdkit_turples = ((taxol, bit, bitI_rdkit) for bit in list(bitI_rdkit.keys())[:12]) Draw.DrawRDKitBits(rdkit_turples, molsPerRow=4, legends=['bit: '+str(x) for x in list(bitI_rdkit.keys())[:12]])
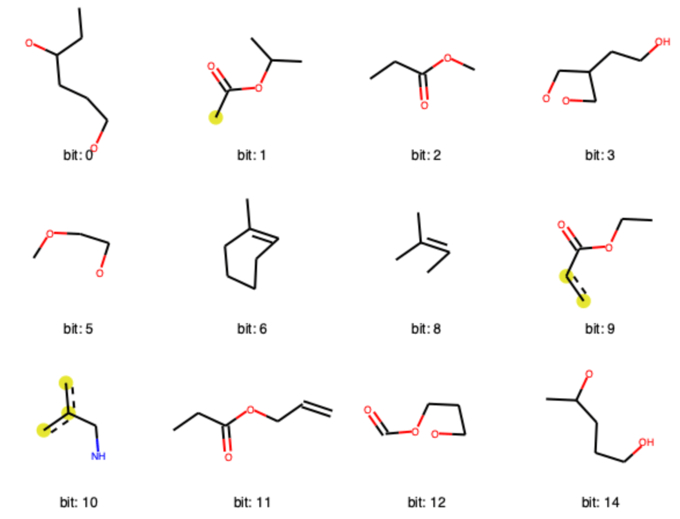
可視化方法としては,
- 原子の位置は分子内の位置と同じように描画
- 描画された結合は全てビット情報の一部
- 芳香族原子は黄色い丸で表現
- 脂肪族原子はデフォルト設定では特に決まった表記なし
といったルールに従っているようです.
複数分子の比較
最後に異なる分子に共通のビット配列を取り出して,その部分構造を見てみましょう.分子としてはZINC15データベースから取得した,酵素をターゲットとしてin vivoで活性のある化合物を抽出したSDFを使ってみます.
suppl = Chem.SDMolSupplier('./enzyme-in-vivo.sdf')
mols = [x for x in suppl if x is not None]
len(mols) ### 653
for mol in mols:
AllChem.Compute2DCoords(mol)
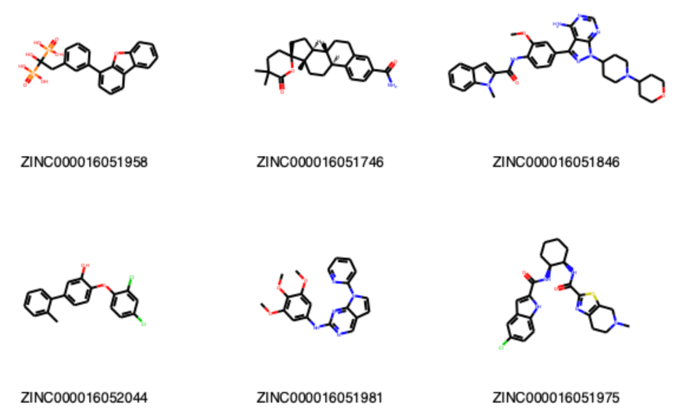
Draw.MolsToGridImage(mols[:6], molsPerRow=3, legends=[m.GetProp('zinc_id') for m in mols[:6]])
最初の6個の分子の構造は以下の通りです.
共通ビットの数え上げ
多数の分子において,どのビットがどの程度の頻度で使われているかを調べてみます.下のコードでは標準ライブラリcollectionsのCounterクラスを使っています.
### フィンガープリントの作成
bit_infos = []
fps = []
for mol in mols:
bit_info = {}
fp = AllChem.GetMorganFingerprintAsBitVect(mol, radius=2, bitInfo=bit_info)
bit_infos.append(bit_info)
fps.append(fp)
### 共通ビットの数え上げ
from collections import Counter
cnt = Counter()
for bit_info in bit_infos:
for bit in list(bit_info.keys()):
cnt[bit] += 1
cnt.most_common(10)
頻出順位トップ10のビット位置と出現回数は以下の通りでした.
[(1380, 611), (1873, 600), (1750, 546), (650, 474), (807, 450), (80, 427), (1057, 415), (1088, 346), (875, 317), (1917, 296)]
共通ビットを有する分子の取得
続いて先ほどの出現回数上位10のビットを全て含む分子を取り出して,構造を比べてみます.
selected_mols = []
n_most_common = 10
bits = [i for i,j in cnt.most_common(n_most_common)]
for (mol, bit_info) in zip(mols, bit_infos):
hit = 0
for bit in bits:
if bit in bit_info.keys():
hit += 1
if hit == len(bits):
selected_mols.append(mol)
len(selected_mols) ### 48
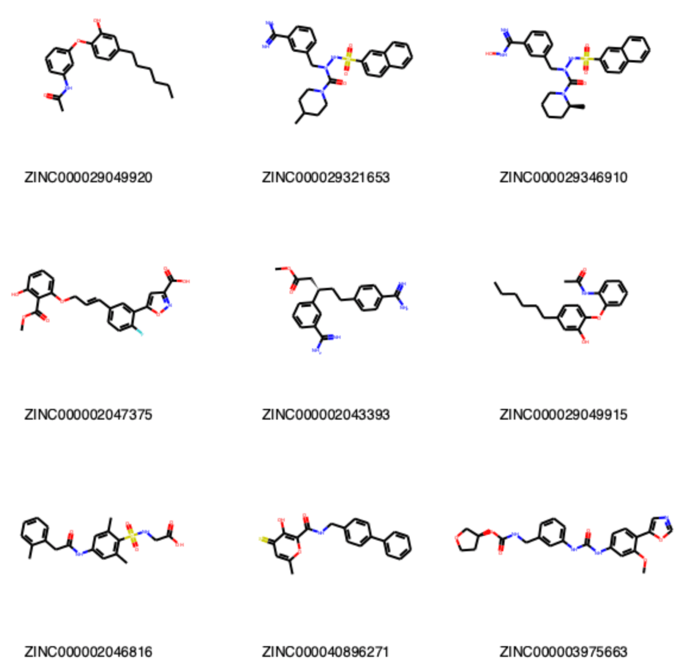
Draw.MolsToGridImage(selected_mols[:9], molsPerRow=3, legends=[x.GetProp('zinc_id') for x in selected_mols[:9]])
48化合物が共通ビットを有しているようです.最初の9化合物の構造は以下の通りでした.似ているような似ていないような気がするでしょうか?
因みに上位10ビットを可視化したものは次のようになりました.芳香族分子+カルボニル部位が基本構造のようです.かなり大雑把な区切りですので,当然選ばれてくる構造もぱっと見では似ていないものになります.
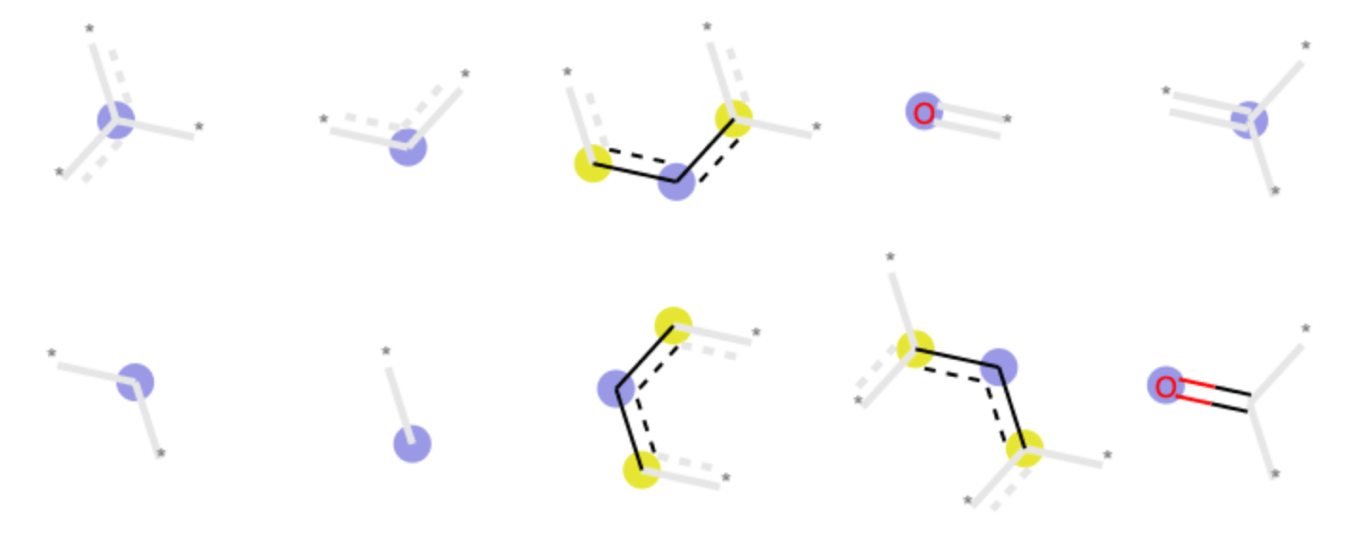
終わりに
今回は2018.09のアップデートから利用可能になったフィンガープリントの可視化コードについて見てきました.これまでブラックボックスに近かったフィンガープリントのビット配列の化学的意味を掴みやすくなったとのではないでしょうか.
RDKitでは分子の性質を可視化する方法として,原子ごとの寄与を類似度マップを用いる方法も実装されていますので,そちらを併せて学習するとよいと思います.
>>次の記事:「RDKitの類似度マップを用いて原子ごとの寄与を可視化する」
コメント