「RDKitでケモインフォマティクスに入門」や「RDKitの分子Molオブジェクトを扱う」
という記事では,
- RDKitのMolオブジェクトはSMILESなどから生成しただけでは各原子の座標情報を保持していないこと(注:下記の囲みも参照)
- 座標情報は2Dまたは3Dの対応するメソッドを用いて生成する必要があること
を説明しました.今回はRDKitで二次元座標を計算する標準手法であるAllChem.Compute2DCoordsメソッドでは構造描画に適した座標が得られにくい場合に,代替手法として有用なCoordGenライブラリーについて紹介してみたいと思います.
復習:RDKitにおける二次元座標
例としてN-メチルプロピオンアミド(N-Methylpropionamide)を見てみます.
from rdkit import Chem
from rdkit.Chem import AllChem
mol = Chem.MolFromSmiles('CCC(=O)NC')
print(Chem.MolToMolBlock(mol, includeStereo=False))
全ての座標が0となっています.
(省略)
0.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 0.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 0.0000 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
(省略)
Compute2DCoordsメソッドを使って,二次元座標を計算します.
AllChem.Compute2DCoords(mol) print(Chem.MolToMolBlock(mol, includeStereo=False))
座標が設定されました.
(省略)
-2.5981 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2990 0.7500 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 -0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0000 -1.5000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2990 0.7500 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
2.5981 -0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
(省略)
それではRDKitのCompute2DCoordsメソッドではきれいな描画ができない例を見ていきます.
CoordGenライブラリー
CoordGenライブラリーはシュレディンガー社による開発になります.UGM-2017の資料(PDF)も参照してみてください.
RDKitの標準アルゴリズムがうまくいかないとき
RDKitの座標構築アルゴリズムではあまりうまく描画ができない例として,
- ビアリールのオルト置換基
- 大環状化合物(マクロサイクル)
- 環内のtrans二重結合
- 金属錯体
などが挙げられます.これらのうちいくつかはCoordGenライブラリーを使うことできれいに描画することが可能になります.
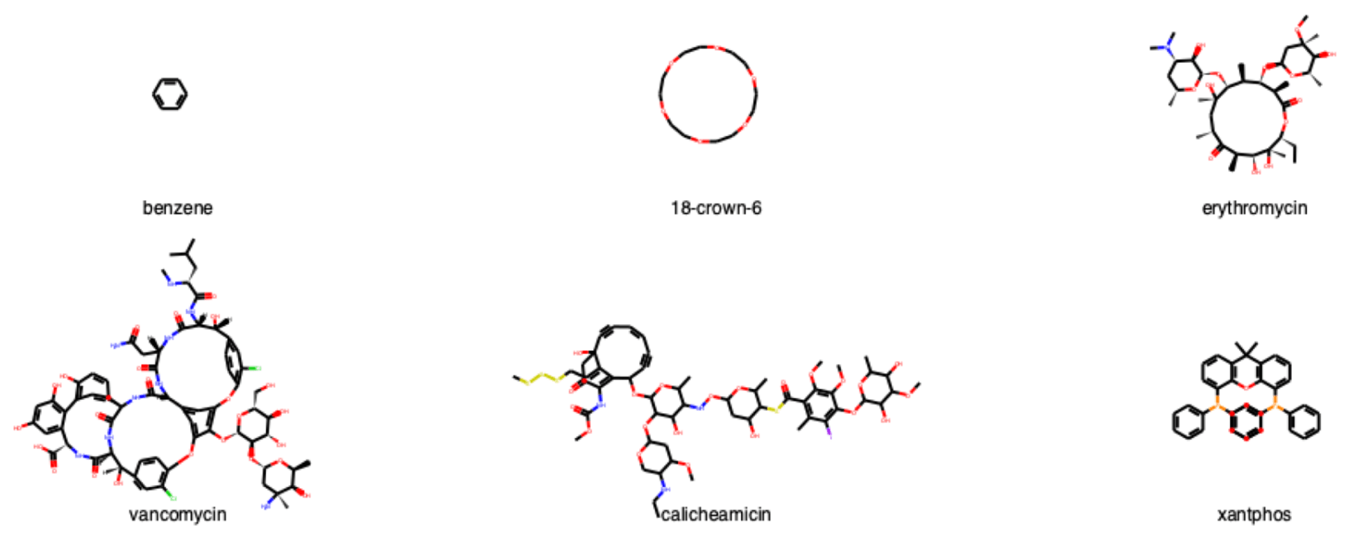
例として
- ベンゼン
- 18-クラウンエーテル
- エリスロマイシン
- バンコマイシン
- カリケアミシン
- キサントフォス
の6化合物について見てみます.
mols = []
mol1 = Chem.MolFromSmiles('C1=CC=CC=C1')
mols.append(mol1) # benzene
mol2 = Chem.MolFromSmiles('C1COCCOCCOCCOCCOCCO1')
mols.append(mol2) # 18-crown-6
mol3 = Chem.MolFromSmiles('CC[C@@H]1[C@@]([C@@H]([C@H](C(=O)[C@@H](C[C@@]([C@@H]([C@H]([C@@H]([C@H](C(=O)O1)C)O[C@H]2C[C@@]([C@H]([C@@H](O2)C)O)(C)OC)C)O[C@H]3[C@@H]([C@H](C[C@H](O3)C)N(C)C)O)(C)O)C)C)O)(C)O')
mols.append(mol3) # erythromycin
mol4 = Chem.MolFromSmiles('C[C@H]1[C@H]([C@@](C[C@@H](O1)O[C@@H]2[C@H]([C@@H]([C@H](O[C@H]2OC3=C4C=C5C=C3OC6=C(C=C(C=C6)[C@H]([C@H](C(=O)N[C@H](C(=O)N[C@H]5C(=O)N[C@@H]7C8=CC(=C(C=C8)O)C9=C(C=C(C=C9[C@H](NC(=O)[C@H]([C@@H](C1=CC(=C(O4)C=C1)Cl)O)NC7=O)C(=O)O)O)O)CC(=O)N)NC(=O)[C@@H](CC(C)C)NC)O)Cl)CO)O)O)(C)N)O')
mols.append(mol4) # vancomycin
mol5 = Chem.MolFromSmiles('CCNC1COC(CC1OC)OC2C(C(C(OC2OC3C#CC=CC#CC4(CC(=O)C(=C3C4=CCSSSC)NC(=O)OC)O)C)NOC5CC(C(C(O5)C)SC(=O)C6=C(C(=C(C(=C6OC)OC)OC7C(C(C(C(O7)C)O)OC)O)I)C)O)O')
mols.append(mol5) # calicheamicin
mol6 = Chem.MolFromSmiles('CC1(C2=C(C(=CC=C2)P(C3=CC=CC=C3)C4=CC=CC=C4)OC5=C1C=CC=C5P(C6=CC=CC=C6)C7=CC=CC=C7)C')
mols.append(mol6) # xantphos
これらの化合物をRDKitの標準方法で描画すると下記のようになります.クラウンエーテルは何員環かすぐにはわかりませんし,エリスロマイシンも同様です.バンコマイシンの場合は大員環に注力しすぎた結果か,中央の芳香環が潰れてしまっています.キサントフォスはリン上のフェニル基が重なってしまっています.
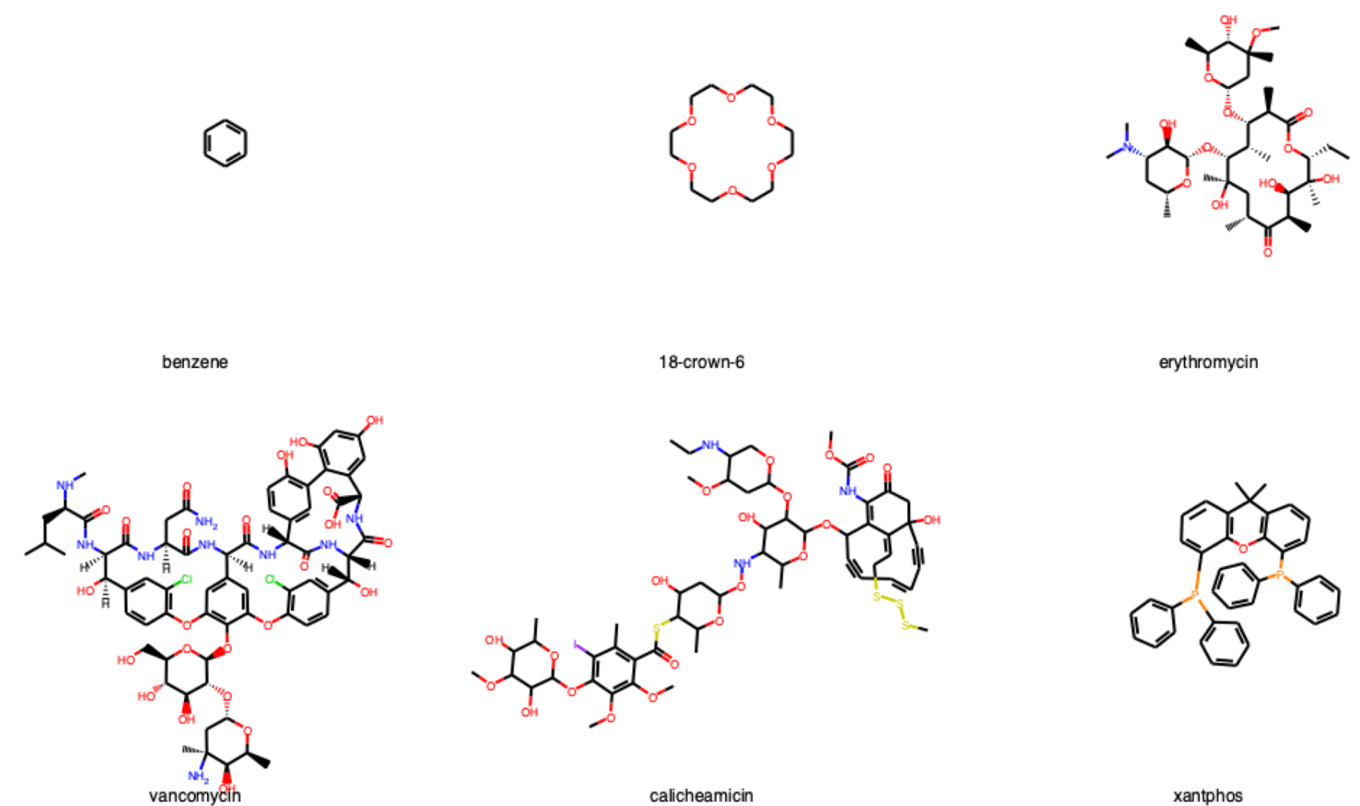
CoordGenの使い方
CoordGenライブラリーはrdkit.Chem下に統合されていますので,importするだけで利用が可能です.二次元座標はAddCoordsメソッドで計算可能です.
from rdkit.Chem import rdCoordGen
for mol in mols:
mol.RemoveAllConformers()
rdCoordGen.AddCoords(mol)
CoordGenを用いることで先ほどの分子は以下のように描画されます.大員環は見やすく描画されていますし,キサントフォスの置換基も重ならないように位置がずれています.
既存コードの書き換えを可能な限り減らしたい場合
既に手元にコードがあって,化合物の描画方法だけを変更したい場合もあるかと思います.そういった際には,rdDepictor.SetPreferCoordGenの設定を変えることで対応可能です.
設定をTrueに変えることで,化合物を描画する際にCoordGenを用いて作成された座標が使われるようになります.
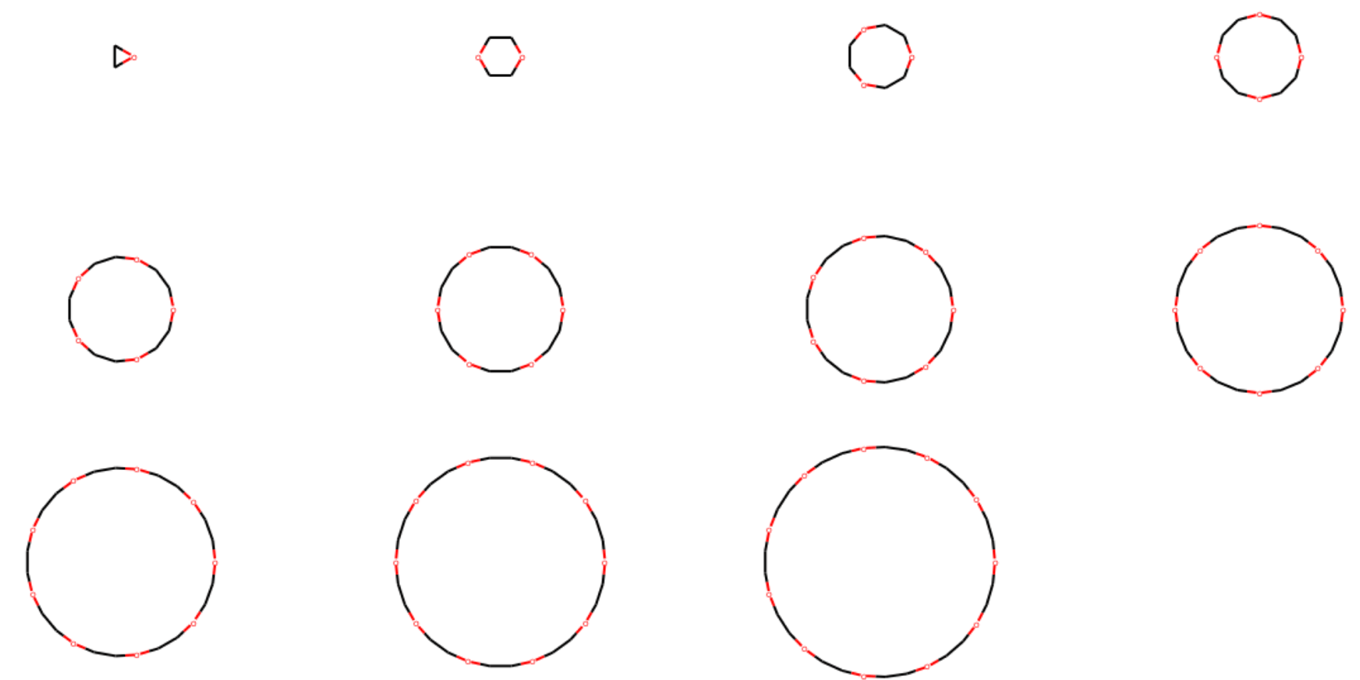
下のコードでは環状のオリゴエチレングリコールをいくつか作成して,2つの方法で描画して比べてみます.
def Make_Crown(num_oxygen=6):
sm = 'O1CC' + (num_oxygen-1)*'OCC'+ '1'
mol = Chem.MolFromSmiles(sm)
return mol
## False
mols = [Make_Crown(x) for x in range(1,12)]
rdDepictor.SetPreferCoordGen(False)
Draw.MolsToGridImage(mols, molsPerRow=4, subImgSize=(300,200))
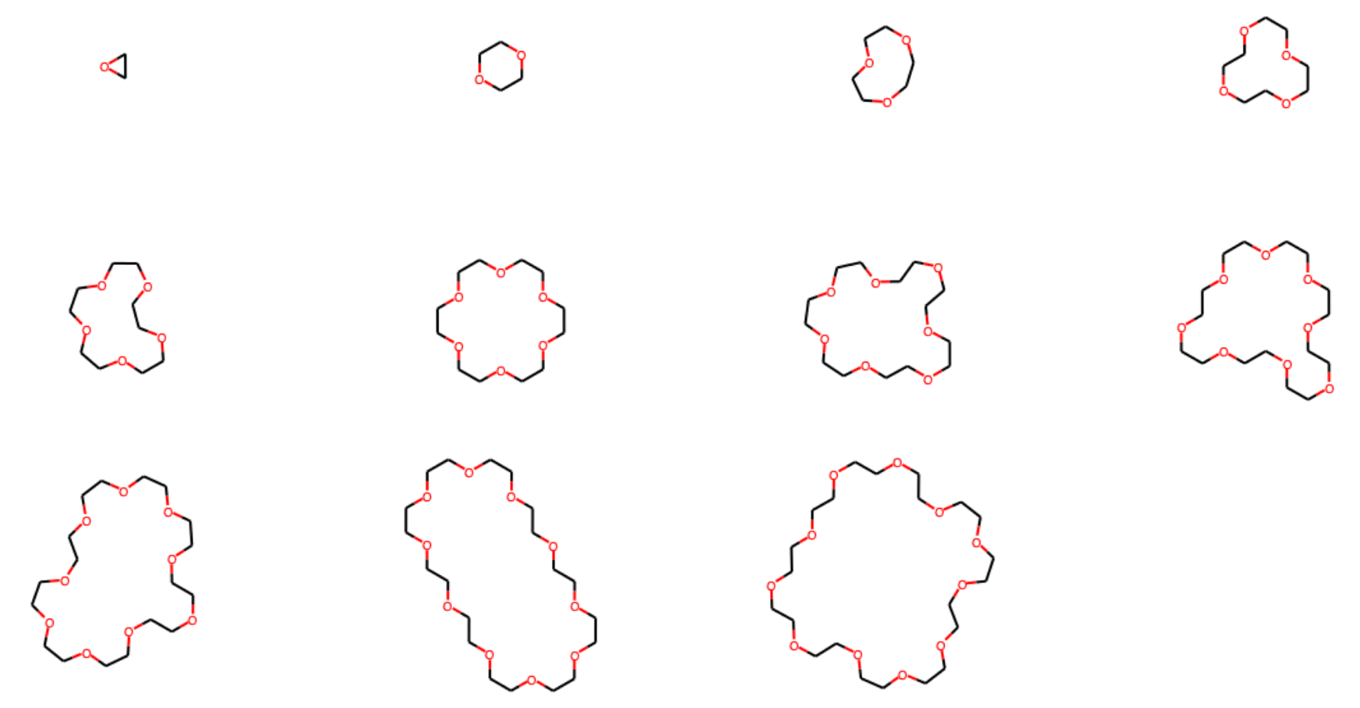
## True
rdDepictor.SetPreferCoordGen(True)
Draw.MolsToGridImage(mols, molsPerRow=4, subImgSize=(300,200))
まずはRDKitの標準設定で描画したものになります.
続いてCoordGenを用いて描画したものになります.
構造式の拡大・縮小
ベンゼンの二次元座標を,
- AllChem.Compute2DCoordsで計算した場合
- rdCoordGen.AddCoordsで計算した場合
にわけて,もう一度見てみます.
(省略)
1.5000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7500 -1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5000 0.0000 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7500 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7500 1.2990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
(省略)
(省略)
-0.3664 -1.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3656 -0.3654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5008 0.1340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3664 -0.3666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3656 -1.3666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4992 -1.8660 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
(省略)
違いがわかるでしょうか?前者の場合は結合距離が「1.5」になっていますが,後者では「1.0」に縮んでいます.即ち,標準設定のままではCoordGenを用いると分子がやや縮小されていると言えます.
これを解消するために座標を引き伸ばす(スケーリング)ことが行われます.スケーリングには
- rdCoordGen.AddCoordsにパラメータを設定して座標を計算する
- AllChem.TransformMolで一旦計算した座標をもとに拡大する
といった2つの方法が考えられます.
AddCoordsの設定を用いる方法
rdCoordGenにはCoordGenParamsと呼ばれる座標を計算するにあたって細かく設定可能なパラメータが存在します.そのうちの設定の1つがcoordgenScalingになります.他のパラメータについては公式ドキュメントを参照してみてください.
このスケールパラメータは使い方がややわかりにくいですが,望みの結合長をxとした場合に「50/x」をパラメータに設定することで望みの結合長を満たすように座標が計算されます.
下記のコードでは結合長「1.5」を設定しています.
m3 = Chem.MolFromSmiles('c1ccccc1')
param = rdCoordGen.CoordGenParams()
param.coordgenScaling = 50/1.5
rdCoordGen.AddCoords(m3, param)
print(Chem.MolToMolBlock(m3))
(省略)
-0.5496 -2.0481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5484 -0.5481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7512 0.2010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0496 -0.5499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0484 -2.0499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7488 -2.7990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
(省略)
先ほどの場合よりも各座標が1.5倍されているのがわかります.
TransformMolを用いる方法
もう1つの方法がChem.AllChemに実装されているTransformMolメソッドを用いる方法になります.このメソッドは4×4の行列を引数とすることで,各軸毎に縮尺を変えることが可能です.
例えば以下の行列ではx軸方向にa倍,y軸方向にb倍,z軸方向にc倍の変換を施します.
$$
\left
(
\begin{array}{cccc}
a & 0 & 0 & 0 \\
0 & b & 0 & 0 \\
0 & 0 & c & 0 \\
0 & 0 & 0 & 0 \\
\end{array}
\right
)
$$
下記のコードでは同様に結合長「1.5」になるようにx,y軸とも1.5倍に拡大しています.
m4 = Chem.MolFromSmiles('c1ccccc1')
scaling = np.zeros([4,4], dtype=np.double)
scaling[0,0] = 1.5
scaling[1,1] = 1.5
rdCoordGen.AddCoords(m4)
AllChem.TransformMol(m4, scaling)
print(Chem.MolToMolBlock(m4))
先ほどの場合と同じ値が得られています.
(省略)
-0.5463 -0.5850 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2061 0.7125 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9537 -0.5880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5069 1.4598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0914 1.4652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0881 2.9649 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3919 0.7176 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
終わりに
今回は「RDKitと二次元構造式:CoordGenで大員環化合物を綺麗に描画」という話題について,
- CoordGenライブラリーの使い方
- 各原子の座標を拡大・縮小する方法
などについて学びました.RDKitの構造式描画については,rdMolDraw2Dモジュールについて学ぶことで理解を深めるのがよいと思います.
>>次の記事:「RDKitでの構造式描画を詳しく解説」

コメント