我々が分子の類似性を評価する際には,「RDKitでフィンガープリントを使った分子類似性の判定」という記事で見たように分子のフィンガープリントを基にしたタニモト係数で評価することが多いです.
この方法自体には問題があるわけではありませんが,何をもって「似ている」とするかは場合によって異なりますので,新しい類似性の判別方法が入り込む余地があります.今回取り上げるFraggleもそういったアルゴリズムになります.
またFraggleと他のフィンガープリントとの違いについては「RDKitブログ: Comparing Fraggle to other fingerprints」でも取り上げられていますので,参照してみてください(英語です).
化合物の類似性評価がどうして必要か
分子構造を眺める際に,「似ている」「似ていない」といった類似性の観点から議論することがよくあります.我々が分子の類似性に興味がある理由として,「類似した化合物は似た性質を持つ」というsimilar property principleというものが挙げられます.ある生理活性を持つ化合物と似た構造の化合物はやはり同じような生理活性を持つというわけです.
上述のように,分子のどんな性質に着目するかによって,「似ている」ことの定義が異なってきます.
Fraggleと既存の類似度評価方法との違い
FraggleはGSK社内で分子類似性を考慮する際に使われている方法のうちの1つで,もともとは2008年にDaylightツールキットを使って実装されていたようです.RDKitでは2013.09リリースから利用可能になっています.
Fraggle開発の動機としては,「分子の中心部での微細な構造変化」に対応するためと述べられています.具体的には以下のようなアザインドール誘導体の位置異性体が例として挙げられています.
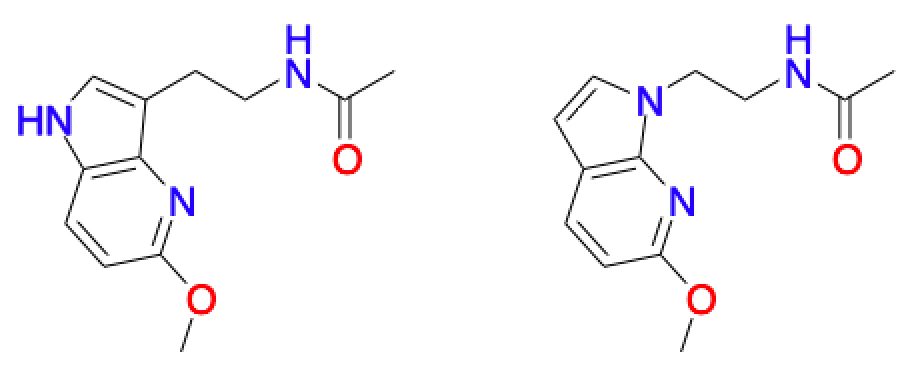
これらの分子をECFP4フィンガープリントで評価した類似度が0.65のようです.これら分子がもっと似ていると思うかどうかは人次第でしょうが,Fraggleではこういった分子の類似度をもっと高く評価するためのアルゴリズムを提供します.
Fraggleとは
Fraggleは上記のアザインドール誘導体のような,分子構造の大局には影響がなさそうな構造変化の影響を受けにくいアルゴリズムを目指しています.
アルゴリズムは分子のフラグメント化を伴う以下の3ステップから成っています.
- 分子のフラグメント化
- Tverskyインデックスを用いて重要なフラグメントの選別
- 選んだフラグメントを用いて対象分子との類似度を判別
それでは具体的にRDKitを用いたFraggleの使い方を見ていきたいと思います.
RDKitでFraggleを使う
「RDKitでフィンガープリントを使った分子類似性の判定」という記事でタニモト係数を学んだ際に,より一般的なTverskyインデックスというものについても触れました.このインデックスは
$$ S_{Tversky} = \frac{c}{\alpha (a-c) + \beta (b-c) + c} $$
という式で表され,タニモト係数は「α=β=1」の場合,Dice係数は「α=β=0.5」の場合に相当することを述べました.なおaは分子Aのビット配列で1が立っている数,bは分子Bのビット配列で1が立っている数,cは分子AとBで共通に1が立っている数になります.
Fraggleのステップ2ではα=0.95, β=0.05の値でフラグメントの選別を行っているようです.選別の閾値は後述の通り0.8ですが,変更可能です.
分子の準備
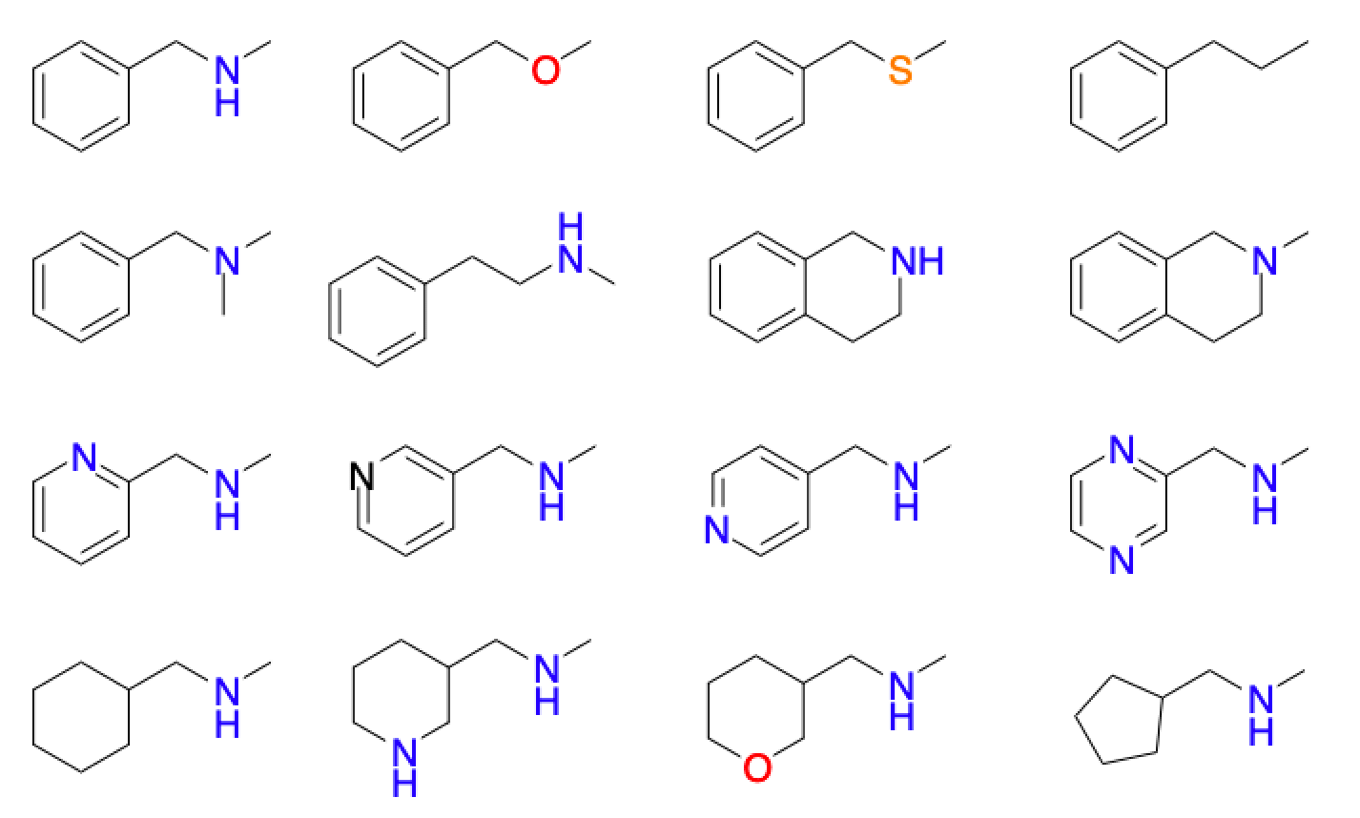
今回は以下のようにN-メチルベンジルアミンを基本としたいくつかの類縁体をモデル分子として扱ってみます.これら分子の類似度がMorganフィンガープリントを使った場合と,Fraggleとでどのように異なってくるかを見ていきます.
from rdkit import rdBase, Chem, DataStructs
from rdkit.Chem import AllChem, Draw
print(rdBase.rdkitVersion) #### 2018.09.1
suppl = Chem.SDMolSupplier('./sample.sdf')
mols = [x for x in suppl if x is not None]
len(mols) ### 16
Morganフィンガープリントを用いた類似度評価
DataStructs.BulkTanimotoSimilarity(refFP, FP_list)
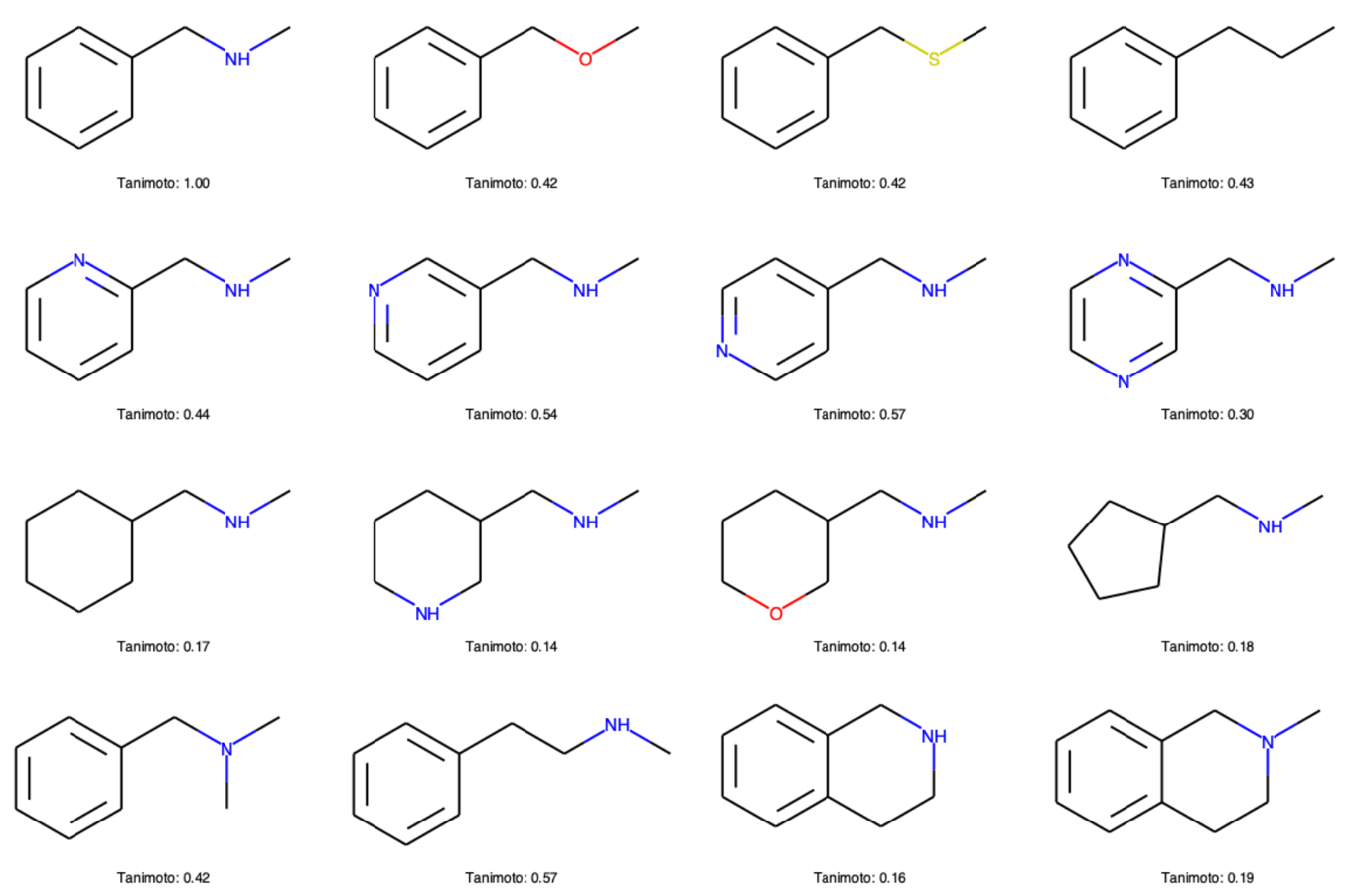
まずはMorganフィンガープリントを用いて類似度を求めてみます.フィンガープリントの生成にはGetMorganFingerprintまたはGetMorganFingerprintAsBitVectを用い,タニモト係数を一気に計算するにはBulkTanimotoSimilarityを使います.
morgan_fp = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 2048) for mol in mols]
tanimoto = DataStructs.BulkTanimotoSimilarity(morgan_fp[0], morgan_fp)
Draw.MolsToGridImage(mols, molsPerRow=4, subImgSize=(300,200), legends=['Tanimoto: {:.2f}'.format(i) for i in tanimoto])
下のように,全体的に類似度が低いと判断されています.
Fraggleを使った類似度評価
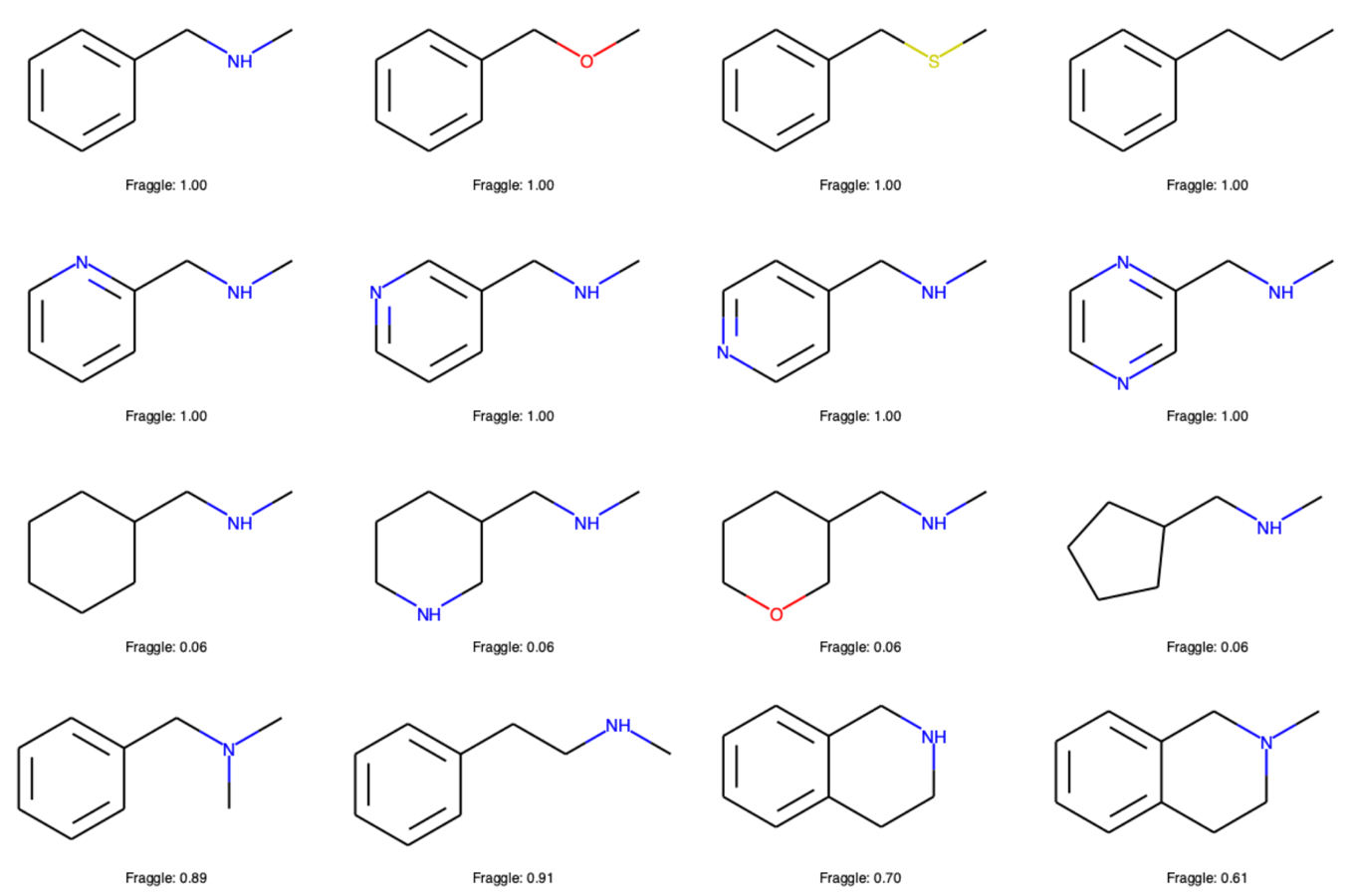
RDKitでFraggleを使うにはChem.FraggleからFraggleSimをimportします.GetFraggleSimilarityメソッドを使うことで2つの分子の類似度を評価できます.戻り値は(類似度,フラグメント構造のSMILES)のタプルになります.先にも述べたようにTverskyインデックスの閾値はデフォルトでは0.8に設定されています.
from rdkit.Chem.Fraggle import FraggleSim
fraggle_similarity = []
for (sim, match) in [FraggleSim.GetFraggleSimilarity(mols[0], mols[i]) for i in range(len(mols))]:
fraggle_similarity.append(sim)
Draw.MolsToGridImage(mols, molsPerRow=4, subImgSize=(300,200), legends=['Fraggle: {:.2f}'.format(i) for i in fraggle_similarity])
芳香環部位の変更はほとんど考慮されていないこと,脂肪鎖部位についても長さや形の変化への感受性が低いことが伺えます.
MorganフィンガープリントとFraggleをプロットしたものが下のグラフです.圧倒的にFraggleの方が類似性が高くでています.
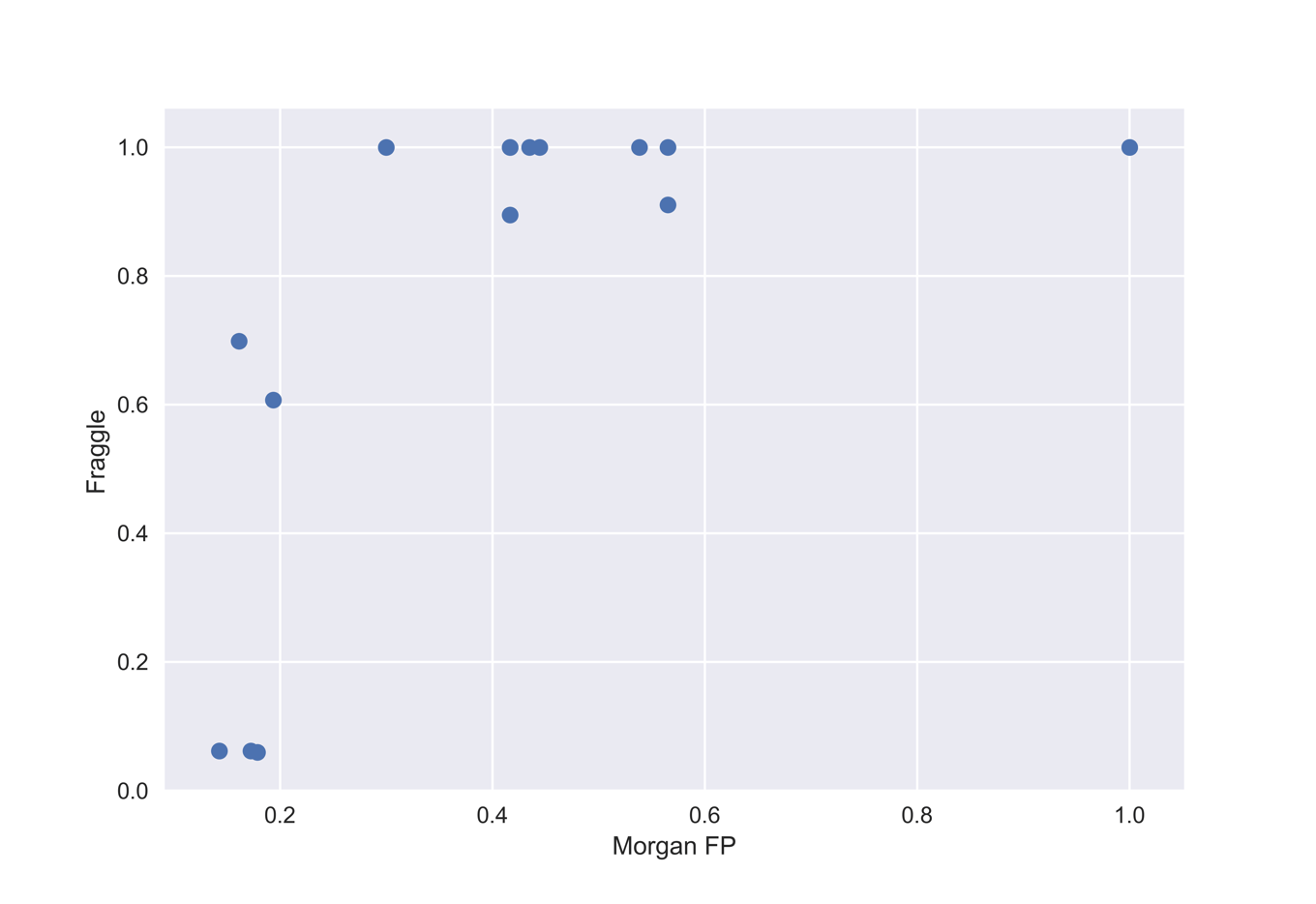
終わりに
今回は「RDKitでFraggleを用いた化合物の類似度評価」という話題について,
- なぜ分子の類似性を評価するのか
- Fraggleとはどういうアルゴリズムなのか
- RDKitではどのようにFraggleを使うのか
というポイントについて説明してきました.特に他のフィンガープリントを用いた類似度の評価方法と比べると,Fraggleは分子中の小さい構造変化に捕らわれにくいことが理解して頂けたかと思います.
色々な方法が存在すると言うことは,裏返せば万能な方法はないということになります.個々の方法の特徴を把握しながら使い分けていけたらよいですね.
コメント