構造式を2次元に描画することは人間が分子の形・性質を理解する第一歩です.これまで「RDKitの分子Molオブジェクトを扱う」という記事ではRDKitにおける分子の扱い方や描画方法を学びました.
また「RDKitを用いた部分構造検索とMCSアルゴリズム」という記事では複数分子の間の共通構造の探索を,実際に構造をハイライトしながら学習してきました.
注意深い方は気がついていたかもしれませんが,RDKitでは複数の描画エンジンが存在していて,用いるメソッドによって描かれる構造式の見た目が違います.今回はRDKitの構造式描画について掘り下げていきながら,2015.03のアップデートより利用可能になったSVG形式での描画方法について説明していきます.やや細かい内容が多いかも知れませんが,裏側で何が行われているかを垣間見ることで理解が深まることも多いはずです.
すぐに使える内容が知りたい方は「rdMolDraw2Dを用いた構造式の描画」から読み始めてください.
・”New Drawing Code”
・”Changes in the 2016.03 Release: Improvements to the molecule drawing code”
・”New drawing options in the 2020.03 release”
必要なモジュールと分子の準備
まずは必要なライブラリのimportと分子を用意しましょう.今回も東京化成から取得したボロン酸ピナコールエステルのSDFを使うことにします.今回取り扱う内容を全て試すには2020.03以降のRDKitが必要です.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
from rdkit.Chem.Draw import rdMolDraw2D
from IPython.display import SVG
print('rdkit version: ', rdBase.rdkitVersion)
# rdkit version: 2020.03.1
suppl = Chem.SDMolSupplier('./sdf_20180823084638.sdf')
mols = [x for x in suppl if x is not None]
len(mols) # 190
構造式の描画方法を復習
Drawパッケージを使った通常の描画方法
まずは「RDKitの分子Molオブジェクトを扱う」という記事でも取り扱ったChem.Drawパッケージを使った方法です.
描画する分子が
- 1つの場合:MolToImage
- 複数の場合:MolsToGridImage
と使い分けることで構造を描いてきました.
Draw.MolToImage(mols[8]) Draw.MolsToGridImage(mols[:10], molsPerRow=5, subImgSize=(200,200))

見た目の違いがわかるでしょうか?ほとんど同じ描画方法のはずなのにMolsToGridImageの方が美しく描画されています.
Jupyter Notebook上での描画
Jupyter上では明示的にDrawモジュールを使わずに構造を表示することができます.さらに構造式マッチも,highlightAtomsを指定しなくても自動で表示されます.


どちらの構造もMolsToGridImageと同じようにきれいに描画されています.
RDKitでの芳香族の扱い
RDKitがSmilesやMOLブロックなどから分子を構築する際には,sanitizationという一連の初期化作業が行われています.その作業の一つに芳香族の処理があります.
次の例でベンゼンを例に見てみます.
c6h6 = Chem.MolFromSmiles('c1ccccc1')
for b in c6h6.GetBonds():
print('{}: {}\t{}'.format(b.GetIdx(),b.GetBondType(),b.GetIsAromatic()))
0: AROMATIC True 1: AROMATIC True 2: AROMATIC True 3: AROMATIC True 4: AROMATIC True 5: AROMATIC True
ベンゼンの全ての結合のタイプがAROMATIC結合として設定されています.また芳香族か否かのフラグでも芳香族という判定です.この2つの指標は一見すると等価に思えますが,実は異なるということをすぐ下で確認します.
芳香族のケクレ化
Chem.SanitizeMol(mol)
結合を芳香族として扱うのを止めるのがKekulizeです.先ほどのベンゼンをケクレ化して,結合のタイプと芳香族化否かの判定を行ってみます.
Chem.Kekulize(c6h6)
for b in c6h6.GetBonds():
print('{}: {}\t{}'.format(b.GetIdx(),b.GetBondType(),b.GetIsAromatic()))
0: DOUBLE True 1: SINGLE True 2: DOUBLE True 3: SINGLE True 4: DOUBLE True 5: SINGLE True
こんどは結合タイプは単結合と二重結合になりましたが,いずれの結合も芳香族と判定されています.このことからRDKit内ではこの2つの指標は異なるものとして扱われていることがわかりました.
ケクレ化した分子を戻したい場合にはSanitizeMolを使うことで初期化可能です.KekulizeもSanitizeMolも,どちらのメソッドも作業をした分子を置き換えることに注意してください.
ケクレ化で結果が変わる例:最大共通構造(MCS)
それではケクレ化することで結合タイプを変えた場合に,影響が出るのはどういった場合でしょうか?例えば「RDKitを用いた部分構造検索とMCSアルゴリズム」でもみたようにMCSが考えられます.下のコードでデフォルトの状態のベンゼンと,ケクレ化する前とでMCSがどのように変わるかを見てみます.
from rdkit.Chem.rdFMCS import FindMCS
c6h6 = Chem.MolFromSmiles('c1ccccc1')
x = FindMCS([c6h6, mols[8]])
Chem.Kekulize(c6h6)
y = FindMCS([c6h6, mols[8]])
Draw.MolsToGridImage([Chem.MolFromSmarts(x.smartsString), Chem.MolFromSmarts(y.smartsString)],
subImgSize=(250,250),
legends=['default', 'Kekulized'])
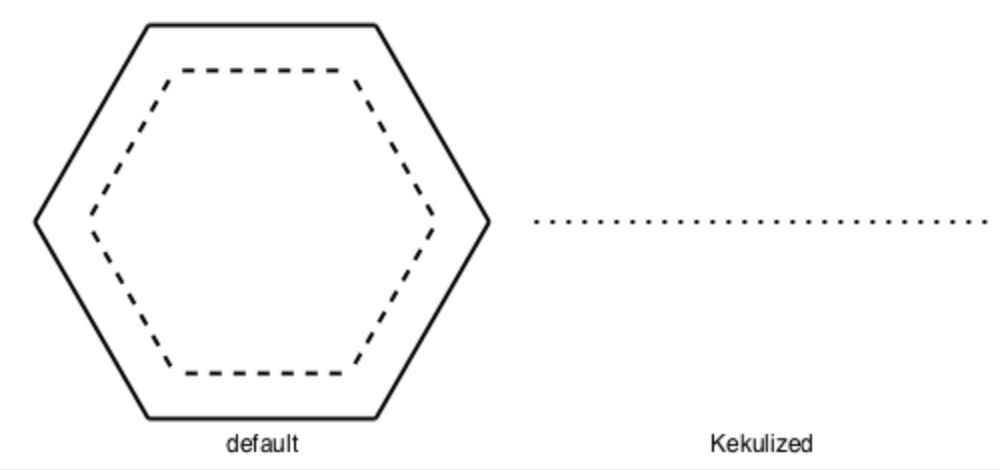
左側はデフォルト設定のベンゼンです.芳香環がそのままマッチしています.MCSのbondCompareオプションは単結合と芳香族結合はマッチしますが二重結合は芳香族結合とはマッチしません.そのためケクレ化した場合には単結合(芳香族結合)1つのみがマッチしています.
Drawモジュールでの描画を理解する
ここでDrawパッケージのオプションを見てみましょう.
| オプション | 説明 |
|---|---|
| size | 描画領域のサイズをタプルでピクセル指定.デフォルトは(300, 300) |
| kekulize | 分子の芳香族部位をケクレ構造にするかどうか.デフォルトはTrue |
| wedgeBonds | 立体の表示にくさび型表記を使うかどうか.デフォルトはTrue |
| highlightAtoms | ハイライトする原子のリスト |
| highlightBonds | ハイライトする結合のリスト |
| highlightColor | ハイライトする部位の色をRGBで指定.デフォルトは[1,0,0]の赤色 |
| legend | 構造式につける凡例の設定 |
kekulizeというオプションを見るとわかりますが,実はDrawモジュールにMOLオブジェクトを渡した際にはデフォルト設定ではケクレ化が行われた上で,描画されています.
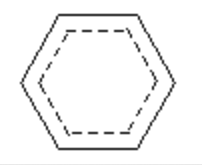
なおこの他にも,分子中の原子がxy座標を持っていない場合は座標を取得してから描画しています.例えば先ほどのベンゼンは座標が設定されていませんが,問題なく描画することが可能です.
Draw.MolToImage(c6h6, kekulize=False)
きちんと芳香族結合が反映された構造式が描画されました.
rdMolDraw2Dを用いた構造式の描画
長々と説明してきましたが,ここからはRDKit 2015.03のアップデートより追加されたChem.rdMolDraw2Dモジュールについて解説していきます.一部機能は2020.03以降のバージョンでないと利用できないことに注意してください.rdMolDraw2DはChem.Drawよりもきれいで,速い描画が可能です.
rdMolDrawモジュールでは
- PNG(Cario)
- SVG
- Qt
などの形式へと書き出すことができます.ここではsvg形式に特化して説明していきます.
いずれの場合も出力先のオブジェクトが異なるだけで,基本的な使い方は同じはずです.
なお先ほど見たようにほとんど同じDrawモジュールによる描画にも関わらずMolsToGridImageの出力の方がきれいに描画されていましたが,これはこちらのメソッドは描画エンジンとして新しいrdMolDraw2Dモジュールを使っているためです.
svg形式とはどのような画像ファイルフォーマットか?
svgファイル(Scalable Vector Graphics)はXMLをベースとした二次元ベクトル型の画像ファイルフォーマットです.
jpegやpngなどの他の画像形式がビットマップデータなのに対し,svgはX二次元ベクトルデータであるため拡大・縮小しても画質が損なわれることがありません.またsvgはXMLに準拠していることから,テキストエディタで中身を確認したり,編集することが可能です.
一般に写真などのデータはビットマップデータ,ロゴ・図形などはベクトルデータでの扱いが適していると言われます.構造式も線を中心として形成される図形のようなものですから,ベクトルデータで扱うことが望ましいわけです.(このブログに載せている画像は設定の都合上,svgをjpgに変換したものを用いています.)
rdMolDraw2Dを用いる際の描画手順
rdMolDraw2Dモジュールを用いた構造式描画は,Drawモジュールのように1行で完結しません.下に示すようにまずはMolDraw2DSVGオブジェクトというコンテナを作成し,そこにMolオブジェクトを追加していくイメージになります.
上記のように必ずしもSVGを用いる必要はありませんが,ここではSVGでの出力のみ扱います.
- コンテナとなるオブジェクトの作成
- コンテナの細かい描画設定
- コンテナに描画したい分子を追加
- コンテナをファイナライズ
- コンテナに書き込んだデータを取り出す
- データを描画,またはファイルとして保存
それでは使い方を見ていきましょう.まずは各ステップの説明をしてから,最後に具体的な描画例を見ていきます.操作の流れは「RDKitによる3次元構造の生成」という記事で扱ったpy3Dmolを用いた立体構造の描画と似ています.
コンテナとなるMolDraw2DSVGオブジェクトの作成
MolDraw2DSVGでは,まず横int1 x 縦int2の描画領域が作成されます.続いてその領域に対して横int3 x 縦int4のサイズのグリッドを作成します.複数分子を表示する場合には各グリッドを順番に使って描画していくことになります.
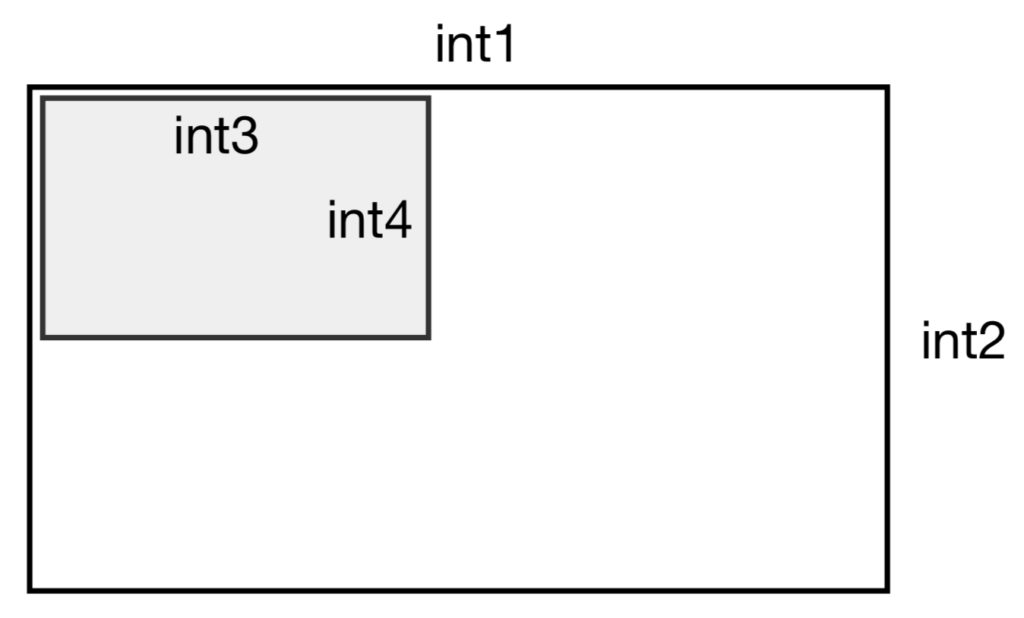
int3/4を指定しない場合は全領域が1つのグリッドとして扱われます.1分子を描く場合は特に設定は必要ありませんが,複数分子を描画する場合にはグリッド設定を適切にしないと同じグリッドに複数分子が重なってしまい思ったような図になりません.
コンテナに分子を追加
MolDraw2DSVG.DrawMolecules(mols)
rdMolDraw2D.PrepareMolForDrawing(mol)
作成した描画領域に対して分子を追加していきます.1分子の場合にはDrawMoleculeを,複数分子の場合にはDrawMoleculesを使います.
指定できるオプションは下の表のようになっていて,大体がDrawモジュールの場合と同じですが,ハイライト設定が原子とその色だけでなく結合なども設定可能になっています.また複数分子を表示する場合,Draw.MolsToGridImageではhighlightAtomListsなどだったのが,DrawMoleculesでは単数の場合と同じようにhighlightAtomsを使います.
| オプション | 説明 |
|---|---|
| highlightAtoms | ハイライトする原子のリスト |
| highlightAtomColors | ハイライトする原子の色 |
| highlightAtomRadii | ハイライトする原子の半径を設定 |
| highlightBonds | ハイライトする結合のリスト |
| highlightBondColors | ハイライトする結合の色 |
| legend(s) | 凡例 |
このまま構造式を表示してみると,芳香族がケクレ化されていません.

さきほどDraw.MolToImageでは実際に構造式を表示する前にメソッド内で分子に対していくつかの内部処理を施していることを見ました.rdMolDraw2Dモジュールではこの内部処理に相当するメソッドPrepareMolForDrawingが実装されています.その処理は主に3つです.なおこのメソッドは処理を施した新しいMolオブジェクトを返し,もとのオブジェクトは変更しません.
- 芳香族のケクレ化
- 不斉炭素に水素を付加し,くさび形の結合で表示
- 原子が座標を持っていない場合に,2次元座標を計算
PrepareMolForDrawingメソッドを用いてから描画を行うとケクレ化された構造式が表示されます.
コンテナの細かい描画設定
コンテナの設定はコンテナ自体のメソッドから設定する項目と,drawOptionsオブジェクトを作成してから設定する項目に分けられます.注意点としてはこれらの設定はコンテナに分子を追加する前に行う必要があります.
コンテナのメソッド
最も使用頻度が高いのはフォントサイズの設定だと思います.現在のフォントサイズはFontSizeで確認できます.コンテナの大きさによってデフォルトの値が変わってきますので,絶対値を指定するよりも現在のサイズの1.3倍のような指定の仕方をしたほうがよいです.
drawOptionsオブジェクト
コンテナの描画設定はコンテナのdrawOptionsオブジェクトを作成して行います.色々な設定項目がありますが,最後に具体例と共に見ていきますので,ここでは概略を掴むだけで構いません.
| オプション | 説明 |
|---|---|
| atomLabelDeuteriumTritium | 重水素をD,トリチウムをTで表記 |
| atomLabels | 原子Idで指定した原子のラベルを設定 |
| additionalAtomLabelPadding | 元素記号と結合との間にどの程度マージンを取るかを,フォントサイズとの割合で指定. |
| circleAtoms | ハイライトするアトムを円で表示するかどうか.デフォルトはTrue |
| continuousHighlight | ハイライト領域を塗りつぶすかどうか.デフォルトはTrue.circleAtomsと併せて設定するときれいに描画できます. |
| fillHighlights | ハイライトするアトムの円を塗りつぶすかどうか.デフォルトはTrue |
| padding | 描画領域のうちマージンをどの程度とるかを0から1の割合で設定.凡例を表示したい場合にはマージンをとらないと構造式と重なってしまいます |
| legendFontSize | 凡例のフォントサイズをpx単位で指定 |
| multipleBondOffset | 多重結合の2つ目以降の線をどの程度離して描画するかをオングストローム単位で指定 |
| addAtomIndices | 原子Idを構造式中に表示.デフォルトではFalse |
| addBondIndices | 結合Idを構造式中に表示.デフォルトではFalse |
| annotationFontScale | 原子や結合に付記する注釈のフォントサイズを,割合で指定.デフォルトは0.75 |
| addStereoAnnotation | 絶対配置R/Sや幾何異性E/Zを表示.デフォルトではFalse |
続いてメソッドです.
| メソッド | 説明 |
|---|---|
| useBWAtomPalette | 構造式を白黒で描画します |
| useDefaultAtomPalette | 構造式描画に用いる色をデフォルトに戻します |
| updateAtomPalette | 構造式描画に用いる色を,原子番号とRGBのタプルを組とする辞書形式で指定します |
| setBackgroundColour | キャンバス色の変更をします |
| setHighlightColour | ハイライト領域のデフォルト色を変更します |
コンテナのファイナライズと書き込んだデータの取り出し
MolDraw2DSVG.GetDrawingText()
コンテナへの書き込みを終えた場合には,FinishDrawingを用いてコンテナのファイナライズを行います.この作業を行うことで書き込んだデータの取り出しが可能になります.開いていたファイルを閉じていると考えてください.
続いてGetDrawingTextで書き込んだデータをstr形式で取り出します.
Jupyterノートブック上での表示
Jupyterノートブック上で表示するにはIPythonライブラリのSVGモジュールを使って表示します.その際に,取り出したstrオブジェクトのままではなぜかうまくい表示されません.
strオブジェクトのメソッドを使って,svg.replace(‘svg:’, ”)とすることでテキストをやや変更することでJupyterノートブックでの構造式表示が可能になります.
なおJupyter上でMolオブジェクトをタイプするだけで構造が表示する場合には,大体はこれら一連の作業と同じことがバックグラウンドで行われているようです.
svg形式でのファイル保存
svgの中身はテキスト形式になっていますので,画像の保存は通常のファイルオブジェクトを用いた書き込みで可能です.
with open(path-to-your.svg, 'w') as f:
f.write(svg)
具体例
rdMolDraw2Dモジュールを使ったsvg形式での出力について一連の作業を説明してきました.いくつか具体的例を通してコードでどのように表現するかを見ていきたいと思います.
ハイライト領域の調整
この例ではデフォルトの原子を円形でハイライトする設定をオフにしてみます.
具体的な作業としては以下の3段階を経ています.
- ベンゼンにマッチする原子のインデックスを取得
- drawOptionsのcircleAtomsとcontinuousHighlightにFalseを設定
- highlightAtomsにリストを渡して構造式を表示
# 1. ベンゼンにマッチする原子のインデックスを取得
highlights = m.GetSubstructMatch(Chem.MolFromSmiles('c1ccccc1'))
# コンテナと分子の準備
view = rdMolDraw2D.MolDraw2DSVG(300, 300)
tm = rdMolDraw2D.PrepareMolForDrawing(m)
# 2. 各種オプションの設定
option = view.drawOptions()
option.circleAtoms=False
option.continuousHighlight=False
# 3. 分子をコンテナに渡す
view.DrawMolecule(tm, highlightAtoms=highlights)
view.FinishDrawing()
# コンテナの描画
svg = view.GetDrawingText()
SVG(svg))
原子ラベルとハイライトの色を変更
この例では
- 原子ラベル:atomLabels
- ハイライトする色:highlightAtomColors
を変更してみます.
加えて,
- ハイライトする原子の半径:highlightAtomRadii
- フォントサイズ:SetFontSize
をやや小さくしています.
さらに二重結合のオフセット幅multipleBondOffsetを狭め,凡例表示用に描画領域のマージンpaddingを設定しています.
描画される構造は以下通りです.

# ハイライトする原子と色,円の半径を設定
atoms = [6,8]
colors = {6: (1,1,0), 8: (1,0,1)}
radii = {6: 0.25, 8: 0.25}
# コンテナの準備
view = rdMolDraw2D.MolDraw2DSVG(300,350)
tm = rdMolDraw2D.PrepareMolForDrawing(m)
# 描画オプションの設定
view.SetFontSize(0.9 * view.FontSize())
option = view.drawOptions()
option.atomLabels[6] = 'N1'
option.atomLabels[8] = 'N2'
option.multipleBondOffset=0.07
option.padding=0.11
option.legendFontSize=20
# コンテナに分子を登録
view.DrawMolecule(tm,
highlightAtoms=atoms,
highlightAtomColors=colors,
highlightAtomRadii=radii,
legend='atom labels')
view.FinishDrawing()
# 描画
svg = view.GetDrawingText()
SVG(svg)
matplotlibを用いてRGBのカラーコードを生成
ハイライトする色はRGBカラーコードのタプルで設定する必要があります.多くの人にとって色の名前とカラーコードは対応していませんので,できることなら’gray’などと色の名前で設定したいところです.
これを実現するメソッドがmatplotlibのColorConverterに実装されています.
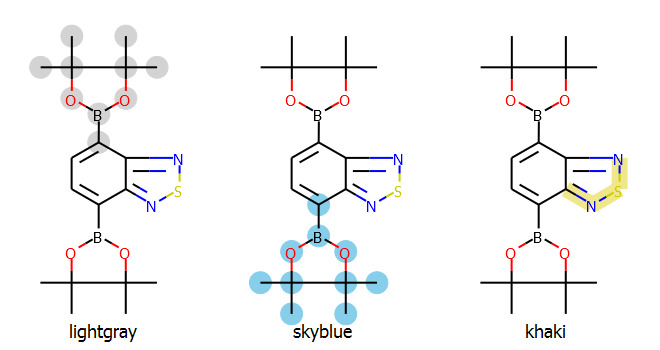
この例では
- 横660x縦350の描画領域を作成し,グリッド1つの大きさを横220x縦350に設定
- 作成した領域に3つの分子を描画
していきます.その過程で,
- ピナコールエステル部位について部分構造マッチを行うことで原子Idを取得
- 1つのマッチ部位を最初の分子で,2つめのマッチ部位を次の分子で違う色でハイライト
- 最後の分子はチアジアゾール環について原子ではなく結合部位だけをハイライト
しています.
from matplotlib.colors import ColorConverter
# 部分構造マッチを取得し,ハイライトする原子の色情報を格納する辞書を作成
matches = m.GetSubstructMatches(Chem.MolFromSmiles('C[B]1OC(C(O1)(C)C)(C)C'))
color_1 = {}
color_2 = {}
color_3 = {}
radius = {}
# ハイライトする部位の色と円の半径を設定
for i,j in zip(matches[0], matches[1]):
color_1[i] = ColorConverter().to_rgb('lightgray')
color_2[j] = ColorConverter().to_rgb('skyblue')
radius[i] = 0.25
radius[j] = 0.25
for i in [6,7,8]:
color_3[i] = ColorConverter().to_rgb('khaki')
# コンテナと分子の準備
tm = rdMolDraw2D.PrepareMolForDrawing(m)
view = rdMolDraw2D.MolDraw2DSVG(660,350, 220, 350)
# オプションの設定
option = view.drawOptions()
option.padding=0.13
option.legendFontSize=18
# コンテナに分子を3つ登録
view.DrawMolecules([tm,tm,tm],
highlightAtoms=[matches[0],matches[1],{}],
highlightAtomColors=[color_1, color_2, {}],
highlightAtomRadii=[radius, radius, {}],
highlightBonds=[[],[], [6,7,8]],
highlightBondColors=[{},{}, color_3],
legends=['lightgray', 'skyblue', 'khaki'])
# 描画
view.FinishDrawing()
svg = view.GetDrawingText()
SVG(svg)
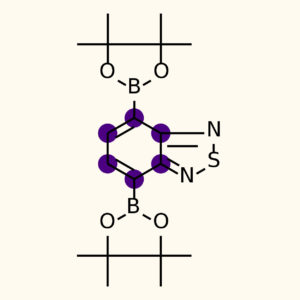
背景とカラーパレットの変更
続いてオプションのメソッドを用いて
- 構造式を白黒で描画(パレットの変更)
- 背景を変更(フローラルホワイト)
- ハイライトする部位の色を変更(インディゴ)
という3つの変更を施してみます.
# 色のRGB表記
floral_white = ColorConverter().to_rgb('floralwhite')
indigo = ColorConverter().to_rgb('indigo')
# ハイライトする部位の半径情報
radii = {i: 0.2 for i in range(6)}
# コンテナの設定
view = rdMolDraw2D.MolDraw2DSVG(300, 300)
# オプションの設定
options = view.drawOptions()
options.useBWAtomPalette() # 白黒構造で描画
options.setBackgroundColour(floral_white) # 背景の色を変更
options.setHighlightColour(indigo) # ハイライトする部位の色を変更
# 分子をコンテナに登録
view.DrawMolecule(m,
highlightAtoms = range(6),
highlightAtomRadii = radii,
highlightBonds = [])
# 描画
view.FinishDrawing()
SVG(view.GetDrawingText())
二色同時ハイライト
これまでのコードでは異なる部位のハイライトは色を変えることができました(例:原子ラベルとハイライトの色を変更)が,同じ原子や結合を2色で塗り分けることはできませんでした.それを可能にする機能が,2020.03のアップデートより追加されたDrawMoleculeWithHighlightsになります.
このメソッドにはMolオブジェクトと凡例に加えて,
- ハイライトする原子と色を指定した辞書
- ハイライトする結合と色を指定した辞書
- ハイライトする原子の半径を指定した辞書
- ハイライトする結合の太さを指定した辞書
を引数にとります.以下に示す具体例で使い方をみていきましょう.
以下のコードではグルタチオンを例にして,2つのアミド結合とシステイン部位の計3部分構造をハイライトしていきます.マッチする部位が一部重複していますので,その部位を二色で塗り分けることになります.
多色ハイライトに加えて,addStereoAnnotationを用いて絶対配置を表示しています.また原子や結合のプロパティにatomNoteやbondNoteを設定しておくとプロパティが構造と一緒に表記される機能を使い,各アミノ酸のカルボニル酸素にアミノ酸名を設定しています.
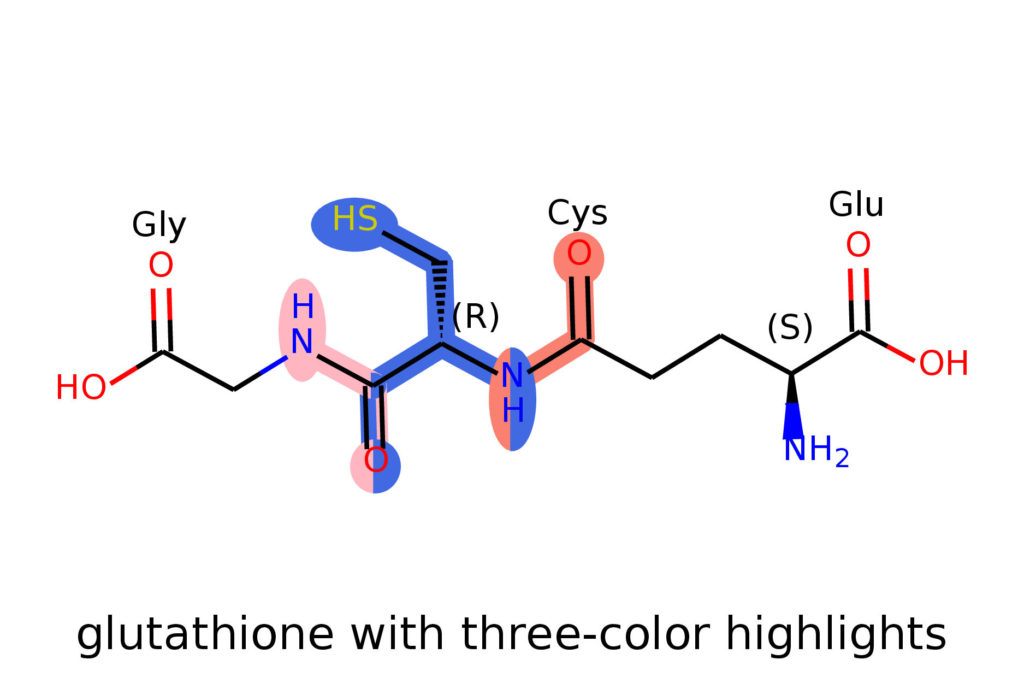
from collections import defaultdict
glutathione = Chem.MolFromSmiles('C(CC(=O)N[C@@H](CS)C(=O)NCC(=O)O)[C@@H](C(=O)O)N')
# アミド結合にマッチする原子
amide_pattn = Chem.MolFromSmarts('NC(=O)')
amides = glutathione.GetSubstructMatches(amide_pattn)
# システインにマッチする原子
cys_pattn = Chem.MolFromSmarts('NC(CS)C=O')
cys = glutathione.GetSubstructMatch(cys_pattn)
# 塗り分ける色の設定
salmon = ColorConverter().to_rgb('salmon') # アミドその1
lightpink = ColorConverter().to_rgb('lightpink') # アミドその2
royalblue = ColorConverter().to_rgb('royalblue') # システイン
# ハイライトする原子を辞書形式で作成
atom_highlights = defaultdict(lambda : [])
raddi_highlights = {}
for i, amide in enumerate(amides):
for atom in amide:
raddi_highlights[atom] = 0.2
if i == 0:
atom_highlights[atom].append(salmon)
else:
atom_highlights[atom].append(lightpink)
for atom in cys:
atom_highlights[atom].append(royalblue)
raddi_highlights[atom] = 0.2
# ハイライトする結合を辞書形式で作成
def get_matched_bonds(mol, matches):
matched_bonds = []
for bond in mol.GetBonds():
atom1 = bond.GetBeginAtomIdx()
atom2 = bond.GetEndAtomIdx()
if atom1 in matches and atom2 in matches:
matched_bonds.append(bond.GetIdx())
return matched_bonds
bond_highlights = defaultdict(lambda : [])
for i, amide in enumerate(amides):
bond_list = get_matched_bonds(glutathione, amide)
for bond in bond_list:
if i == 0:
bond_highlights[bond].append(salmon)
else:
bond_highlights[bond].append(lightpink)
for bond in get_matched_bonds(glutathione, cys):
bond_highlights[bond].append(royalblue)
# カルボニル酸素にアミノ酸名をプロパティとして登録
glutathione.GetAtomWithIdx(13).SetProp('atomNote', 'Gly')
glutathione.GetAtomWithIdx(3).SetProp('atomNote', 'Cys')
glutathione.GetAtomWithIdx(17).SetProp('atomNote', 'Glu')
# コンテナの作成とオプション設定
view = rdMolDraw2D.MolDraw2DSVG(450,300)
options = view.drawOptions()
options.addStereoAnnotation = True
options.annotationFontScale = 1.0
options.legendFontSize = 20
view.SetFontSize(1.3 * view.FontSize())
# 分子をコンテナに登録
view.DrawMoleculeWithHighlights(glutathione,
'glutathione with three-color highlights',
dict(atom_highlights),
dict(bond_highlights),
raddi_highlights,
{})
# 描画
view.FinishDrawing()
SVG(view.GetDrawingText())
終わりに
今回は「RDKitでの構造式描画を詳しく解説」という話題について,少し細かい内容から始めて,2015年より追加された描画コードを用いてSVG形式での書き出しについて詳細に見てきました.
rdMolDraw2Dモジュールを使うと,きれいな構造式を出力することが可能になります.また非常に細かい設定を行うことが可能なことも理解できたかと思います.
個人的にはこれら2つのモジュールの関係は,matplotlibでグラフを作成していく際に,1) MatLab風にpyplot.plot(x,y)で描画してく方法と,2) Figureオブジェクトを作ってからそこに描画していく方法,が混在しているのと似ていると感じています.matplotlibでは細かい設定を行いたい場合には後者のオブジェクト指向の方法を使う必要がありますが,前者の直感的なわかりやすさ・簡便さから両者が共存しています.
将来的にMolsToGridImageと同じように,MolToImageの描画方法も裏でrdMolDraw2Dを使うように変わる可能性もあるかもしれません.それでも探索的な使い方のDrawモジュールによる簡便な方法と,rdMolDraw2Dを直接用いて細かく設定を行う方法との使い分けが続くことになるのではないでしょうか(実はDraw.DrawingOptionsでも細かい設定が可能です).
今回は2次元構造の描画について学びましたので,次回は3次元構造の描画方法について学んでいきましょう.
>>次の記事:「py3Dmolを使って化学構造をJupyter上で美しく表示する」
コメント