「計算手法とエネルギー・最適化構造の関係:コンフォメーション探索における注意点」という記事では,Psi4を用いて様々な計算手法を用いてエネルギー計算を行うことで,
- 計算手法によって考慮できる相互作用が異なる
ことを説明しました。その結果
- 構造最適化できないことがある
- コンフォマー間のエネルギー差が逆転する場合がある
ことなどを具体例を通して見てきました。
ところで,エネルギー計算により得られるのは分子のエネルギーだけではありません。今回はシュレディンガー方程式の解である波動関数から得られる,様々な情報について見ていきたいと思います。

ライブラリの準備
今回もPsi4を量子化学計算エンジンとし,RDKitも使いながらコードの使い方を見ていきます。Psi4で用いる計算資源(CPU/RAM)については各人の環境にあわせて設定するようにしてください。また環境構築が難しい方用に,Google ColabでのPsi4の使い方を「Google ColabでPsi4:量子化学計算用のpython環境を手軽に構築」という記事で解説しています。参照してみてください。

## ライブラリのインポート
import psi4
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
import time
import rdkit
from rdkit import Chem
from rdkit.Chem import AllChem, Draw, PandasTools
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ', rdkit.__version__) # rdkit version: 2023.03.1
print('psi4 version: ', psi4.__version__) # psi4 version: 1.8
print('numpy version: ', np.__version__) # numpy version: 1.24.3
## SMILESをxyz形式に変換
def sm2xyz(smiles: str) -> str:
"""
SMILESで入力された分子をXYZ形式に変換する
Args:
smiles: 分子のSMILES
Returns:
str: 分子のXYZ形式
"""
mol = Chem.AddHs(Chem.MolFromSmiles(smiles))
AllChem.EmbedMolecule(mol)
AllChem.UFFOptimizeMolecule(mol)
conf = mol.GetConformer(-1)
xyz = '0 1'
for atom, (x,y,z) in zip(mol.GetAtoms(), conf.GetPositions()):
xyz += '\n'
xyz += f'{atom.GetSymbol()}\t{x}\t{y}\t{z}'
return xyz
## psi4の設定:各自の環境に合わせてください
psi4.set_num_threads(4)
psi4.set_memory('10GB')
Psi4のWavefunctionオブジェクトについて
psi4.core.Wavefunction.variable(key)
Psi4ではエネルギー計算や構造最適化計算の際にreturn_wfn引数をTrueにすることで,エネルギーに加えて波動関数Wavefunctionオブジェクトを返します。Wavefunctionオブジェクトにはエネルギーなどの値に加えて,「psi4とCubeファイル:分子軌道や静電ポテンシャルマップの可視化」という記事でも解説しているように,分子軌道などに関する情報が格納されています。さらにWavefunctionオブジェクトをもとにして,電荷や双極子モーメント,結合次数などの情報を取得することが可能です。
Wavefunctionオブジェクトに格納されている情報の多くは,Wavefunction.variables()でアクセスできる辞書オブジェクトとして格納されています。以下のコードでは,メタン分子をHF/STO-3Gレベルでエネルギー計算を行い,得られたWavefunctionオブジェクトの中身を表示しています。
methane = psi4.geometry(sm2xyz('C'))
psi4.set_output_file('methane_hf.log')
_, wfn = psi4.energy('hf/sto-3g',
molecule=methane,
return_wfn=True)
for key in wfn.variables():
print(key, '\t', wfn.variable(key))
エネルギーなどの他に双極子モーメントなどの値が格納されているのが確認できます。
NUCLEAR REPULSION ENERGY 13.200439741568875 ONE-ELECTRON ENERGY -78.9270492286628 PCM POLARIZATION ENERGY 0.0 PE ENERGY 0.0 SCF ITERATION ENERGY -39.724747984078306 SCF ITERATIONS 5.0 SCF TOTAL ENERGY -39.724747984078306 TWO-ELECTRON ENERGY 26.00186150301562 CURRENT DIPOLE [ 1.95569996e-07 2.00469358e-07 -1.61656964e-07] SCF DIPOLE [ 1.95569996e-07 2.00469358e-07 -1.61656964e-07]
Psi4における波動関数の解析法について
psi4.oeprop(wfn, property)
psi4.propertiesを用いることで所望の分子の性質を計算したい可能です。基本的な使い方はpsi4.energyやpsi4.optimizeと同じで,計算レベルと対象分子を指定します。さらにproperties引数で求めたい性質のリストを与えます。計算した値は全てWavefunctionオブジェクトに格納されますので,忘れずにreturn_wfn引数をTrueにしておきましょう。
また既に構造最適化計算などでWavefunctionオブジェクトが手元にある場合もあると思います。このような場合に波動関数の解析に使えるのがpsi4.oeprop(One-Electron properties)です。Wavefunctionオブジェクトと求めたい性質を引数として列挙します。
どちらの方法も以下のテーブルにあるような性質が計算可能です。またCCSDなど一部の計算手法では分極率など他の性質も計算可能です。詳しくは公式のマニュアルを参照してみてください。
| オプション | 内容 |
|---|---|
DIPOLE |
双極子モーメント |
QUADRUPOLE |
四極子モーメント |
GRID_ESP |
静電ポテンシャル |
ESP_AT_NUCLEI |
各原子における静電ポテンシャル |
MULLIKEN_CHARGES |
Mulliken電荷 |
LOWDIN_CHARGES |
Löwdin電荷 |
WIBERG_LOWDIN_INDICES |
Wiberg結合次数 |
MAYER_INDICES |
Mayer結合次数 |
MBIS_CHARGES |
MBIS電荷 |
以下のコードでは計算方法を確認するために,メタンに対しHF/6-311+G(2d,p)レベルで各原子における静電ポテンシャル(ESP_AT_NUCLEI)を計算し,実行速度を確認してみます。ここでは計算した値よりもメソッドの使い方に着目してみてください。
## psi4.properties
start = time.time()
_, wfn_prop = psi4.properties('hf/6-311+g(2d,p)',
molecule=methane,
properties=['ESP_AT_NUCLEI'],
return_wfn=True)
end = time.time()
print(f'psi4.properties: {end - start: .2f} sec')
## psi4.oeprop
_, wfn_oeprop = psi4.energy('hf/6-311+g(2d,p)',
molecule=methane,
return_wfn=True)
start = time.time()
psi4.oeprop(wfn_oeprop, 'ESP_AT_NUCLEI')
end = time.time()
print(f'psi4.oeprop: {end - start: .2f} sec')
実行結果は以下のようになりました。新たにシュレディンガー方程式を解かないpsi4.oepropの方が当然速くなっています。しかしoepropでは指定したWavefunctionオブジェクトが必要な情報を全て含んでいるかをチェックしません。そのため,公式フォーラムによると,psi4.oepropよりもpsi4.propertiesの利用が推奨されているようです(リンク)。
psi4.properties: 0.31 sec psi4.oeprop: 0.00 sec
電子密度解析とは何か
有機化学反応の多くは,「カチオン」と「アニオン」,「正電荷」と「負電荷」によって理解されています。例えばカルボニル基の求電子性は「C=O」の結合の電荷の偏りによって説明されます。
このような形式的な「電荷」を,波動関数の絶対値の二乗として得られる電子密度を用いて各原子に割り当てることを密度解析(population analysis)といいます。
割り当てるルールによっていくつかの方法が知られていますが,よく用いられるのは
- Mulliken(マリケン)密度解析
- Löwdin(ローディン)密度解析
の2つで,どちらもpsi4で利用可能です。
他の電荷としては
- 自然密度解析(NPA)
や静電ポテンシャルへのフィッティングによって電荷を決める
- Merz–Kollman電荷
などが有名です。さらにPsi4では新しめの手法であるMBIS法(Minimal Basis Iterative Stockholder,論文リンク)も実装されています。
名古屋大学情報連携基盤センターニュース Vol.7, No.1-2008.2-
Mulliken電荷とLöwdin電荷
イオン結合性化合物の場合に,Mulliken電荷の方がよい結果を与えることが知られています。一方で,共有結合性化合物ではどちらもあまり変わらないことから,一般的にはMulliken電荷が用いられています。
とはいえ,先ほど述べたように電荷とは観測できない概念的なものですので,興味のある化合物群で傾向を見て判断するのが重要です。
Mulliken電荷
psi4.oeprop(wfn, ‘MULLIKEN_CHARGES’)
wfn.variable(‘MULLIKEN CHARGES’)
Mulliken密度解析では基底関数系にSTO-3Gなどの最小基底系を利用することを想定しています。そのため,大きな基底関数系,特に分散関数を含むものを用いた場合には電荷分布が大きく変化することがあります。このような基底関数系による一貫性の無さがMulliken電荷の問題点として広く認識されています。
以下のコードでは塩化メチルについて,基底関数系の大きさを変えていくことでMulliken電荷がどのように変化していくかを見ていきます。
MeCl = psi4.geometry(smi2xyz('CCl'))
basis_set = ['sto-3g', '3-21G', 'cc-pvdz', 'aug-cc-pvdz', 'cc-pvtz',
'aug-cc-pvtz', 'cc-pvqz', 'aug-cc-pvqz', 'aug-cc-pv5z']
df = pd.DataFrame()
psi4.set_output_file('MeCl.log')
psi4.optimize('hf/sto-3g', molecule=MeCl)
for basis in basis_set:
theory = f'hf/{basis}'
_, wfn_mecl = psi4.properties(theory,
molecule=MeCl,
properties=['MULLIKEN_CHARGES'],
return_wfn=True)
df[basis] = wfn_mecl.variable('MULLIKEN CHARGES')
df.index = [MeCl.symbol(i) for i in range(MeCl.natom())]
df.round(2)
結果は以下の表のようになりました。電気陰性度から考えると炭素の方が塩素よりもポジティブになりそうですが,多くの場合に塩素原子の方がポジティブになっていることがわかります。また分散関数の有無によって値が大きくぶれてしまい,計算レベルをあげても一定値に収束しないことが伺えます。
| sto-3g | 3-21G | cc-pvdz | aug-cc-pvdz | cc-pvtz | aug-cc-pvtz | cc-pvqz | aug-cc-pvqz | aug-cc-pv5z | |
|---|---|---|---|---|---|---|---|---|---|
| C | -0.14 | -0.75 | -0.06 | 0.13 | -0.14 | -0.67 | -0.15 | -0.67 | -0.28 |
| CL | -0.17 | -0.04 | -0.20 | -0.19 | -0.21 | -0.25 | -0.18 | -0.20 | -0.31 |
| H | 0.10 | 0.26 | 0.09 | 0.02 | 0.12 | 0.31 | 0.11 | 0.29 | 0.20 |
| H | 0.10 | 0.26 | 0.09 | 0.02 | 0.12 | 0.31 | 0.11 | 0.29 | 0.20 |
| H | 0.10 | 0.26 | 0.09 | 0.02 | 0.12 | 0.31 | 0.11 | 0.29 | 0.20 |
Löwdin電荷
psi4.oeprop(wfn, ‘LOWDIN_CHARGES’)
wfn.variable(‘LOWDIN CHARGES)
Löwdin電荷についても同様にpsi4.propertiesやpsi4.oepropを用いて求めることができます。
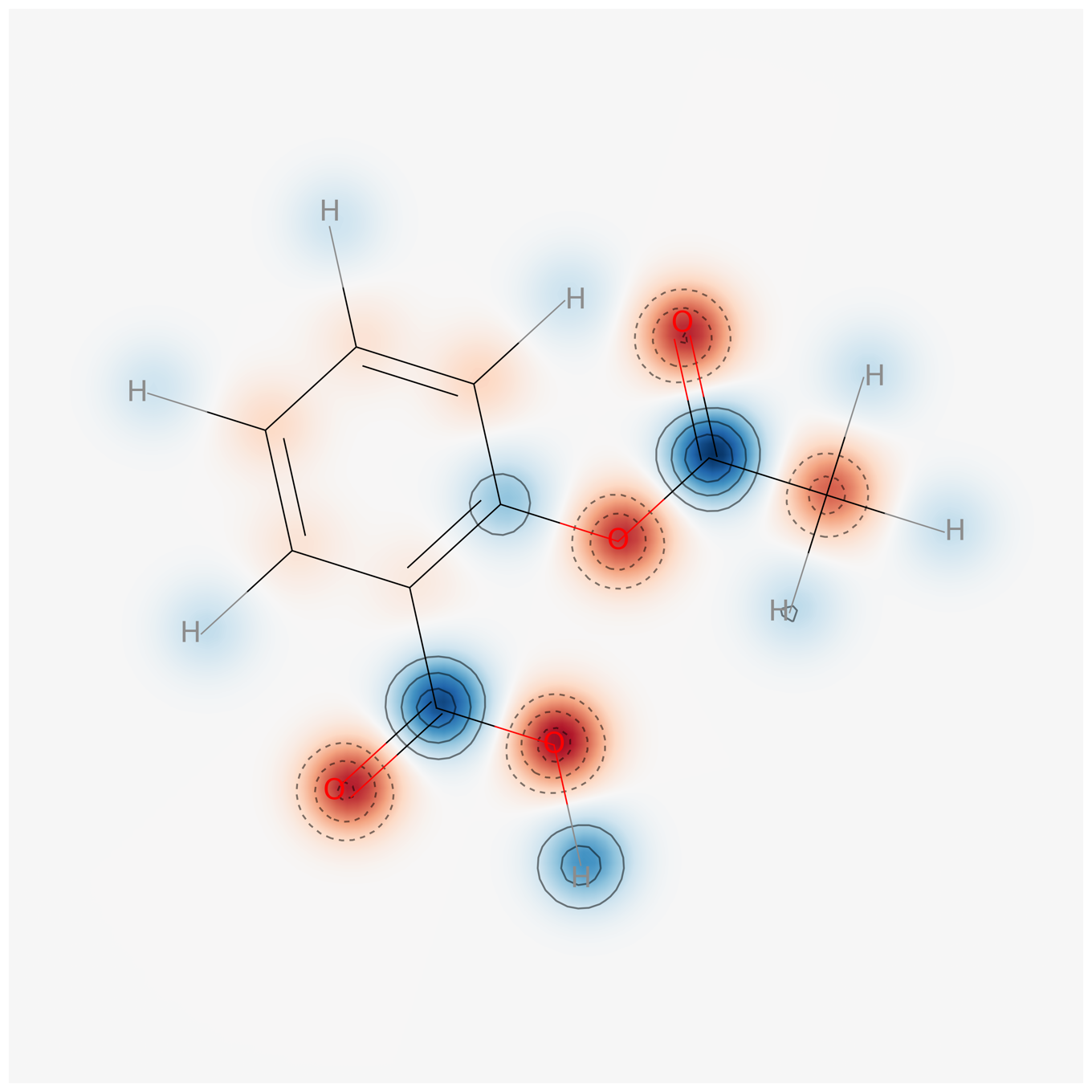
Mulliken電荷をRDKitの類似度マップで可視化
「RDKitの類似度マップを用いて原子ごとの寄与を可視化する」という記事でGasteiger電荷について行った電荷マップの可視化をMulliken電荷でも行ってみます。
分子にはアスピリンを用いることとし,HF/STO-3Gで計算します。
## 分子の構築
psi4.set_output_file('aspirin.log')
aspirin = psi4.geometry(sm2xyz('CC(OC1=C(C(=O)O)C=CC=C1)=O CC(=O)OC1C=CC=CC=1C(O)=O'))
_, wfn_asp = psi4.optimize('hf/sto-3g',
molecule=aspirin,
return_wfn=True)
rdkit_aspirin = Chem.AddHs(Chem.MolFromSmiles('CC(OC1=C(C(=O)O)C=CC=C1)=O CC(=O)OC1C=CC=CC=1C(O)=O'))
## Mulliken電荷の計算
psi4.oeprop(wfn_asp, 'MULLIKEN_CHARGES')
mulliken = wfn_asp.variable('MULLIKEN CHARGES')
## 類似度マップで可視化
from rdkit.Chem.Draw import SimilarityMaps
fig = SimilarityMaps.GetSimilarityMapFromWeights(rdkit_aspirin, mulliken, colorMap='RdBu')
青い部分が正電荷を帯びている部分になります。
双極子モーメント
psi4.oeprop(wfn, ‘DIPOLE’)
psi4.variable(‘SCF DIPOLE’)
双極子モーメントの各座標成分はpsi4.variable(‘SCF DIPOLE’)で取得可能です。双極子モーメントのXYZ成分がnumpy配列として取得できますので,分子全体の双極子モーメントの大きさを求めたい場合は自分でベクトルの大きさを計算する必要があります。
$$ dipole = \sqrt{x^2 + y^2 + z^2} $$
また出力された値の単位はSI単位系(au)ですので,汎用されるデバイ(D, debye)へと変換するには2.54を乗じる必要があります。Psi4ではこのような定数はpsi4.constantsに準備されていますので,単位の変換は容易に可能です。
下のコードではメタノールの双極子モーメントをHF/STO-3Gレベルで計算したところ,双極子モーメントは1.51 Dでした。
au2debye = psi4.constants.dipmom_au2debye
methanol = psi4.geometry(sm2xyz('CO'))
psi4.set_output_file('methanol.log')
_, wfn_meoh = psi4.optimize('hf/sto-3g', molecule=methanol, return_wfn=True)
psi4.oeprop(wfn_meoh, 'DIPOLE')
dipole_methanol = wfn_meoh.variable('SCF DIPOLE')
dipole = np.sqrt(np.sum(dipole_methanol ** 2))
print(f'dipole moment: {au2debye * dipole: .2f} D')
## 1.51
Wiberg結合次数
psi4.oeprop(wfn, ‘WIBERG_LOWDIN_INDICES’)
wfn.variable(‘WIBERG LOWDIN INDICES’)
よく構造化学の分野の論文で使われているWiberg結合次数も求めることが可能です。結果はwfn.variable(‘WIBERG LOWDIN INDICES’)で結合次数行列がPsi4のMatrixオブジェクトとしてアクセス可能です。値を取り出すにはnumpy配列へと変換してから利用する必要があります。
以下の例ではスチレンを用いて,通常の二重結合と芳香族結合の結合次数が異なることを確かめてみましょう。
styrene = psi4.geometry(sm2xyz('C=Cc1ccccc1'))
psi4.set_output_file('stylene_Wiberg.log')
_, wfn_stylene = psi4.optimize('hf/sto-3g', molecule=styrene, return_wfn=True)
psi4.oeprop(wfn_stylene, 'WIBERG_LOWDIN_INDICES')
df_stylene = pd.DataFrame(wfn_stylene.variable('WIBERG LOWDIN INDICES').to_array())
df_stylene.index = [styrene.symbol(i) for i in range(styrene.natom())]
df_stylene.round(2)
出力は以下のように16原子 x 16原子の結合次数行列になります。下表で太字にしたように,
- 二重結合:1.95
- 芳香族結合:1.40から1.46
と違いが出ています.
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| C | 0.00 | 1.95 | 0.00 | 0.03 | 0.00 | 0.02 | 0.00 | 0.03 | 0.98 | 0.98 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| C | 1.95 | 0.00 | 1.05 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.98 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| C | 0.00 | 1.05 | 0.00 | 1.41 | 0.00 | 0.11 | 0.00 | 1.40 | 0.00 | 0.01 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 |
| C | 0.03 | 0.00 | 1.41 | 0.00 | 1.45 | 0.00 | 0.11 | 0.00 | 0.00 | 0.00 | 0.00 | 0.98 | 0.00 | 0.01 | 0.00 | 0.01 |
| C | 0.00 | 0.01 | 0.00 | 1.45 | 0.00 | 1.44 | 0.00 | 0.11 | 0.00 | 0.00 | 0.00 | 0.00 | 0.98 | 0.00 | 0.01 | 0.00 |
| C | 0.02 | 0.00 | 0.11 | 0.00 | 1.44 | 0.00 | 1.44 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.98 | 0.00 | 0.01 |
| C | 0.00 | 0.01 | 0.00 | 0.11 | 0.00 | 1.44 | 0.00 | 1.46 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.98 | 0.00 |
| C | 0.03 | 0.00 | 1.40 | 0.00 | 0.11 | 0.00 | 1.46 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.98 |
| H | 0.98 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.98 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.00 | 0.98 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.00 | 0.00 | 0.00 | 0.98 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.00 | 0.00 | 0.01 | 0.00 | 0.98 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.98 | 0.00 | 0.01 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.98 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
| H | 0.00 | 0.00 | 0.00 | 0.01 | 0.00 | 0.01 | 0.00 | 0.98 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 | 0.00 |
終わりに
今回は「計算化学における電荷:psi4を用いた電子密度解析」という話題について,
- Psi4のWavefunctionオブジェクトと波動関数解析について
- 電子密度解析とは何か
- Mulliken電荷とLöwdin電荷
- Psi4を用いた密度解析のやりかた
- RDKitの類似度マップを用いた可視化
- 双極子モーメントの求め方
- Wiberg結合次数の求め方
などについて説明していきました。
今回取り扱った以外にもpsi4.Wavefunctionオブジェクトからは,HOMOやLUMOといった分子軌道に関する情報も取得可能で,外部プログラムと組み合わせることで軌道の可視化も可能になります。こちらについては,「psi4とCubeファイル:分子軌道や静電ポテンシャルマップの可視化」という記事で説明しています。
これまでエネルギー計算と構造最適化計算について説明を重ねてきました。次回は,調和振動子近似の説明からpsi4における振動数計算について説明することで計算化学について理解を深めていきたいと思います。
>>次の記事:「Psi4における振動数計算:IRスペクトルや異性体間のエネルギー差を計算」
コメント
たいへん勉強になります。
ありがとうございます。
一つ教えて頂きたいことがございます。
psi4 を用いて、下記のような励起準位の情報取得は可能でしょうか?
(psi4のHPと睨めっこしていますが、量子計算初心者ということもあり行き詰まっております)
もし可能であれば、参考になるサイト等あれば教えて頂ければ幸いです。
####
Excited State 2: Singlet-A 3.8723 eV 320.19 nm f=0.1017
=0.000109 ->111 0.66378
110 ->113 -0.20418
####
ご質問ありがとうございます.
GaussianのCISまたはTD-DFTのアウトプットファイルからの抜粋とお見受けしました.
TD-DFTについては近々リリース予定(?)のpsi4 version 1.4で利用可能になるようです.ChemRxivに上がっているプレプリントのFig6にも簡単な例が載っていまして,
手許の環境ではベンゼンを対象としてこちらのコードは動かすことができました.
しかし汎関数をCAM-B3LYPに変えるとTDに対応していない旨のエラーが出るなど,何が実装されているか細かくコードレベルで見ていかないとわからないのが現状です.第1,第2励起エネルギーなども取得できましたが,今の段階ではマニュアルが揃っておらず学習コストが高そうです.
他の(無償)ソフトですと,やはりFireflyかGamessを用いるのがよいと思います.日本語ですと例えば以下の記事が参考になると思います.
– 「紫外可視吸収スペクトルを計算してみよう」:こちらの記事では,TD-DFTを用いたUV/Visスペクトルの描画方法が丁寧に解説されています
– 「ホルムアルデヒドの励起状態計算」:こちらの記事ではホルムアルデヒドを例にCIS,TD-DFT,EOM-CCSDを用いた垂直遷移と,CISを用いた励起状態での構造最適化の解説がなされています
今後ともよろしくお願いいたします.
早速のご回答ありがとうございます。
TD-DFT の抜粋でございます。
どこを探しても説明が見当たらなかったので、TD-DFTがまだ実装されていないとのこと納得しました。
Gamess を用いるか、psi4 1.4 のリリースを待つか少し検討してみます。
理論計算、インフォオマティクス関連の記事楽しみにしております。
今後ともよろしくお願いいたします。
いつもたいへん参考にさせていただいております。
ありがとうございます。
1つ教えていただきたいのですが分極率はpsi4で計算できるのでしょうか。
もし可能であれば参考になるものを教えていただければと思います。
よろしくお願いします。
コメントありがとうございます。分極率(polarizability)に関してですが,一部の計算手法では実装されています。本文の方に追記しておきました。