我々の目はモノの形やパターンを読み取る能力に優れています.化学者が用いる構造式は人間の目で理解しやすいように設計された分子の表現です.例えば下のアスピリンの構造式を見るだけで,化学者は多くの情報を瞬時に理解できます.
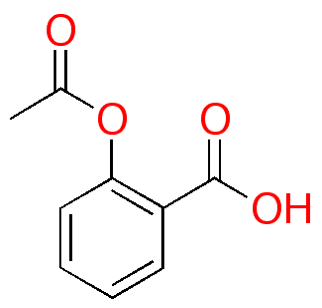
ところがコンピュータにとってはこのような構造式の画像ファイルはとても理解しにくいものです.
今回はコンピュータにとって理解しやすい化合物の表現について,特にMOLファイルとSDFというものについて学んでいきます.特にSDFは構造情報の受け渡しによく使われるフォーマットですので,
- どこでSDFを手に入れるか
- どうやってSDFを作成するか
についても説明していきます.
分子のグラフ表現:原子間の結合の表し方
分子は原子をノード,結合をエッジと見なしたグラフと考えられます.グラフではある原子が他の原子とどのように繋がっているかを表現することができます.なお原子間の結合数がわかれば水素原子は後から付加可能ですので,コンピュータ上で分子を表現する際には水素原子は省略されることが多いです.
例えば下のグラフ構造はなんとなくイソブタン(2−メチルプロパン)に見えないでしょうか?
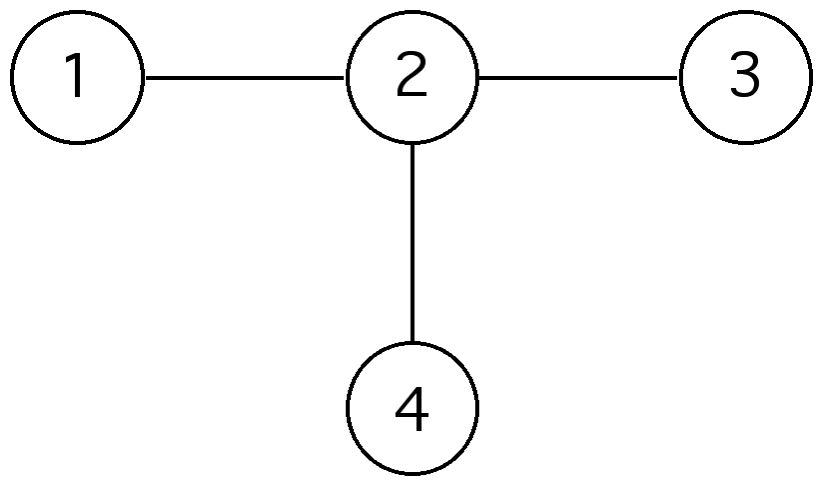
グラフ構造においては原子間の繋がりだけが重要ですので,下のように結合の有無を「0」と「1」で表現可能です.
$$ \left(
\begin{array}{cccc}
0&1&0&0 \\
1&0&1&1 \\
0&1&0&0 \\
0&1&0&0 \\
\end{array}
\right ) $$
このように分子内の結合を表現した行列は
- 対称行列
- ほとんどの要素が0
という特徴があります.また原子数が大きくなると行列もどんどん大きくなってしまいますので,結合を作っている部分のみを取り出した方が便利です.原子間の結合を表すのに下のようなフォーマットを考えます.
このフォーマットに従うと,例えば下の表は1番目と2番目の原子が単結合で繋がっていて,1番目と4番目の原子が単結合で,,,という上の行列と同じ情報を下のように表わすことができます.
1 2 1 2 3 1 2 4 1
この記法を使うことで,伝える情報量を保ちながら文字数を減らすことに成功しました.このリストを「結合リスト」と呼びます.
原子の種類と座標
これまでどのように原子同士が結合しているかを示す表現方法を学びました.分子を実際に構築するにはもう少し情報が必要です.すなわち,
- 原子の種類
- 原子の座標
がわからないと分子を組み立てられません.色々な表記が考えられますが,一般によく使われるのは下記の書式です.
このスタイルに従うと,例えば先ほどのイソブタンは下のように書けます.
-0.7145 -0.6188 0.0000 C
0.0000 -0.2062 0.0000 C
0.7145 -0.6188 0.0000 C
0.0000 0.6188 0.0000 C
このリストを「アトムリスト」と呼びます.
MOLファイル
これまで「結合リスト」と「アトムリスト」を説明してきましたが,いよいよMOL(読み方:モル)ファイルの説明に入ります.
MOLファイルとは?
もともとMOLファイルはMolecular Design Limited社の運営するデータベース群の入出力フォーマットでしたが,現在ではケモインフォマティクスにおけるデータ交換フォーマットとしてデファクトスタンダードの1つになっています.またその歴史的由来から「MDL MOLファイル」とも呼ばれます.
MOLファイルの構成
MOLファイルの構成は,基本的には「アトムリスト」と「結合リスト」を組み合わせて,ヘッダーとフッターを追加したものになります.
下の例ではChemDrawで書いたアラニンをMOLファイルに変換したものになります.各行の説明は以下の通りです.
| 行数 | 説明 |
|---|---|
| 1 | <ヘッダー>化合物IDなど |
| 2 | <ヘッダー>作成したいアプリケーションなど |
| 3 | 空行 |
| 4 | 原子数,結合数,キラルの有無,などの数え上げ |
| 5-10 | アトムリスト |
| 11-15 | 結合リスト |
| 16 | 終了 |
alanine.cdxml
ChemDraw09291817512D
6 5 0 0 0 0 0 0 0 0999 V2000
-0.3572 -0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 0.2063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0693 0.2051 0.0000 N 0 0 0 0 0 0 0 0 0 0 0 0
1.0693 -0.2045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3572 1.0284 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3572 -1.0284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0
1 3 1 0
2 4 1 0
2 5 2 0
1 6 1 6
M END
なお4行目のカウントリストでは
- 原子数とアトムリストの数
- 結合数と結合リストの数
などが一致していないといけません.またGaussViewで作成したSDFは4行目の項目数が多く,RDKitで読み込もうとするとエラーになります.
今回は中性分子ですが,カチオンなど電荷が存在する場合は結合リストと終了表示の間にフッターとして書き込まれます.
SDF
SDFは「Structure-Data File」というもので,拡張子が「.sdf」であることからSDFファイルとも呼ばれることもありますが,ここでは単にSDFと呼んでいくことにします.
SDFの構成
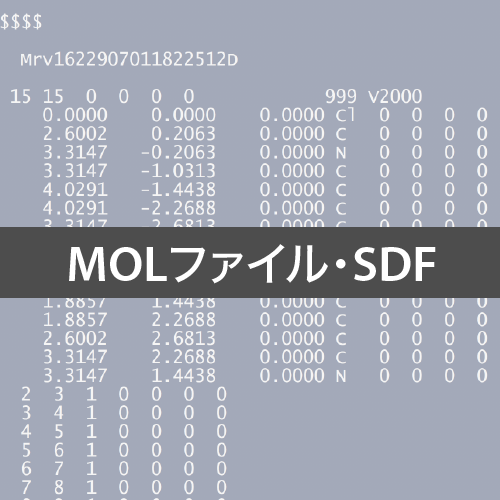
SDF(読み方:エスディーエフ)は基本的にMOLファイルをまとめて,複数分子を1つのファイルに収めたものです.分子の区切り記号として「$$$$」が使われます.
SDFの特徴として,別途分子のプロパティ・属性を持たせることが出来るという点があげられます.例えば試薬会社から入手したSDFには,カタログナンバーやCAS番号などが登録されています.
それでは具体例を見ながら見ていきましょう.フォーマットは表の通りで,「> <プロパティ名>」でプロパティを設定します.
| 行数 | 説明 |
|---|---|
| 1- | 最初の分子のMOLブロック |
| 3 | MOLブロック終了 |
| 4 | プロパティ名 |
| 5 | プロパティ設定 |
| 6 | 空行 |
| 7- | 次のプロパティ |
| 22 | 最初の分子の終了 |
| 23- | 次の分子のMOLブロック |
下の例はfluorochemから取得したSDFの抜粋になります.プロパティとして
- カタログナンバー
- 化合物名
- CAS番号
が設定されているのがわかります.
・・・MOLブロック・・・ M END > <Code> 006795 > <Name> Methyl 2,5-bis(trifluoroethoxy)benzoate > <CAS> 35480-31-0 $$$$ ・・・次の分子のMOLブロック・・・ Mrv17140 01082123222D
どこでMOLファイル・SDFを手に入れるか?
例えば,
- 個人的にツールを使って分析したい
- インハウスで簡単なデータベースを作成したい
といった場合にまず必要になるのは化合物データです.ここではどこでMOLファイル・SDFを手に入れるかについて,いくつか方法を紹介していきます.
試薬会社のウェブサイト
もっとも手軽なのが試薬会社のウェブサイトから手に入れる方法です.代理店経由で仕事として頼むとウェブサイトからダウンロードできなくても,カタログのSDFが手に入ることはままありますが,ここでは個人的に使いたい場合にダウンロードできるという観点から紹介しています.
当然すべてのサプライヤーをチェックしきれていませんが,SDFが取得できる会社は思ったより少ない印象です.
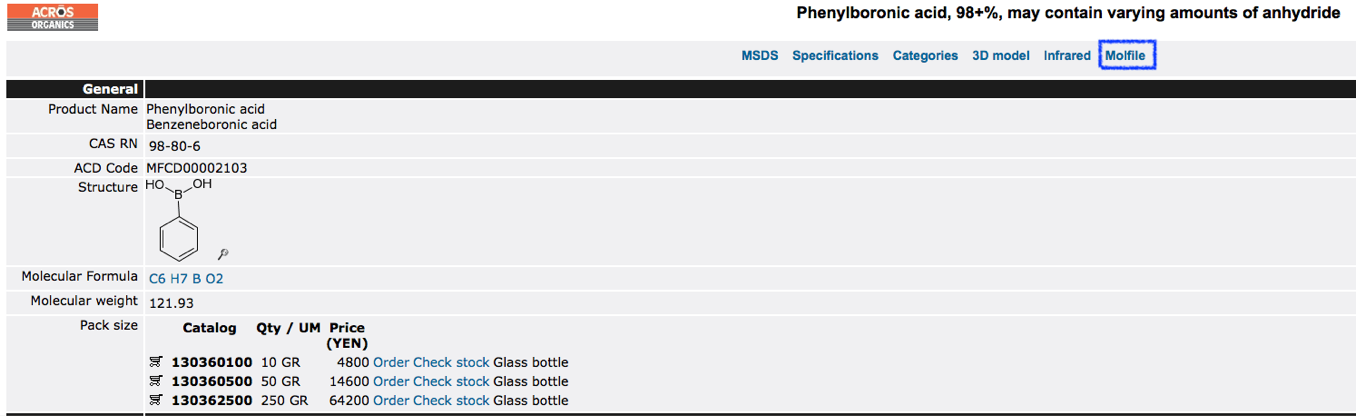
Acros
Acrosでは化合物の個別ページからMOLファイルのダウンロード可能です.MOLファイルだけですと,正直あまり使うことはなさそうです.
fluorochem
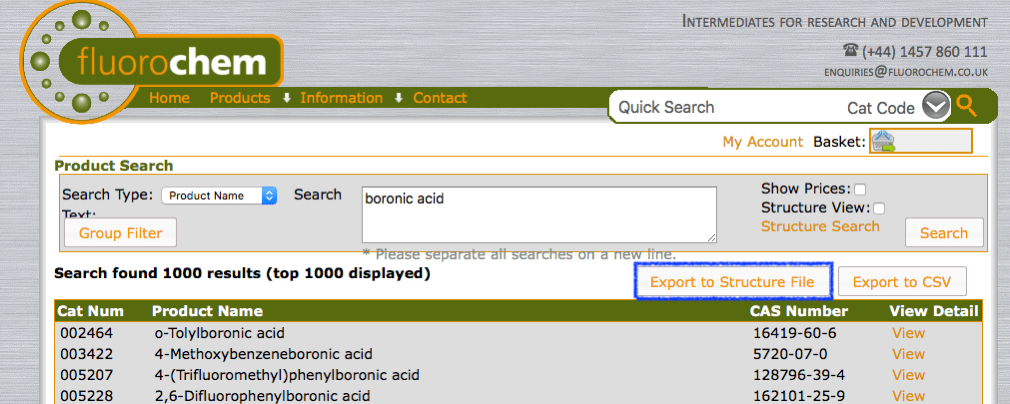
fluorochemでも化合物検索をした後のリストがSDFとしてダウンロード可能です.
Apollo Scientific
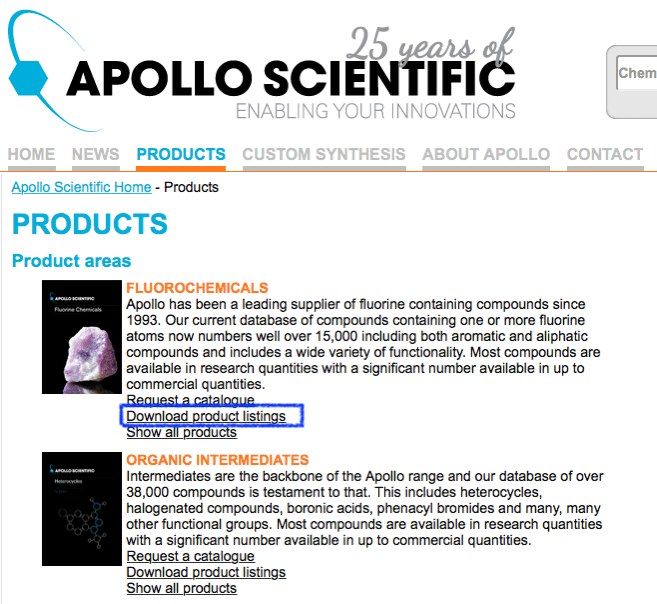
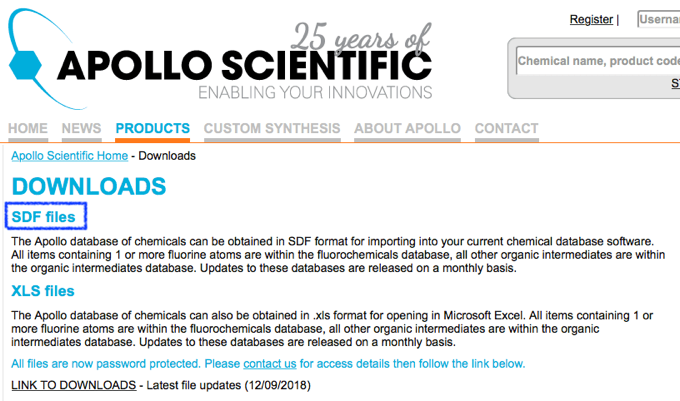
Apollo Scientificでは試薬カタログのSDFがダウンロード可能ですが,パスワードがかかっていますのでメールアドレスを登録する必要があります.
Enamine
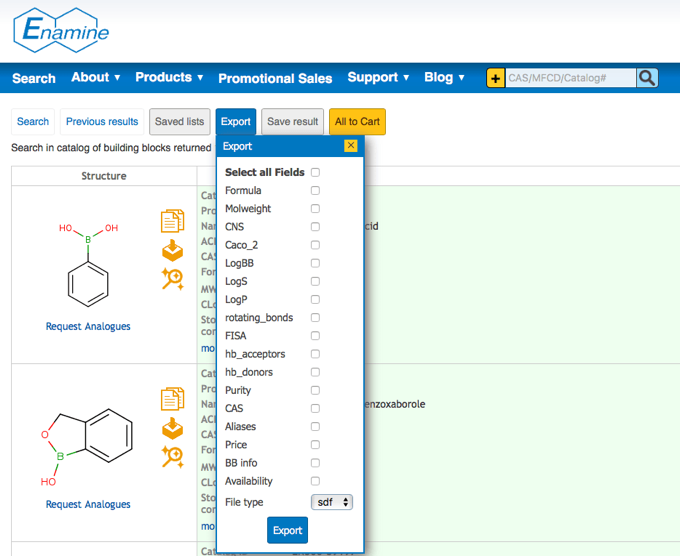
Enamineからは試薬検索結果,カタログのどちらもSDFでダウンロード可能です.
こちらが試薬の検索結果です.付記するプロパティを選択可能です.
カタログのダウンロードはこちらから.一部抜粋と全ての化合物が選択可能です.
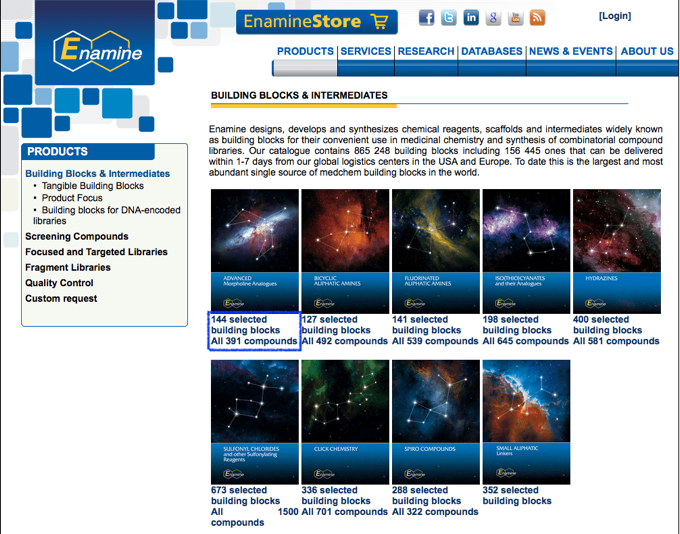
Matrix Scientific
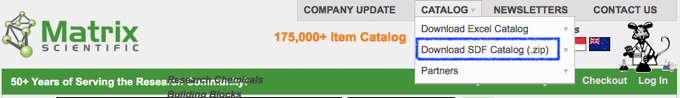
Matrix Scientificでは試薬カタログがSDFとしてダウンロード可能です.
データベース
化合物データベースからもMOLファイルやSDFの取得が可能です.
PubChem
アメリカNIHが管理するPubChemでは化合物の構造・物性データや,アッセイ結果などが収載されています.
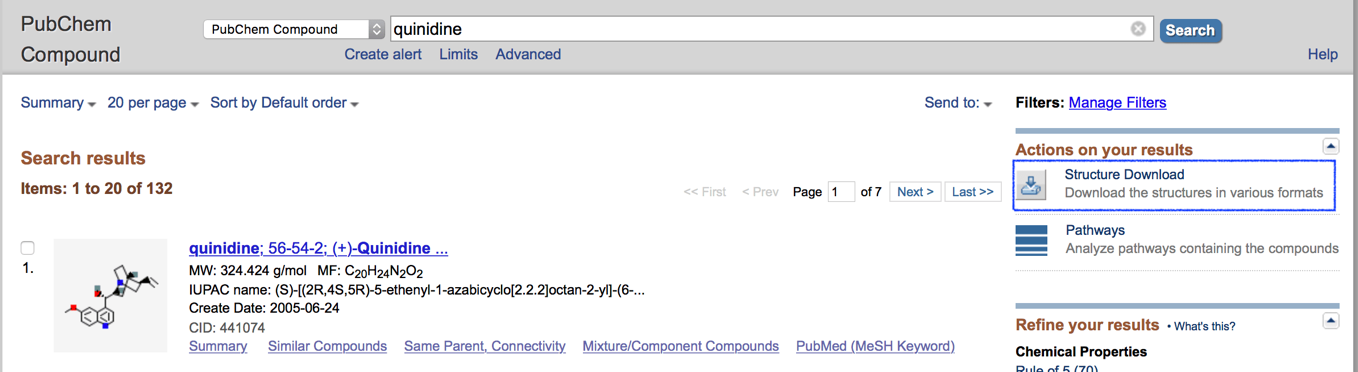
化合物を検索した場合にはリストのSDFが取得可能です.下の例では「quinidine」で検索しています.
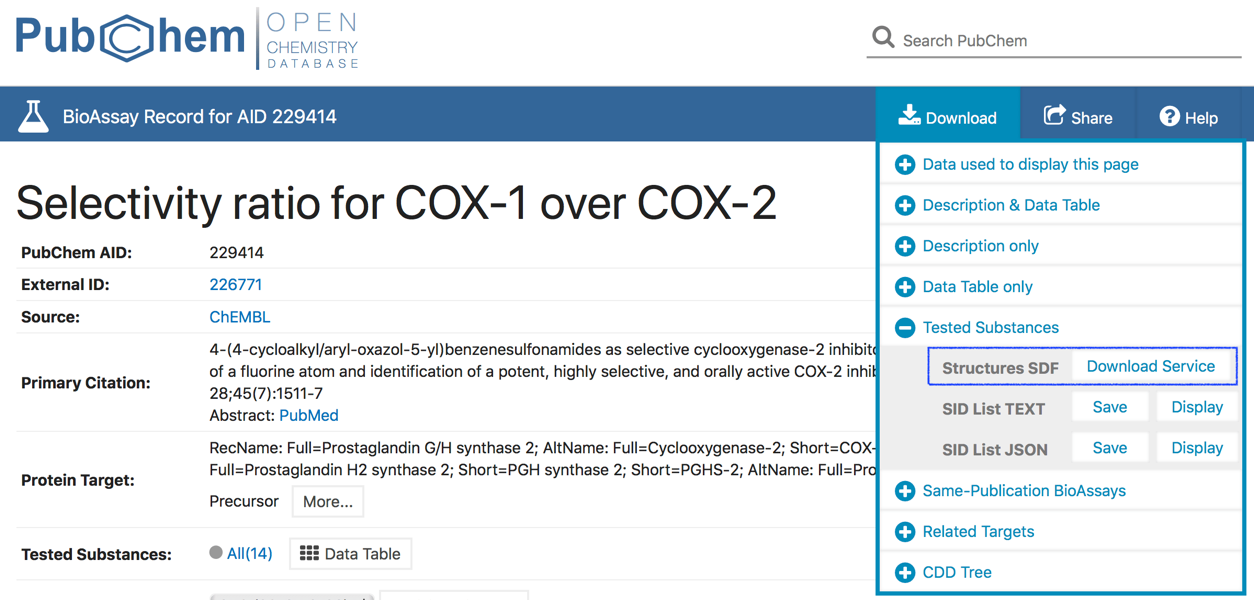
アッセイでも化合物リストの取得が可能です.
ChEMBL
文献より生理活性物質の情報を集めたChEMBLからも化合物情報の取得が可能です.
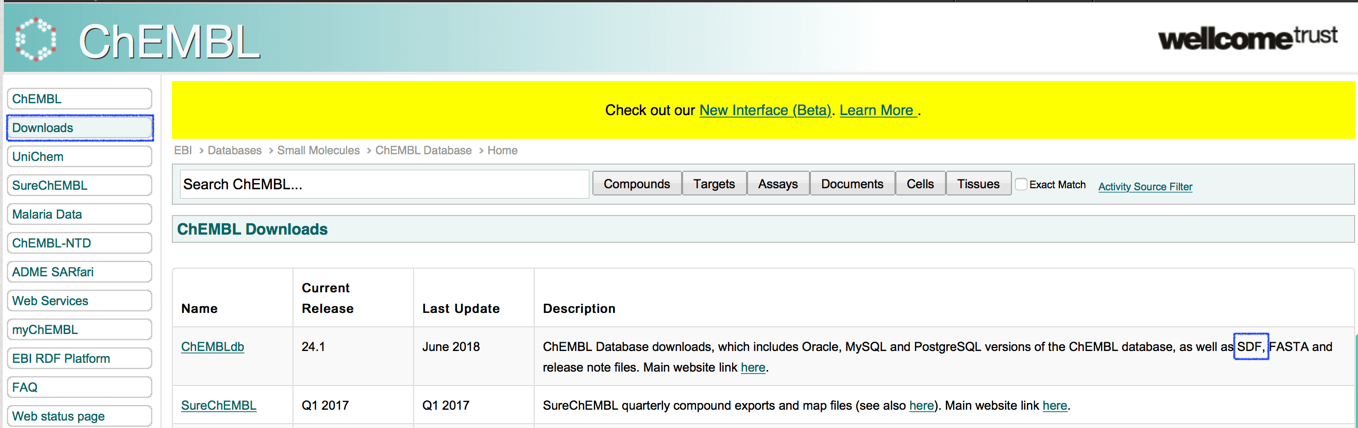
全データベースをダウンロードしたい場合は,「Downloads」から行います.
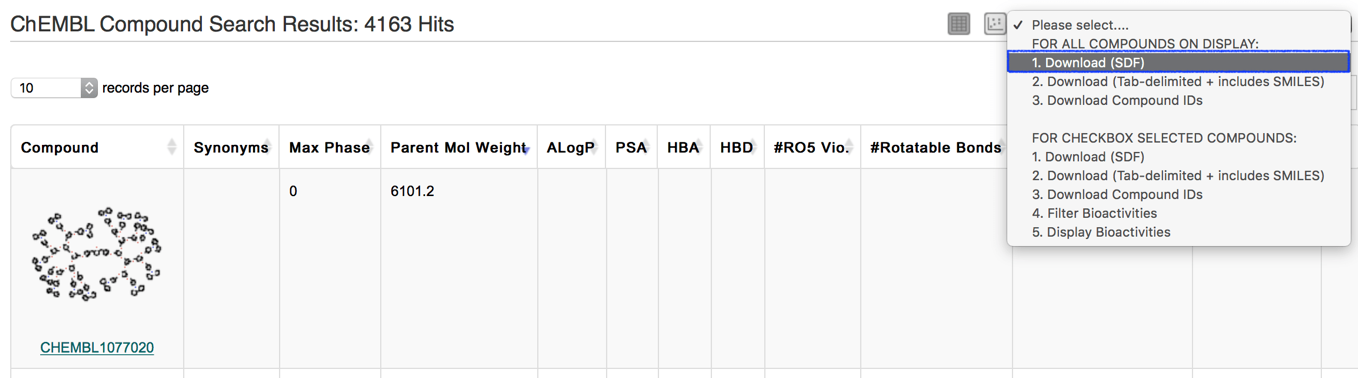
また検索結果の取得も可能です.この例ではカルバゾール骨格を有するリガンドを検索しました.
ソフトを用いて自分で作成
MOLファイルやSDFは極めて一般的な化合物情報フォーマットなので,多くのソフトがこれらの形式での書き出しに対応しています.ここでは実験化学者に馴染みのあるChemDrawによる方法と,ケモインフォマティクス用のpythonライブラリであるRDKitを使った方法について触れます.
ChemDraw
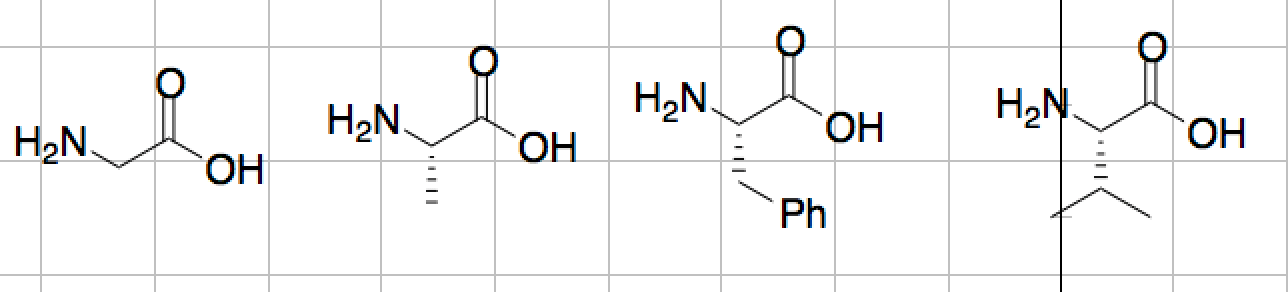
例えば下のように4つのアミノ酸を描画したChemDrawファイルがあるとします.
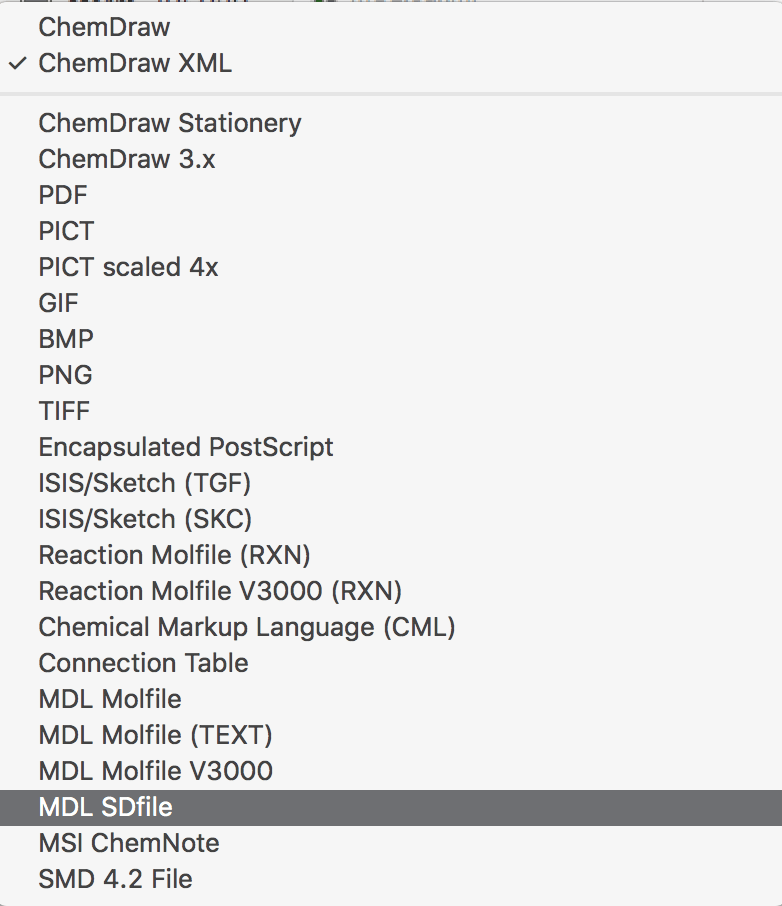
これを別ファイルで保存(save as)しようとすると,選べるファイル形式の中にSDFがありますのでそれを選ぶだけで,これらの構造が記入されたSDFが得られます.
RDKit
RDKitの場合はまず分子であるMOLオブジェクトを作成することになります.もちろんSDFからも作成できますが,SMILESなど他の方法から作ったMOLオブジェクトをMOLファイルにしたいとします.
MOLブロックへの書き出しが容易にできますので,それをファイルオブジェクトとして保存すればよいです.
from rdkit import Chem
mol = Chem.MolFromSmiles(‘C1=CC=CC=C1’)
mb = Chem.MolToMolBlock(mol)
with open('./benzene.mol', 'w') as f:
f.write(mb)
終わりに
今回はコンピュータが扱いやすい化合物情報のフォーマットについて,特にMOLファイルとSDFについて説明してきました.
MOLファイルは「コネクションテーブル」と呼ばれるものを基本とした化学情報フォーマットになります.次回は「線形表記法」と呼ばれるSMILES記法とInChI記法についてみていきたいと思います.
>>次の記事:「SMILES記法は化学構造の線形表記法」
コメント