創薬化学の歴史において天然物は非常に重要な位置を占めています.現在でも多くの薬は天然物やその誘導体です.
そのため過去には大手製薬会社は「天然物化学」部門を所有していましたが,スループットの低い天然物のスクリーニングは徐々に採算が合わなくなって閉鎖に追い込まれていきました.
ところが,その後のコンビナトリアルケミストリーの台頭と失敗を経て,化合物ライブラリーの質の重要性が明らかとなりました.例えば「ケモインフォマティクスで多様な化合物ライブラリーを構築する」という記事で説明したように,スクリーニングのヒット率向上のためには,ライブラリー化合物の構造多様性を上げることなどが意識されています.
創薬化学で一般的に合成される化合物と比べると,天然物の構造的特徴としては,
- 縮環構造が多い
- 不斉中心が多い
- 芳香環の数が少ない・sp3炭素が多い
- 酸素原子の割合が多く,窒素原子の割合は少ない
などが挙げられます.
こういった構造的特徴を何らかの方法で評価し,天然物らしい化合物をライブラリーに組み込むことができれば,化合物ライブラリーの質を向上させることができる可能性があります.
今回の記事では,
- ケモインフォマティクスにおける「天然物らしさ」とはなにか
- RDKitを用いてどのように化合物の天然物らしさを評価するか
について学んでいきたいと思います.
「Natural Product-likeness Score and Its Application for Prioritization of Compound Libraries」J. Chem. Inf. Model. 2008, 48, 68–74. (DOI: 10.1021/ci700286x)
天然物化合物の特徴とは?
「天然物らしさ」をライブラリーデザインの指標とするためには,天然物化合物の特徴を理解する必要があります.合成化合物と比較すると,天然物は構造的にはさきほど述べたように
- 縮環構造が多い
- 不斉中心が多い
- 芳香環の数が少ない・sp3炭素が多い
- 酸素原子の割合が多く,窒素原子の割合は少ない
という特徴を有します.また物理化学的パラメータとしては
- 脂溶性が高い
- 極性表面積が多い
などといった特徴があることが知られています.
過去にもこのような指標をもとに「天然物らしさ」を評価しようという試みはたくさん発表されています.
また構造多様性や天然物に頻出する骨格に着目したライブラリーの合成指針として,Schreiberの提唱した「多様性指向型合成:DOS」やWaldmannが推進している「生物指向型合成:BIOS」などもよく知られるところです.
今回とりあげるNP-likenessスコアは,「sp3炭素の割合」などといった単一の指標ではなく,部分構造の網羅的な解析によるスコア化を行ったものになります.
天然物らしさ・Natural Product-likeness (NP-likeness)スコアとは?
「Natural Product-likeness Score and Its Application for Prioritization of Compound Libraries」という論文の著者らは,ある化合物と天然物とがケミカルスペース中に位置する近さをNP-liknessとしてスコア化を試みています.薬らしさ・drug-likenessに類似した考え方といえるでしょう.
対象とする天然物はDNP (Dictionary of Natural Products)から取得し,対イオンなどのフラグメント部位の整理などを行っています.重要な点として,天然物に頻出する部分構造を取得するために,配糖体に関してはシュガー部位を除いたアグリコンの構造を分析対象としています.
得られた構造を一定のルールに従ってフラグメント化し,各フラグメントごとに天然物(NP)によく見られる部位と合成化合物(SM)によく見られる部位の重み付けを行うことでスコア化を行います.基本的な考え方としては,ある化合物を与えたときに「天然物」「合成物」を返す2クラス分類問題のような形です.
$$ \sum_{i=1}^{N} log\Bigl((NP数_i)/(SM数_i) \times (SM合計)/(NP合計)\Bigr) $$
NP-likenessスコアは-5から5の間をとり,大きい数字がより天然物である可能性が高いと言えます.
RDKitにおけるNP-likenessの実装
必要なスクリプトの入手
「RDKitで合成難易度を評価して化合物をスクリーニング」という記事で扱ったSAスコアと同様に,NP-likenessの計算もRDKitのコアモジュールには含まれていません.
GitHubのrdkit/Contrib/NP_Score/から必要なスクリプトを入手します.下の写真の青枠で囲まれている2つのファイルをダウンロードします.
スクリプト内のコメントに記載があるとおり,オープンソース化にともない天然物データは上述のDNPではなく,各種オープンソースデータベースから集めた約5万化合物を用いたようです.
必要な分子の準備
抗菌系の生理活性物質には天然物由来のものが多いと予想されますので,今回はZINC15データベースから取得したSDFを用いて計算を行っていきます.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, PandasTools
print(rdBase.rdkitVersion) ### 2018.09.1
df = PandasTools.LoadSDF('bacteria.sdf')
len(df) ### 450
450個の分子を含むpandasのデータフレームを用いて作業を行っていきたいと思います.
RDKitを用いて天然物らしさの評価
scoreMol関数は
- mol: 分子MOLオブジェクト
- fsscore: NP-likeness計算に用いるモデル
の2つを引数にとります.さきほどダウンロードした「publicnp.model」ファイルの中身がpythonのpickleで保存されたモデルになります.
下記のコードでは
- pickleファイルの読み込み
- NP-likenessスコアの計算
- ヒストグラムの作成
- スコアの最も大きい分子の取得
を順番に行っています.
import pickle
### 1) pickleの読み込み
with open('./publicnp.model', 'rb') as f:
fs = pickle.load(f)
### 2) NP-likenessの計算
import npscorer
fs_scores = [npscorer.scoreMol(m, fs) for m in df.ROMol]
df['np-likeness'] = fs_scores
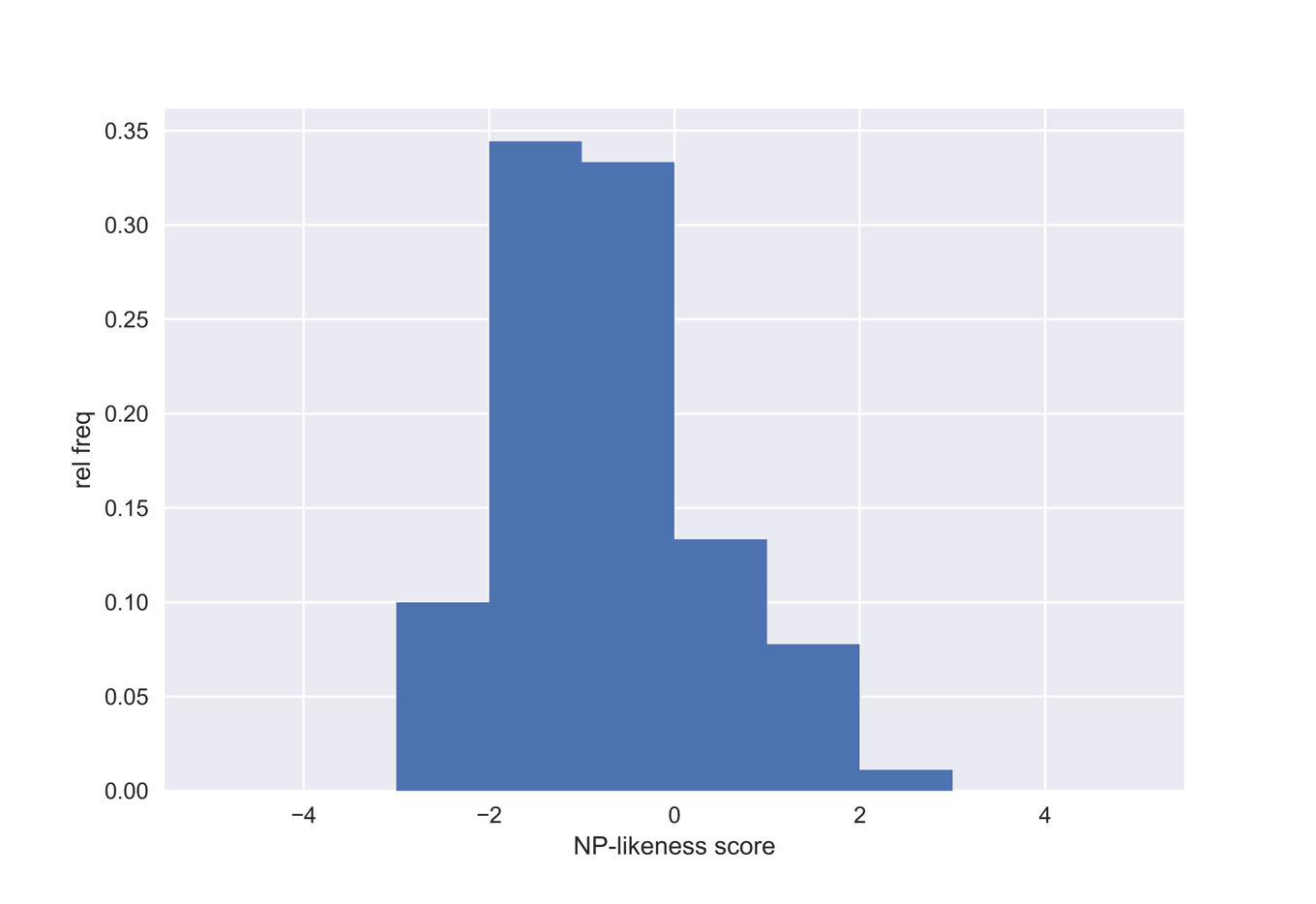
### 3) ヒストグラムの作成
with mpl.style.context('seaborn'):
plt.hist(fs_scores, density=True, bins=range(-5,6))
plt.xlabel('NP-likeness score')
plt.ylabel('rel freq')
### 4) スコアの最も大きい分子の取得
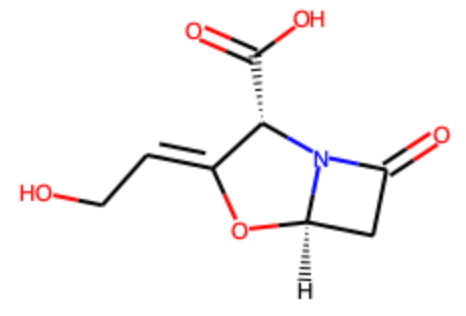
df.ROMol[df['np-likeness'].idxmax()]
今回の分子の中で最も天然物らしい化合物は下記のβーラクタムのようです.
取得した天然物らしい化合物の特徴を可視化
最後に計算したNP-likenessスコアを用いて簡単な分析を行ってみましょう.
上述したように,合成化合物と比べると天然物は
- 芳香環の数が少ない・sp3炭素が多い
- 脂溶性が高い
- 極性表面積が多い
といった特徴があることが知られています.そこでそれぞれを示す分子記述子として
- Fsp3
- MolLogP
- TPSA
を選択し,分子量も合わせて計算することで,NP-likenessスコアの正負によって違いがあるかを見てみましょう.
以下のコードでは,
- 各記述子をpandasのmap関数を使って一括計算
- NP-likenessスコアの正負で条件分け
- 2×2のグリッドに各記述子ごとのヒストグラムを作成
を行っています.
### 1. 記述子計算
from rdkit.Chem import Descriptors
df['MolLogP'] = df.ROMol.map(Descriptors.MolLogP)
df['MW'] = df.ROMol.map(Descriptors.MolWt)
df['TPSA'] = df.ROMol.map(Descriptors.TPSA)
df['Fsp3'] = df.ROMol.map(Descriptors.FractionCSP3)
### 2. スコアの正負で場合分け
df_np = df[df['np-likeness'] >= 0]
df_sm = df[df['np-likeness'] < 0]
len(df_np) ### 100
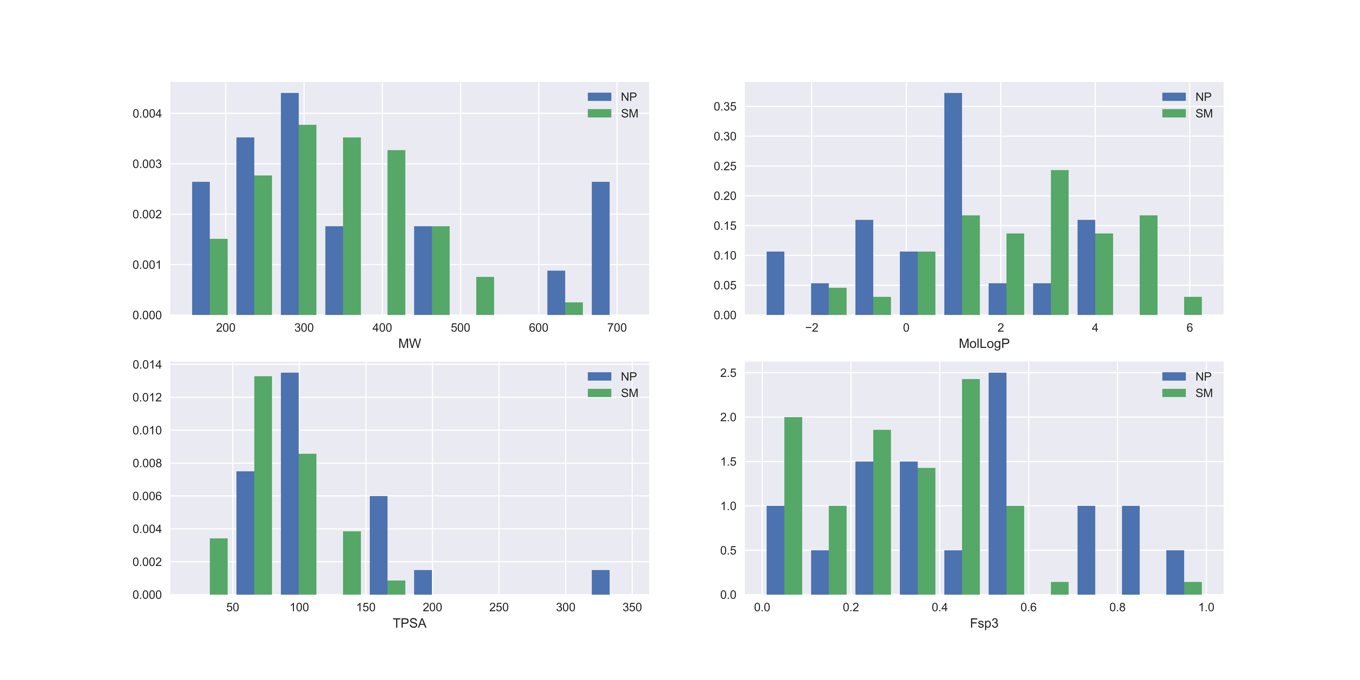
### 3. 記述子ごとにヒストグラムの作成
import itertools
descs = ['MW', 'MolLogP', 'TPSA', 'Fsp3']
with mpl.style.context('seaborn'):
fig, ax = plt.subplots(2,2, figsize=(16,8))
for (i,j), desc in zip(itertools.product(range(2), range(2)), descs):
ax[i,j].hist([df_np[desc], df_sm[desc]], density=True, label=['NP', 'SM'])
ax[i,j].set_xlabel(desc)
ax[i,j].legend(loc='best')
スコアが0以上の化合物は100個のようです.ヒストグラムからはLogPについて予想に反してNP群のほうが小さいことがわかります.一方で,NP群の方がTPSAは大きく,Fsp3も高い傾向がみてとれました.
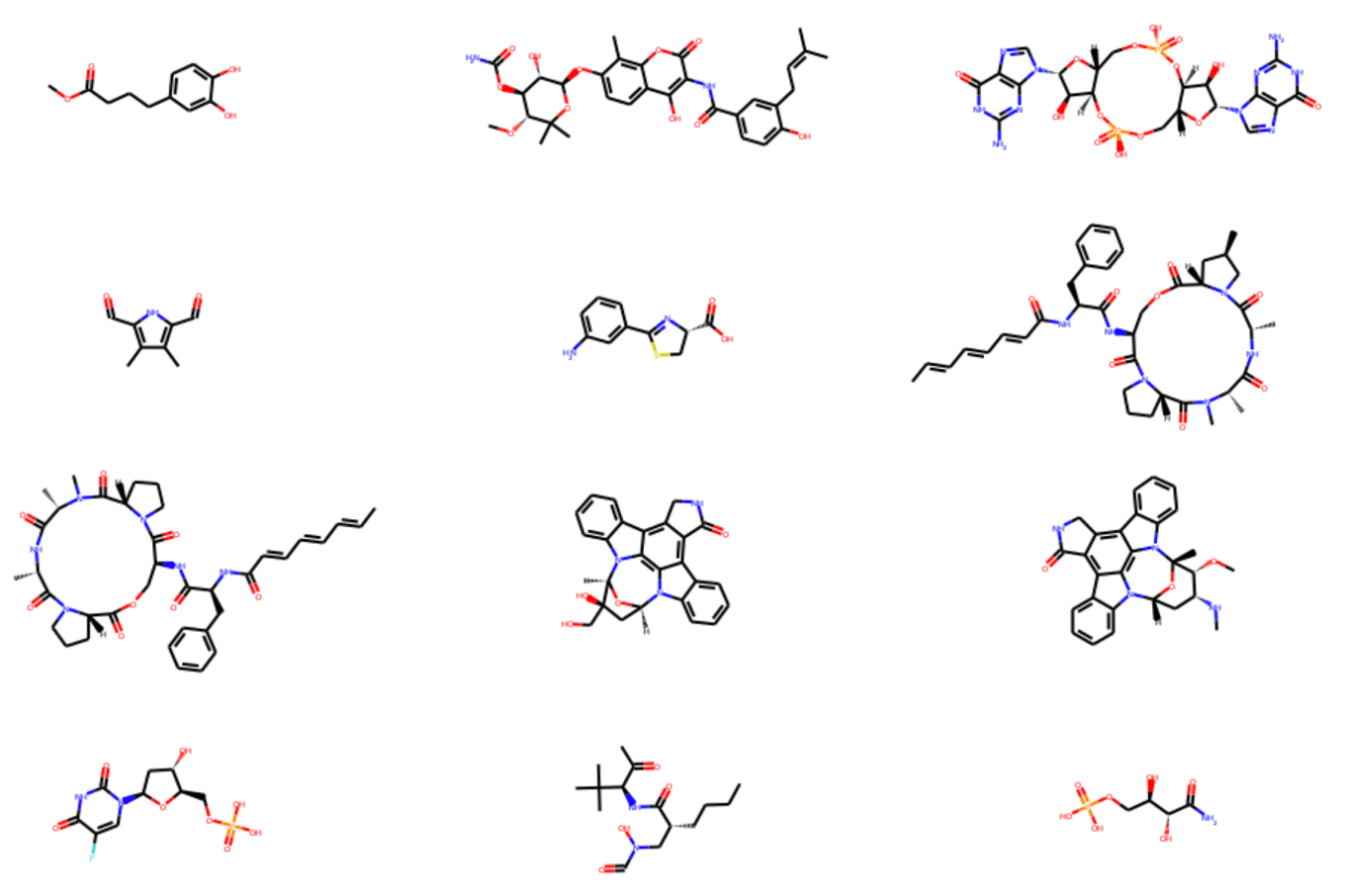
またスコアが正の天然物らしい化合物のうち,最初の12個は以下のような構造でした.環状ペプチドや複雑な縮環構造を有するいかにも天然物っぽい構造の中に,フルオロウラシル構造が混じっているのが見てとれます.
リン酸エステル構造の出現頻度が高いことが水溶性が高めになっている要因かもしれません.
df_np[df_np.ROMol >= Chem.MolFromSmiles('OP(O)(=O)O')]['MolLogP'].describe().round(2)
df_sm[df_sm.ROMol >= Chem.MolFromSmiles('OP(O)(=O)O')]['MolLogP'].describe().round(2)
count 15.00 mean -2.44 std 0.65 min -3.05 25% -3.05 50% -2.70 75% -1.57 max -1.57 Name: MolLogP, dtype: float64 count 5.0 mean 3.5 std 0.0 min 3.5 25% 3.5 50% 3.5 75% 3.5 max 3.5 Name: MolLogP, dtype: float64
実際に部分構造検索を行ってみたところ,リン酸構造を有する化合物はNP群では100分子中15分子存在し,その平均MolLogPは-2.44だとわかりました.一方でSM群では350分子中5分子だけで,平均MolLogPも3.5と極性の偏りが小さいようです.
終わりに
今回は「RDKitで天然物らしさをスコア化して化合物ライブラリーの指標にする」という話題について
- 天然物らしい化合物の特徴
- 天然物らしさをどのように評価するか
- RDKitにおけるNP-likenessスコアの実装
- NP-likenessと他の記述子との間の簡単な分析
という順番で解説していきました.
NP-likenessスコア単体ではあまり役に立ちませんが,一般的な記述子に加えて,天然物らしさを化合物ライブラリー構築の際に考慮することで,特徴的なライブラリーができあがるかもしれません.頭の片隅に入れておくとよい指標だと思います.
次回は「天然物らしさ」に変えて,「薬らしさ」をどのように定量するかという話題を扱ってみたいと思います.
>>次の記事:「RDKitで薬らしさを定量的に評価する」
コメント