ケモインフォマティクスでは考えられる化合物全ての集合を「ケミカルスペース」と呼びます.その数は実に1060以上にも及ぶと言われています.また化合物を何らかの特徴量をもとに投影したものをケミカルスペースと言うこともあります.
実験的にケミカルスペースの全てを網羅するのは無理のあるスケールですので,コンピュータの出番になります.つまり「ケミカルスペース」はケモインフォマティクスにおける格好の研究課題です.
例えば,ケミカルスペース中の化合物同士の「距離」を考えることで化合物の類似性を表すことができます.本ブログでもこれまで,化合物ライブラリー中から遠い(似ていない)化合物を選ぶための戦略についていくつかの記事で紹介してきました.
また生理活性を有する化合物に共通の部分構造が存在するという考えがあります.そのため広大なケミカルスペースを効率的に探索する方法として,既存構造を基にして生理活性化合物のバーチャルライブラリーを構築する戦略を紹介しました.
今回の記事では,そもそも「ある一定の重原子数を有する化合物はどれくらいの数存在するのか?」というケミカルスペースに対する本質的な問いに対する答えを追求するプロジェクトから生まれたGDB(Generated DataBase)について紹介します.
GDBはスイスのベルン大学のJean-Louis Reymondの研究グループによって行われているプロジェクトで,これまで
- 重原子の数が11以下のGDB-11
- 13以下のGDB-13
- 17以下のGDB-17
が報告されています.
よく用いられるデータセットはGDBデータベースを基にして作られていることから,ケモインフォマティクスにおいて非常に影響力の大きいプロジェクトと言えます.
“The Chemical Space Project” Acc. Chem. Res. 2015, 48, 722-730.
各種化合物データベースに収載されている化合物数
GDBについて触れる前に,既知のケミカルスペースがどの程度の大きさかを見てみましょう.主要な化合物データベースについて,2020年2月現在の収録化合物数を下のテーブルにまとめてみました.
| データベース | 収録化合物数 |
|---|---|
| PubChem | 102 M |
| ChemSpider | 81 M |
| ZINC15 | 230 M |
| Chembl | 1.9 M |
何れのデータベースについても,近年登録化合物数はすごいスピードで増えています.例えばPubChemには2017年11月では93.9 Mの化合物数でしたが2年程度で8 M個増えています.
それでもこれらの数はケミカルスペースの中のごくごく一部に過ぎません.
GDBデータベース作成方法
ここではGDBデータベースがどのように作成されているかを見ていきます.
概略としては
- 全部炭素のグラフを数え上げ
- 芳香族化などの処理をして同一化合物の除去
- 好ましくない構造を削除
- 炭素をへテロ原子に置換
- 一部の例外を除き,へテロ–へテロ結合を有するものを削除
などの処理を行っているようです.
化合物のグラフ表現を数え上げる
「MOLファイル・SDFとはどんな化学情報ファイルなのか?」という記事でも触れたように,化合物は原子をノード,結合をエッジと見ることで数学的なグラフと考えることができます.
例えば下の図は炭素が4個の分子の結合様式を表しています.それぞれ
- n-ブタン
- 2-メチルプロパン
を表現していることが容易にわかると思います.単結合のみのC4グラフとしては他に
- 4員環のシクロブタン
- 3員環+メチル基のメチルシクロプロパン
が考えられるでしょう.
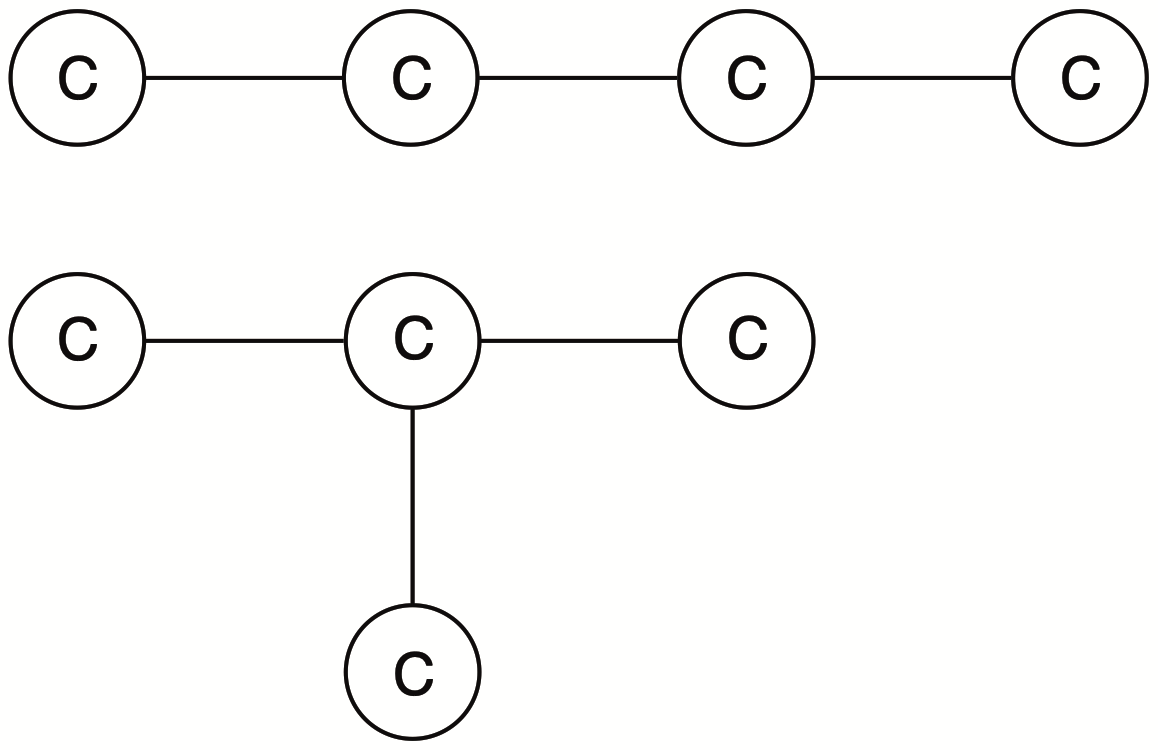
GDBプロジェクトではまず11個や13個などの一定数以下の炭素で構成されるグラフ構造を全て取得します.この際,興味があるのは有機化合物ですから最大結合数は4で固定しています.
炭素原子をヘテロ原子で置換
得られたグラフ構造をもとに,炭素をヘテロ原子(酸素,窒素,ハロゲン等)で置換していきます.この作業を系統的に実施することで,考えられる全ての組合せを取得します.
不安定な構造の除去
結合数を最大4に絞ることでオクテットルールに則った構造を生成することができましたが,化学者の目線からは好ましくない化合物が多く見られました.
そこで生成されたグラフから
- 多環性の3員環や4員環等を含む歪んだ構造
- 9員環未満の環状アルキンや橋頭位の二重結合
- 容易に加水分解されそうなエナミン,エノール,酸ハロゲン化物
- 酸化還元に活性な官能基
- オキシム等を除いたへテローへテロ原子結合
などの構造を有する化合物を除去していきました.
GDBデータベースの特徴
グラフの生成段階では多環状構造が大半を占めていましたが,生成されたデータベースに存在する分子としては単環または二環性の分子の割合が多くなりました.
| 生成されたグラフ | GDB中の分子 | |
|---|---|---|
| 非環状 | 0.5% | 5.8% |
| 単環性 | 4.3% | 25.9% |
| 二環性 | 16.9% | 37.3% |
| 三環性 | 34.6% | 22.8% |
| 多環性 | 43.8% | 8.1% |
<GDB-13の内訳:J. Am. Chem. Soc. 2009, 131, 8732. より改変>
またGDB化合物群はGDB-11(C,N,O,F)で26.4M個,GDB-13(C,N,O,Cl,S)で977M個になりました.分子量はそれぞれ153,180程度であり,大半の化合物のlogPは-2から2の間でした.
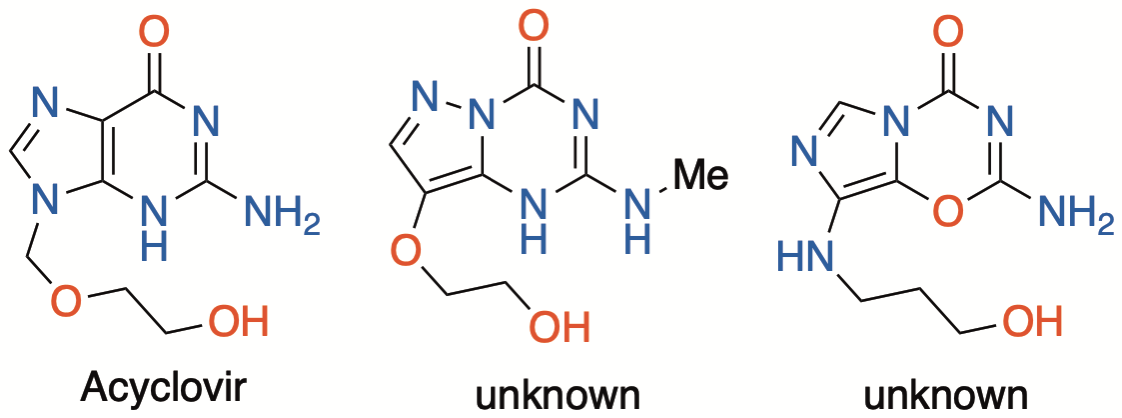
GDB化合物中には数多くの未知化合物が含まれています.例えば,既存医薬品と同一の分子式を有し,タニモト係数で類似度の高いながらも報告のない構造も数多く存在しました.
下記では論文記載の例として,抗ウイルス薬アシクロビルを示しています.
GDBデータベースのダウンロード
GDBデータベースはJean-Louis Reymondグループの該当ページからSMILESのリストとして入手可能です.
GDB-13のフルsmilesデータは圧縮後のファイルサイズで2.6GBと非常に巨大になっていますので,いくつかに分けたファイルや,ある項目に沿ってキュレーションされたデータセットも公開されています.
前述のようにGDB化合物は,分子量やlogPのからリード化合物やFBDDのフラグメントとして適していると考えられ,
- リード化合物向け
- メドケム向け
- FBDD向け
- 香料向け
のように各種用途別に選別されたデータセットがあります.
また不安定な構造のために除かれた化合物については「Dark Matter Universe」と呼ばれ,GDB-9相当の化合物の分子を集めたものが公開されています.
GDBデータセットの読み込み
最後に簡単にGDBデータセットの中身を見てみます.今回はGDB-13の中から炭素と酸素のみを含むデータセット(gdb13.co.tgz)を使います.
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw from rdkit.Chem.Draw import IPythonConsole import os import numpy as np import matplotlib as mpl import matplotlib.pyplot as plt import seaborn as sns sns.set() %matplotlib inline
tarファイルの中身は重原子の数ごとにファイルが分かれてsmilesが記載されています(全部で13個のファイル).今回は分子を読み込む際に5000以上の分子が存在するものについては,ランダムに5000個だけを読み込んでいます.その結果,全部で31869個の分子を読み込みました.
gdb13co = []
smiles_list = [str(i)+'.co.smi' for i in range(1,14)]
for file in smiles_list:
with open(os.path.join(os.getcwd(),'gdb13.co',file), 'r') as smiles:
sms = smiles.read().strip().split('\n')
if len(sms) > 5000:
index = np.random.choice(range(len(sms)), size=5000, replace=False)
for i in index:
sm = sms[i]
mol = Chem.MolFromSmiles(sm)
if mol:
gdb13co.append(mol)
else:
for sm in sms:
mol = Chem.MolFromSmiles(sm)
if mol:
gdb13co.append(mol)
print(len(gdb13co)) ## 31869
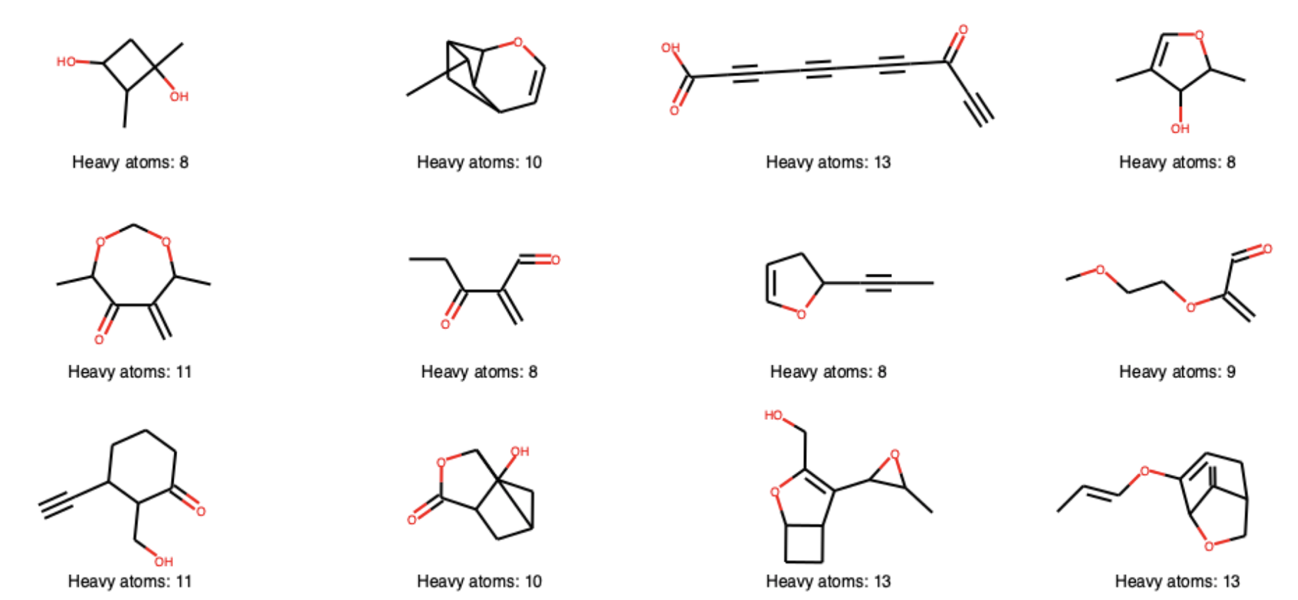
読み込んだ分子のうちいくつかを可視化してみます.
draw_mols = [gdb13co[i] for i in np.random.choice(range(len(gdb13co)), size=12, replace=False)]
Draw.MolsToGridImage(draw_mols, molsPerRow=4, subImgSize=(250,150),
legends=['Heavy atoms: '+str(mol.GetNumHeavyAtoms()) for mol in draw_mols])
炭素と酸素だけから成る分子でも多様な構造が考えられることが見てとれます.
最後に「RDKitで立体的な分子の形を記述する」という記事で説明したPMIプロットを作成してみます.
PMIプロットは分子の形をdisk状化合物(ベンゼン),rod状化合物(アセチレン),sphere状化合物(アダマンタン)の3軸で表現した際に,分子がどこに位置するかを可視化するものです.
既存の生理活性物質ではdiskからrod状エリアに多く分布することが知られており,より三次元的なsphere領域へ化合物を移行が望ましいとされています.
npr1 = []
npr2 = []
for m in gdb13co:
if m.GetNumHeavyAtoms() > 1:
m_h = Chem.AddHs(m)
AllChem.EmbedMolecule(m_h, AllChem.ETKDGv2())
m = Chem.RemoveHs(m_h)
if m.GetNumConformers() == 1:
r1 = AllChem.CalcNPR1(m)
r2 = AllChem.CalcNPR2(m)
npr1.append(r1)
npr2.append(r2)
一般的な化合物群よりも随分とsphere領域(右上)にまで広がっているのが見てとれます.

終わりに
今回は「ケミカルスペースプロジェクトとGDBデータベース」という話題について,
ある重原子の数が一定数以下の化合物を網羅的に含んだGDBデータベースについて紹介しました.
GDBが
- グラフ構造を基に炭素からへテロ原子への置換を系統的に行うことで生成されていること
- 未知化合物を大量に含むデータベースであること
などについて解説しました.
次回はGDB中の化合物を基に,量子化学計算を行ったQM-9というデータセットを紹介したいと思います.QM-9は機械学習プロジェクトにおいて非常によく使われるデータセットになります.
>>次の記事:「QM9は量子化学計算に基づいた機械学習用の大規模データセット」

コメント