これまでに非常に多くの化合物が単離または合成されており,その化学的性質に関する膨大な知見が蓄積されています.これら化合物の情報を一カ所に集めたものが化学データベースになります.今回とりあげるPubChemもそういったデータベースのうちの1つです.
この記事ではpythonを使ってPubChemにアクセスし化合物情報を取得する方法を解説していきます.
化学データベースの種類
化学物質に特化して作られたデータベースとして最も有名なものはアメリカ化学会が管理するCASだと思いますが,他にも化合物の性質・用途に応じた様々なデータベースが存在しています.下のテーブルに代表的なものをいくつかまとめます.
| データベース | 説明 |
|---|---|
| CAS | アメリカ化学会が運営.非常に巨大 |
| PubChem | アメリカNIH傘下のNCBIが運営するデータベース |
| ChemSpider | 英国化学会が所有する化合物データベース |
| ZINC15 | 主にバーチャルスクリーニング用途に市販化合物を集めたデータベース |
| ChEMBL | 文献情報から化学構造と生物活性データを集めたデータベース |
| PDB | 主に生体高分子の固相構造を集めたデータベース |
| CCDC | 主に低分子の固相構造を集めたデータベース |
| SDBS | 有機化学者にお馴染みのスペクトルデータを集めた国産のデータベース |
なかでもPubChemとChemSpiderは収載項目も多く,APIによる自動取得が可能なことから使いやすいデータベースと言えます.
PubChemとは
PubChemはアメリカのNIH傘下のNCBIによって2004年に管理・運営が開始された化合物データベースで,原子数1000以下かつ1000結合以下の比較的小さな分子が収録されています.PubChemのデータベースは次の3つに分けられていて,ウェブ上から自由に使うことが可能です.
| データベース | 説明 |
|---|---|
| Compound | 完全に単一の化合物が収録されているデータベースです.2017年11月時点で9390万エントリーが登録されています |
| Substance | Compoundとは異なり,抽出物などの混合物が収録されています.2017年11月時点で2360万エントリーが登録されています |
| BioAssay | このデータベースでは名前の通り,生物アッセイの結果が収録されています.2017年11月時点での登録数は125万エントリーが登録されています |
PubChemPyとは
このPubChemデータベースにpythonからアクセスするためのライブラリがPubChemPyです.上述の3つのデータベースにアクセスする方法と,取得したエントリーから物性などの様々な性質を取り出す方法が実装されています.注意点としては,
- 常にインターネットアクセスが必要
- 検索クエリーはPubChem側で処理されるため機密情報を含んだ構造などでは扱いにくい
といった点が挙げられます.
インストール
PubChemPyの公式ドキュメントではpipの利用が推奨されています.特に依存関係はなく,対応するpythonのバージョンは2.7, 3.5, 3.6のようです.
pip install pubchempy
なお以下のコードではpubchempyをpcpとしてimportしていることを前提とします.
import pubchempy as pcp
Compoundデータベースからの検索
Compoundデータベースからは
- 化合物名
- 分子式
- SMILES
- InChI
- SDF
- CID(Compound ID)
を用いて検索が可能です.よく使うのは化合物名とSMILESだと思います.
化合物の検索・取得
このメソッドでは指定した検索結果をリストで返します.下記の例では’alanine’という名前で検索し,その結果をCIDとIUPAC名を表示しています.この場合はラセミ,D体,L体の3種類がヒットしています.
aa = pcp.get_compounds('alanine', 'name')
for i in aa:
print('CID: {}\tName: {}'.format(i.cid, i.iupac_name))
CID: 602 Name: 2-aminopropanoic acid CID: 5950 Name: (2S)-2-aminopropanoic acid CID: 71080 Name: (2R)-2-aminopropanoic acid
上記の例のように,検索で取得したCompoundクラスにはcidやiupac_nameをはじめとして様々なレコードがあります.レコードの中には立体情報に関連したものもありますが,デフォルトの検索設定では2D情報しか取得してくれません.3D情報も取得したい場合にはrecord_type=’3d’を設定してあげる必要があります.
先の検索で5950がL-alanineのcidだとわかりましたので,以下のコードではCIDによる検索を行いつつ3次元情報も取取得しています.
l_ala_3d = pcp.get_compounds(5950, 'cid', record_type='3d') l_ala_3d = l_ala_3d[0] l_ala_3d.mmff94_energy_3d, l_ala_3d.volume_3d # (1.5212, 68.8)
化合物を検索して直接物性値などを取得
欲しいレコードが決まっている場合は,Compoundクラスを丸ごと取得してからレコードにアクセスするのではなく,検索段階で望みのレコードだけを一気に持ってくることも可能です.大量のデータを処理したい場合などには,この方が速度面で有利になります.
今回は先ほど見たalanineの検索に対して,
- IUPAC名
- 分子式
- 分子量
- XlogP
- tPSA
- カノニカルSMILES
の6つの情報を取得してみます.欲しい情報はリストとして渡します.
properties = ['IUPACName', 'MolecularFormula', 'MolecularWeight', 'XLogP', 'TPSA', 'CanonicalSMILES'] a = pcp.get_properties(properties, 'alanine', 'name')
得られた結果は下のようにヒット化合物ごとにレコードを記録した辞書のリストになります.
[{'CID': 602,
'MolecularFormula': 'C3H7NO2',
'MolecularWeight': 89.094,
'CanonicalSMILES': 'CC(C(=O)O)N',
'IUPACName': '2-aminopropanoic acid',
'XLogP': -3,
'TPSA': 63.3},
{'CID': 5950,
'MolecularFormula': 'C3H7NO2',
'MolecularWeight': 89.094,
'CanonicalSMILES': 'CC(C(=O)O)N',
'IUPACName': '(2S)-2-aminopropanoic acid',
'XLogP': -3,
'TPSA': 63.3},
{'CID': 71080,
'MolecularFormula': 'C3H7NO2',
'MolecularWeight': 89.094,
'CanonicalSMILES': 'CC(C(=O)O)N',
'IUPACName': '(2R)-2-aminopropanoic acid',
'XLogP': -3,
'TPSA': 63.3}]
pandasデータフレームでの出力
取得したデータを辞書形式ではなくpandasのデータフレームとして取得できるとその後のデータ処理が捗ることが多いです.
- get_compounds()
- get_properties()
- get_substances()
の3つのメソッドではas_dataframe=Trueオプションを指定することで,Compoundクラスのリストなどとしてではなく,pandasのデータフレームとして結果を取得できます.こちらの方がその後の分析がやりやすくなると思いますのでおすすめです.
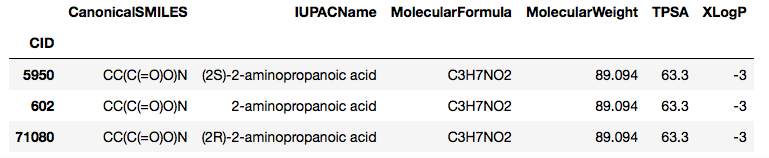
例えば上記の’alanine’の例では下記のようなデータフレームに格納されます.
a = pcp.get_properties(properties, 'alanine', 'name', as_dataframe=True)
具体例:アミノ酸誘導体の一括検索
以前にpandasを用いてウェブからテーブルを読み込むスクレイピング方法を紹介しましたが,今回はこの方法を用いて以下の操作を行ってみます.
- Wikipediaのアミノ酸のページから3文字表記のリストを取得
- 各アミノ酸のN末にBoc基を付加した構造をPubChemで検索(例:Boc-Ala-OH)
- いくつかのレコードを指定してget_properties()を用いて検索を行い,得られた結果をpandasのdataframe形式で保存
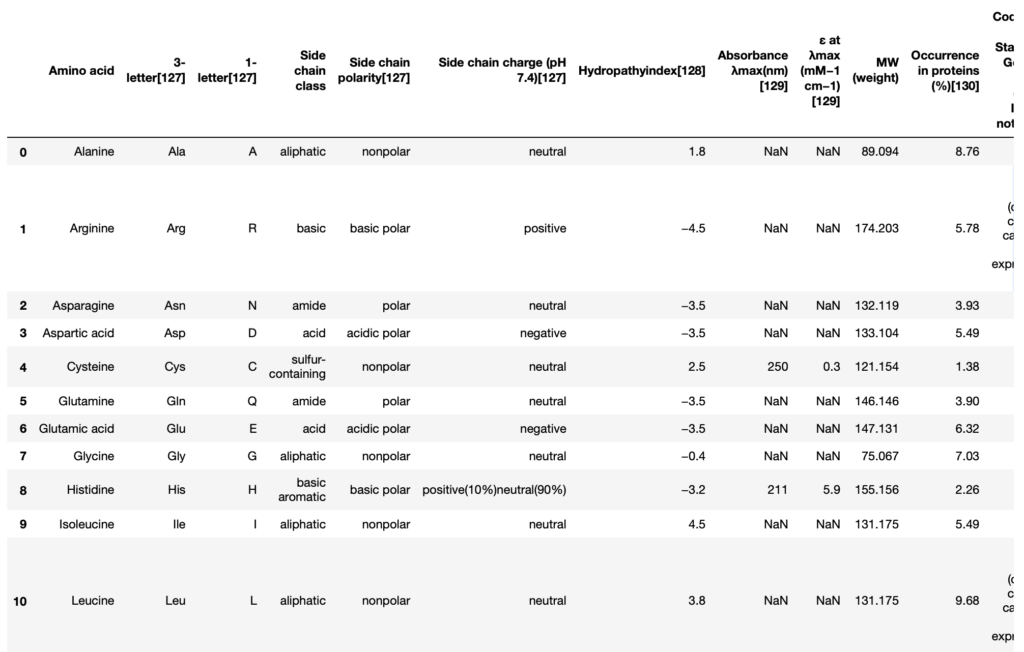
# Wikipediaからテーブルを取得して整形
dfs = pd.read_html('https://en.wikipedia.org/wiki/Amino_acid')
df = dfs[2]
names = df['3-letter[127]']
この時点のdfは以下のようです.
# 得られたリストを使ってPubChemからプロパティを取得し,データフレーム型に加工
def Boc_AA_OH(name_list):
properties = ['iupacname', 'molecularformula', 'molecularweight', 'xlogp', 'tpsa', 'canonicalsmiles']
return_list = []
for n in name_list:
AA = 'Boc-' + n + '-OH'
x = pcp.get_properties(properties, AA, 'name')
if len(x) == 1:
return_list.append(x[0])
else:
return_list.append('NA')
print('{} was not retrived properly.')
return return_list
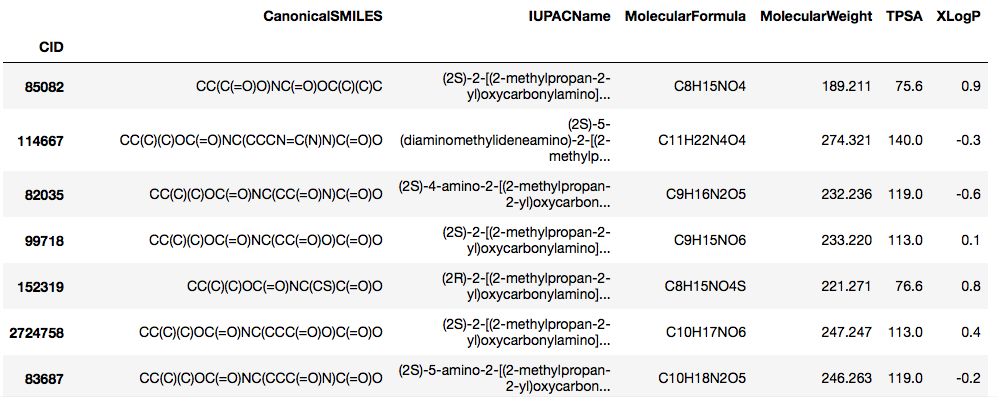
y = Boc_AA_OH(names)
aa_df = pd.DataFrame(y)
aa_df.index = aa_df['CID']
aa_df = aa_df.drop('CID', axis=1)
得られたデータフレームは以下のようなものになりました.
Substanceデータベースからの検索
基本的にはCompoundデータベースと使い方は同じです.cidの代わりにsidを使います.混合物のデータベースですので,例えば’alanine’という名前で検索しても以下のように大量にヒットしてきます.
b = pcp.get_substances('alanine', 'name')
len(b)
b[:10]
155 [Substance(4590), Substance(125505), Substance(585842), Substance(841591), Substance(6436532), Substance(7885836), Substance(8143558), Substance(10527484), Substance(11528441), Substance(11528607)]
終わりに
今回のエントリーではpythonを使って化学情報を扱う例として,pubchempyを用いたPubChemからの化合物情報取得の自動化を行いました.実際には今回のような情報の取得だけで作業が完結することはありません.
次回以降ではそもそもどのように化合物をコンピュータ上で表現するかから始めます.pythonのケモインフォマティクス用ライブラリRDKitを用いて分析を行っていきましょう.
>>次の記事:「RDKitでケモインフォマティクスに入門」

コメント
具体例:アミノ酸誘導体の一括検索
実行したところ
KeyError: ‘3-letter[136]’
というようなエラーメッセージが出てきて
うまく実行できませんでした。
対処はどのようにすればいいでしょうか?
コメントありがとうございます.
執筆時からWikipediaのページが編集されたようで,記載のコードではエラーが出るようになっていました.
新しく直しておきましたので,そちらを使ってみてください.