これまで化合物の変異原性データや溶解度データを用いて,いくつかの機械学習アルゴリズムでモデルを構築してきました.これらのモデルは分子の構造・特徴を何らかの形で入力情報として与えることで,変異原性の有無や溶解度の値が出力される「教師あり学習」モデルでした.
その際,分子の入力情報としては構造から得られるフィンガープリントを用いてきました.すなわち0か1の値から構成される疎なベクトルとして分子を表現してきました.
分子の入力情報の表現方法としては,例えばリピンスキールールのように「分子量」「脂溶性(CLogP)」「水素結合受容体・供与体の数」を基にして入力ベクトルを作成することも可能です.今回はこういった「分子記述子」をもとに機械学習モデルを作成する場合の注意点とデータの前処理について学んでいきます.
「ESOL: Estimating Aqueous Solubility Directly from Molecular Structure」
J. Chem. Inf. Comput. Sci. 2004, 44, 1000–1005.
(DOI: 10.1021/ci034243x)
ESOLの論文で用いられている記述子
データセットの引用元であるEstimated SOLubility(ESOL)の論文では,
- clogP
- 分子量:MW
- 回転可能結合数:RB
- 芳香族指数:AP
の4つの記述子を用いて重回帰分析を行うことで
$$ Log(S_w) = 0.16 – 0.63clogP – 0.0062MW + 0.066RB -0.74AP $$
という式を得ています.
今回はclogPをRDKitのMolLogPで代用することとし,同様に4つの記述子を用いてモデル構築を行っていきます.その際に記述子間のスケールの違いが精度に与える影響を見ていくことにします.
必要なライブラリのインポートと分子の準備
まずは必要なライブラリのインポートと分子の読み込みを行い,その後必要な記述子の計算を行いましょう.
以下のコードでは
- ライブラリのインポート
- pandasのデータフレームとして情報を読み込む
- 分子MOLオブジェクトの構築
- 各記述子の計算
という順番で作業を行っています.
# 1. ライブラリのインポート
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
import pandas as pd
import bumpy as np
import matplotlib as mpl
import matplotlib.pyplot as plt
import seaborn as sns
print(rdBase.rdkitVersion) # 2018.09.1
mpl.style.use('seaborn')
# 2, 3. pandasデータフレームとして読み込み
df = pd.read_csv('./ci034243xsi20040112_053635.txt')
PandasTools.AddMoleculeColumnToFrame(frame=df, smilesCol='SMILES')
# 4. 記述子の計算
df['logP'] = df.ROMol.map(Descriptors.MolLogP)
df['MW'] = df.ROMol.map(Descriptors.MolWt)
df['RB'] = df.ROMol.map(Descriptors.NumRotatableBonds)
df['AP'] = df.ROMol.map(lambda x: len(x.GetAromaticAtoms())/x.GetNumHeavyAtoms())
data = df[['logP', 'MW', 'RB', 'AP']]
targets = df['measured log(solubility:mol/L)']
data.shape, targets.shape # ((1144, 4), (1144,))
続いて「線形モデルを用いた化合物の溶解度予測:通常最小二乗法,Ridge回帰,Lasso回帰」で見たように,線形モデルを用いてモデルを構築してみましょう.
from sklearn.linear_model import LinearRegression from sklearn.model_selection import train_test_split X_train, X_test, y_train, y_test = train_test_split(data, targets, random_state=0) X_train.shape, X_test.shape, y_train.shape, y_test.shape # ((858, 4), (286, 4), (858,), (286,)) lr = LinearRegression().fit(X_train, y_train) lr.intercept_, lr.coef_
(0.2109051991025006, array([-0.72000131, -0.00681906, 0.01908224, -0.37619135]))
係数と切片の値を用いると,scikit-learnの線形モデルで得られた式は
$$ Log(S’_w) = 0.21 – 0.72clogP – 0.0068MW + 0.019RB -0.38AP $$
という形になります.
論文では実際には今回以上のデータを用いてモデルを構築していますが,各記述子の係数の桁が同じですのでESOLの論文に近いモデルが得られたと考えてよいでしょう.
特徴量のスケールの重要性
「RDKitとscikit-learnで機械学習:変異原性をk-最近傍法で予測」という記事でk-最近傍法を説明したときに,下記の図を用いました.
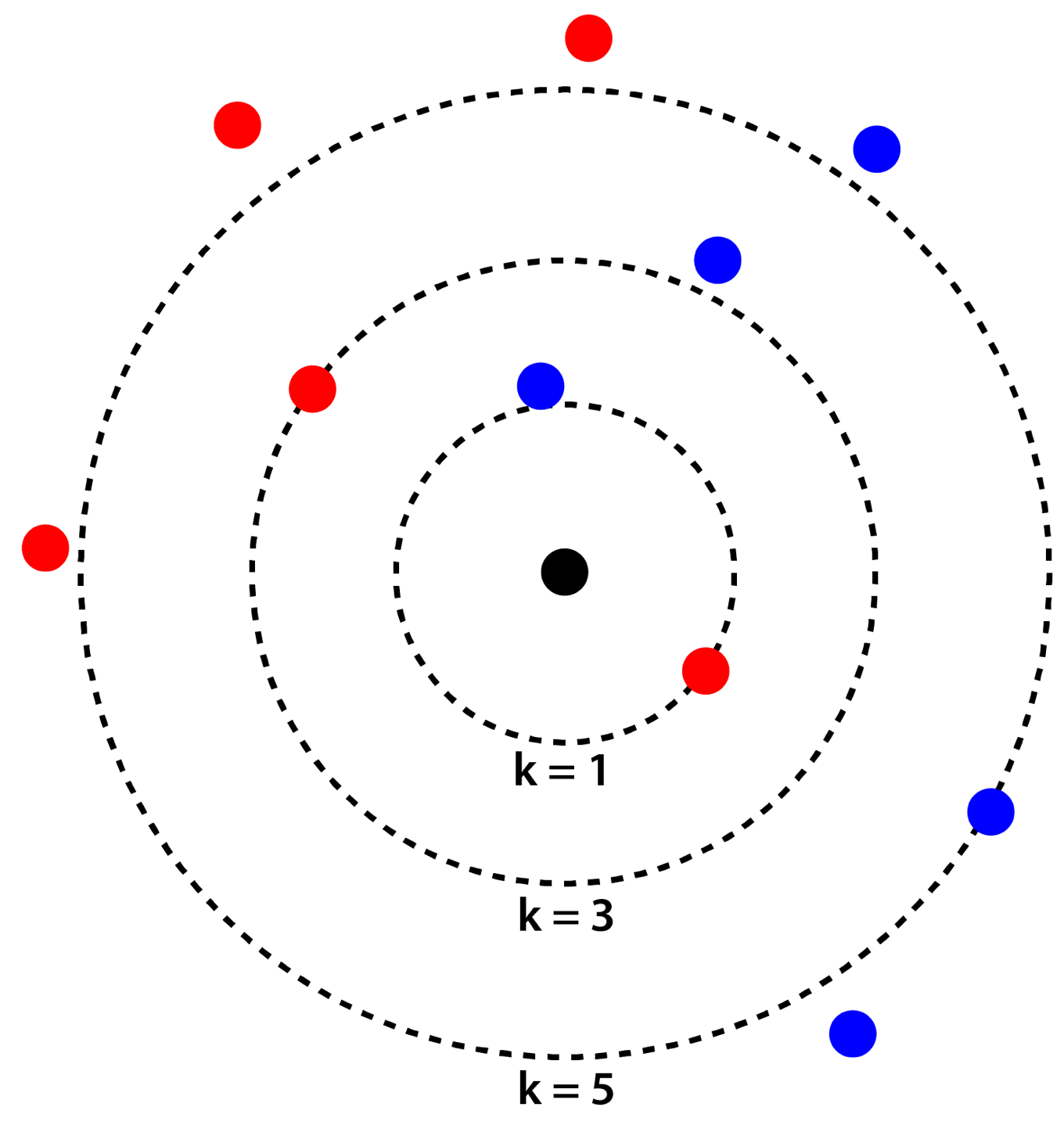
k-最近傍法では「距離」に着目して判断しますので,各特徴量が同じ縮尺であることが期待されます.試しに上の図の縦軸方面を縮小してみると下のようになります.
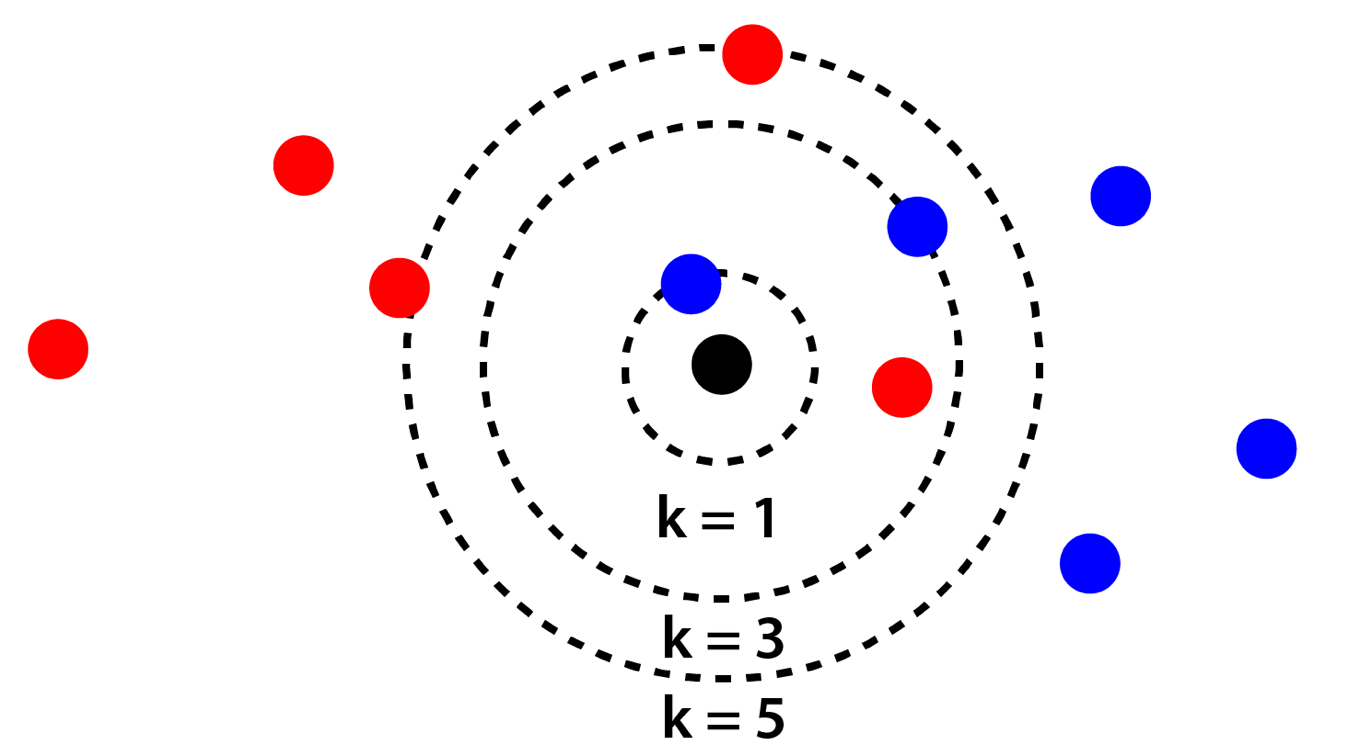
この場合,スケールの大きい横軸方面の情報はほとんど使われずに,判断がされることになります.このように機械学習アルゴリズムには特徴量のスケールに敏感なものが存在します.
そこで特徴量のスケールを変換する前処理を行ってからモデルを構築することが行われます.例えば
- データを平均0,分散1に揃える
- データが0から1の間に入るようにする
といった処理です.こういった前処理は頻繁に行われますのでscikit-learnにも自動で行ってくれるクラスが実装されています.
scikit-learnを用いた前処理と注意点
sklearn.preprocessing.MinMaxScaler()
scikit-learnでは前処理はpreprocessingに実装されています.それぞれ
- StandardScalerはデータを平均0,分散1になるように変換
- MinMaxScalerはデータが0から1の間に入るように変換
といった処理を行います.fitメソッドでデータ変換方法の学習,transformメソッドで実際にデータ変換を行います.前処理を行う際に重要な点は,データの平均などを求めるにあたってテスト用データを使わないことです.これをやってしまうと,テスト用データの漏洩がおきてしまいます.
下のコードではk-最近傍法を用いて,
- 前処理を行わないままモデルを構築
- MinMaxScalerで処理をした後でモデル構築
を行い,精度を比較しています.fitメソッドは訓練用データに対して行っていることを確認してください.
from sklearn.preprocessing import MinMaxScaler
from sklearn.model_selection import train_test_split
from sklearn.neighbors import KNeighborsRegressor
knn = KNeighborsRegressor().fit(X_train, y_train)
print('knn train score: {:.3f}'.format(knn.score(X_train, y_train)))
print('knn test score: {:.3f}'.format(knn.score(X_test, y_test)))
scalar = MinMaxScaler().fit(X_train)
X_train_scaled = scalar.transform(X_train)
X_test_scaled = scalar.transform(X_test)
knn_scl = KNeighborsRegressor().fit(X_train_scaled, y_train)
print('knn_scl train score: {:.3f}'.format(knn_scl.score(X_train_scaled, y_train)))
print('knn_scl test score: {:.3f}'.format(knn_scl.score(X_test_scaled, y_test)))
予想通りスケールを変換した方が精度が高くなりました.
knn train score: 0.770 knn test score: 0.636 knn_scl train score: 0.896 knn_scl test score: 0.845
交差検証と前処理の注意点
先ほど,前処理にあたっては「テスト用データを利用しないこと」が重要と述べました.これは交差検証においてもあてはまります.すなわち交差検証にあたっては「検証用データを使わずに前処理を行う」必要があります.
つまり上で行ったようにデータの前処理を行った上で,交差検証を行うと以下のような状態になってしまい望ましくありません.

こういった検証用データの漏洩を防ぐためには,交差検証ループの中で検証用データを分けた後でスケール変換を行う必要があります.この作業を自動化するための仕組みがscikit-learnには実装されています.それがPipelineと呼ばれるものです.

Pipelineは「前処理」「モデル構築」といった複数の作業を1つにまとめる役割を果たしてくれます.そしてPipelineオブジェクトを用いて交差検証を行うことで,上図のように検証用データを除いて前処理を行った上で,モデル構築を行うことができます.
Pipelineでは処理を行いたい順番に,(処理の名前,オブジェクト)というタプルのリストを引数とします.下のコードはk-最近傍法を用いてパイプラインと交差検証を組み合わせて用いています.「scaler」と「knn」がPipelineに渡す処理の名前になります.
from sklearn.pipeline import Pipeline
from sklearn.model_selection import cross_val_score
pipe = Pipeline([('scaler', MinMaxScaler()), ('knn', KNeighborsRegressor())])
cv_scores = cross_val_score(pipe, X_train, y_train, cv=10)
print(cv_scores)
[0.78535774 0.78718731 0.85627787 0.83554423 0.7968093 0.85275787 0.89584956 0.82153032 0.82850489 0.80480031]
具体例:サポートベクターマシンとグリッドサーチ
最後にもう1つ具体例として,サポートベクターマシンと呼ばれる機械学習アルゴリズムを用いてみます.k-最近傍法と同様に,サポートベクターマシンも特徴量のスケールに敏感であることが知られています.このアルゴリズムはCとgammaという2つのハイパーパラメーターを持っていますので,これらの値を,「交差検証を用いてElastic Netを化合物の溶解度データに対して最適化」という記事で見たようにグリッドサーチを用いて最適化してみることにします.
なおサポートベクターマシンは,データ数数千から1万以下程度の比較的小さなデータセットに対してよく用いられる強力なアルゴリズムで,回帰・分類の両方に用いることができます.scikit-learnではsklearn.svmに実装されています.
グリッドサーチでパイプラインの利用
下のコードではCの値を6つ,gammaの値を6つスクリーニングします.その際に渡すパラメータグリッドの設定方法はPipelineの名前に「__」(アンダースコアを2つ)を付けて設定します.得られた結果はヒートマップとして可視化しています.「C=10, gamma=10」のモデルが最良の結果を与えたようです.
from sklearn.pipeline import Pipeline
pipe = Pipeline([('scaler', MinMaxScaler()), ('svm', SVR())])
param_grid = {'svm__C': [0.001, 0.01, 0.1, 1, 10, 100],
'svm__gamma': [0.001, 0.01, 0.1, 1, 10, 100]}
grid = GridSearchCV(pipe, param_grid=param_grid, cv=10)
grid.fit(X_train, y_train)
result = pd.DataFrame(grid.cv_results_)
scores = np.array(result.mean_test_score).reshape(6,6)
fig = plt.figure()
ax = fig.add_subplot(111)
mat = ax.matshow(scores, cmap='Reds', alpha=0.8, vmin=0)
fig.colorbar(mat)
ax.set_xticks(range(6))
ax.set_xticklabels([0.001, 0.01, 0.1, 1, 10, 100])
ax.set_xlabel('gamma')
ax.set_yticks(range(6))
ax.set_yticklabels([0.001, 0.01, 0.1, 1, 10, 100])
ax.set_ylabel('C')

最後にテスト用データの精度も確認してみましょう.
print('train score: {:.3f}'.format(grid.score(X_train, y_train)))
print('test score: {:.3f}'.format(grid.score(X_test, y_test)))
train score: 0.892 test score: 0.872
終わりに
今回は「データ分析と前処理:パイプライン処理で化合物の溶解度を推定」という話題について,
- 前処理の重要性と種類
- 前処理を行う際の注意点
- scikit-learnのPiplineクラスを用いた交差検証時の前処理方法
などについて学びました.最後にはサポートベクターマシンという機械学習アルゴリズムを用いてハイパーパラメーターのチューニングを実施することで,学んだ内容の復習を行いました.
これまでscikit-learnのscoreメソッドを用いてモデルの善し悪しを評価してきました.回帰においてR2値は標準的な評価方法ですが,分類の場合は正答率は必ずしも適切でない場合があります.
例えば毒性試験では,陰性を陽性と判断する「偽陽性」と,陽性を陰性と判断してしまう「偽陰性」では,後者の方がプロジェクトにおけるダメージが大きいです.そのため,後者のペナルティが大きくなるような評価基準の方が好ましいと言えます.次回はこういった例のように,作成した機械学習モデルを評価する様々な基準について学んでいきたいと思います.
>>次の記事:「機械学習モデルの評価方法:化合物の変異原性の有無を題材に」

コメント