「RDKitでケモインフォマティクスに入門」という記事では,RDKitの分子であるMolオブジェクトの作成方法を中心に学習しました.今回はそのMolオブジェクトに対してどのような操作が行えるかを学んでいきます.
化学は分子を中心に扱う学問ですので,Molオブジェクトの扱いに習熟することは大変重要です.
SDFの入手と必要なライブラリのインポート
まずは必要なライブラリをimportし,分子を読み込んでおきましょう.SDFとしては前回と同じく,fluorochemから入手したサリチル酸誘導体を使います.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
print('rdkit version: {}'.format(rdBase.rdkitVersion))
# rdkit version: 2020.03.3
suppl = Chem.SDMolSupplier('./salicylate_jan_2021.sdf')
mols = [mol for mol in suppl if mol is not None]
print(len(mols))
# 431
Molオブジェクトの構成要素
Molオブジェクトは分子ですので,当然原子から構成されています.また分子は原子と原子の間に結合が形成されることによりできあがります.こういった化学者の感覚通りに,RDKitのMolオブジェクトにも
- Atomオブジェクト
- Bondオブジェクト
などが用意されています.
AtomオブジェクトとBondオブジェクトは互いにリンクしており,
- Atomオブジェクトはどの結合を形成しているか
- Bondオブジェクトはどの原子から結合が形成されているか
といった情報が相互に記録されています.
Atomオブジェクト
Mol.GetAtomWithIdx(idx)
Atomオブジェクトには
- 全原子をまとめて取得
- 原子のインデックスを使って選択的にアクセス
することが可能です.
このようにして得られたAtomオブジェクトには,
| 原子の情報 | メソッド |
|---|---|
| インデックス | atom.GetIdx() |
| 原子番号 | atom.GetAtomicNum() |
| 元素記号 | atom.GetSymbol() |
| 質量 | atom.GetMass() |
| 混成 | atom.GetHybridization() |
| 環内原子か否か | atom.IsInRing() |
| 芳香族か否か | atom.GetIsAromatic() |
| 結合リスト | atom.GetBonds() |
などのように,様々な要素を取得するメソッドが用意されています.
ここでは431分子のうち最初の3分子について,sp3混成の炭素原子のインデックスを表示してみることにします.注意点としてはGetHybridization()で得られる結果はChem.rdchem.HybridizationType.SP3というオブジェクトなので,このままでは’SP3’という文字列とは比較できません.そこでstringオブジェクトに変換する作業が必要になります.
for mol in mols[:3]:
print(mol.GetProp('Name'))
for atom in mol.GetAtoms():
if atom.GetSymbol() == 'C' and str(atom.GetHybridization()) == 'SP3':
print('index for sp3 carbon: {}'.format(atom.GetIdx()))
print('###')
Methyl 2,5-bis(trifluoroethoxy)benzoate index for sp3 carbon: 10 index for sp3 carbon: 12 index for sp3 carbon: 13 index for sp3 carbon: 14 index for sp3 carbon: 18 ### Methyl 2-(trifluoromethoxy)benzoate index for sp3 carbon: 9 index for sp3 carbon: 11 ### 2-Phenoxybenzoic acid ethyl ester index for sp3 carbon: 13 index for sp3 carbon: 16 ###
Bondオブジェクト
Mol.GetBondWithIdx(idx)
Mol.GetBondBetweenAtoms()
Bondオブジェクトについても,
- 全結合をまとめて取得
- 結合のインデックスを使ってアクセス
によって取得が可能です.
また結合を形成する2つの原子のインデックスがわかっている場合には,その結合を直接指定することも可能です.
Bondオブジェクトについても下表のように
| 結合の情報 | メソッド |
|---|---|
| 結合のインデックス | bond.GetIdx() |
| 結合の種類 | bond.GetBondType() |
| 芳香族結合か否か | bond.GetIsAromatic() |
| 環内結合か否か | bond.IsInRing() |
| 始点原子を取得 | bond.GetBeginAtom() |
| 始点原子をIdで取得 | bond.GetBeginAtomIdx() |
| 終点原子を取得 | bond.GetEndAtom() |
| 終点原子をIdで取得 | bond.GetEndAtomIdx() |
様々な要素に対して対応するメソッドが設定されており,これらの情報が容易に取得可能になっています.
ここでは431分子の最初の2分子について,芳香族結合を構成する原子のインデックスを出力してみます.下のコードでは,
- bond.GetIsAromaticで芳香族結合か否かを判断
- 結合始点と終点の原子をGetBeginAtom, GetEndAtomで取得
- 元素記号とインデックスを取得
という手順で処理をしています.
for mol in mols[:2]:
print(mol.GetProp('Name'))
for bond in mol.GetBonds():
if bond.GetIsAromatic():
atom_a = bond.GetBeginAtom()
atom_b = bond.GetEndAtom()
print('bond between {}{}-{}{} is aromatic.'.
format(atom_a.GetSymbol(), atom_a.GetIdx(),
atom_b.GetSymbol(), atom_b.GetIdx()))
print('---')
どちらの分子もベンゼン環を1つ有するのみですので,芳香族結合は6本ずつです.
Methyl 2,5-bis(trifluoroethoxy)benzoate bond between C0-C1 is aromatic. bond between C0-C2 is aromatic. bond between C1-C4 is aromatic. bond between C2-C6 is aromatic. bond between C4-C9 is aromatic. bond between C6-C9 is aromatic. --- Methyl 2-(trifluoromethoxy)benzoate bond between C0-C1 is aromatic. bond between C0-C2 is aromatic. bond between C1-C5 is aromatic. bond between C2-C6 is aromatic. bond between C5-C10 is aromatic. bond between C6-C10 is aromatic. ---
RingInfoオブジェクト
環状構造は分子において特別な部分構造ですので,RDkitでは環構造情報に特化したRingInfoオブジェクトが存在しています.
ここでは以下に示す多環性スピロ化合物を例にRingInfoオブジェクトについて見ていきたいと思います.
spiro = Chem.MolFromSmiles('C1CCC12CCCC23CC3')
spiro_ring = spiro.GetRingInfo()
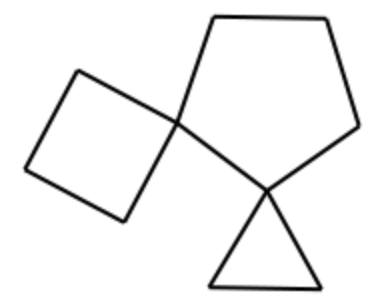
この分子のAtomとBondのインデックスは以下のようになっています.黒数字がAtom,青数字がBondオブジェクトのインデックスに対応しています.
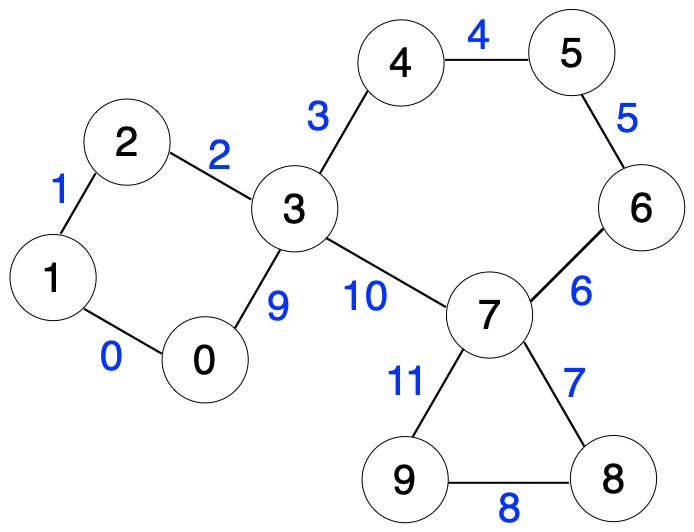
環を構成するAtomやBondの情報
RingInfo.AtomRings()
RingInfo.BondRings()
分子内に存在する環構造について,それぞれ構成するAtomやBondの集合をタプルのタプルで返します.
print(spiro_ring.AtomRings()) print(spiro_ring.BondRings())
上で示した番号と一致しています.
((0, 3, 2, 1), (4, 3, 7, 6, 5), (8, 7, 9)) ((9, 2, 1, 0), (3, 10, 6, 5, 4), (7, 11, 8))
環サイズや環数に関する情報
IsAtomInRingOfSize(atomIdx, ringSize)
MinAtomRingSize(atomIdx)
それぞれ
- 原子が何個の環に含まれているか
- 原子が指定サイズを環を形成しているか
- 原子を含む最小の環サイズ
について返すメソッドになります.
同様にBondオブジェクトに関しても対応するメソッドが存在しています.
print(spiro_ring.NumAtomRings(3)) # 2 print(spiro_ring.IsAtomInRingOfSize(0,4)) # True print(spiro_ring.MinAtomRingSize(3)) # 4
Molオブジェクトのプロパティ
SetProp(name, value)
GetPropNames()
Molオブジェクトにはプロパティと呼ばれる任意の値を付加可能です.
- プロパティの設定にはSetProp
- プロパティの参照にはGetProp
を使います.
もし元のSDFに最初からプロパティが設定してある場合にはオブジェクト作成と同時にそれらの値も設定されています.先ほどのコードでもNameというプロパティを参照している箇所がありました.これはMolオブジェクト作成に利用したSDFに最初から設定されていた値になります.
既にMolオブジェクトにどのようなプロパティが存在するかを知りたい場合にはGetPropNamesで取得可能です.
他に特別なプロパティとしてSDFのタイトル行に相当する_Nameというものもあります.こちらはマジックプロパティと呼ばれる隠れプロパティ扱いになっており,GetPropNamesでは取得できません.
下のコードでは設定されているプロパティとその値の一覧を表示してます.
for prop in mols[0].GetPropNames():
print('{}: {}'.format(prop, mols[0].GetProp(prop)))
Code: 006795 Name: Methyl 2,5-bis(trifluoroethoxy)benzoate CAS: 35480-31-0

分子の2次元構造描画
今回取り扱うの分子は全て原子の座標を持っていますが,もしも初期設定されていないようでしたら,以前見たようにAllChem.Compute2DCoordinateで分子を描画する前に計算しておく必要があります.
a. Chem.Draw.MolToImage(mol)
b. Chem.Draw.MolToFile(mol, file_name)
2) 複数分子の描画
a. Chem.Draw.MolsToImage(mols)
b. Chem.Draw.MolsToGridImage(mols, molsPerRow, subImgSize, legends)
メソッド名やオプションが色々とあってややこしいですが,まず出力先として,
- ファイルに保存したい場合にはMolToFile
- Jupyter Notebookにそのまま打ち出したい場合にはMolToImage
を使います.
また描画したい分子の数が複数の場合には,Molsと複数形にします.
一番利用頻度が多いのがMolsToGridImageだと思います.このメソッドはオプション指定で,
| オプション名 | 内容 |
|---|---|
| molsPerRow | 一列あたりに描画する分子の数 |
| subImgSize | 分子1つの大きの設定 |
| legends | 分子毎に凡例をつける |
など,細かく設定可能になっています.以下のコードで具体例を見てみましょう.
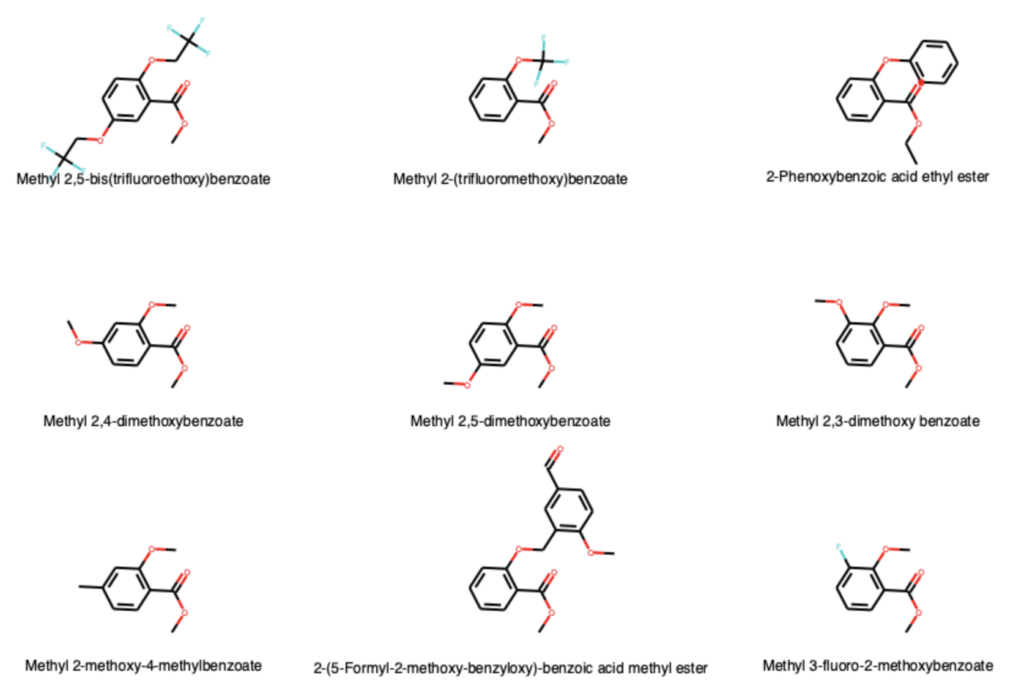
# 複数分子の描画(凡例付き)
Draw.MolsToGridImage(mols[:9], molsPerRow=3, subImgSize=(300,200),
legends=[x.GetProp('Name') for x in mols[:9]])

テンプレート構造を用いた構造の整列
先ほどのグリッドイメージでは化合物の並び方が一致していないため,類似構造がぱっと見ではわかりにくいです.そこでサリチル酸をテンプレートとして分子を回転させてみます.
その用途には,Chem.AllChemにあるGenerateDepictionMatching2DStructureを使います.テンプレートのMolオブジェクトを作成して2次元座標の計算を行うだけで整列が行われます.
# テンプレート作成
import pubchempy as pcp
tmp = pcp.get_compounds('salicylic acid', 'name')
tmp = tmp[0]
tmp_smiles = tmp.canonical_smiles
template = Chem.MolFromSmiles(tmp_smiles)
AllChem.Compute2DCoords(template)
# テンプレートをもとに2次元座標の再計算
for mol in mols:
if mol.HasSubstructMatch(template):
AllChem.GenerateDepictionMatching2DStructure(mol, template)
# グリッドイメージで再び描画
Draw.MolsToGridImage(mols[:9], molsPerRow=3, subImgSize=(300,200),
legends=[x.GetProp('Name') for x in mols[:9]])
今回のケースではもともとの描画の方が美しく,整列後3個目のフェノキシエーテルなどは構造が重なってしまっていますが,作業のポイントはわかっていただけると思います.
部分構造の取り扱い
先ほどのテンプレートを用いた描画でさりげなく使いましたが,Molオブジェクトには部分構造を引数に取るメソッドが用意されています.例えば部分構造を有するか否かを調べることで,分子のフィルタリングに利用可能です.
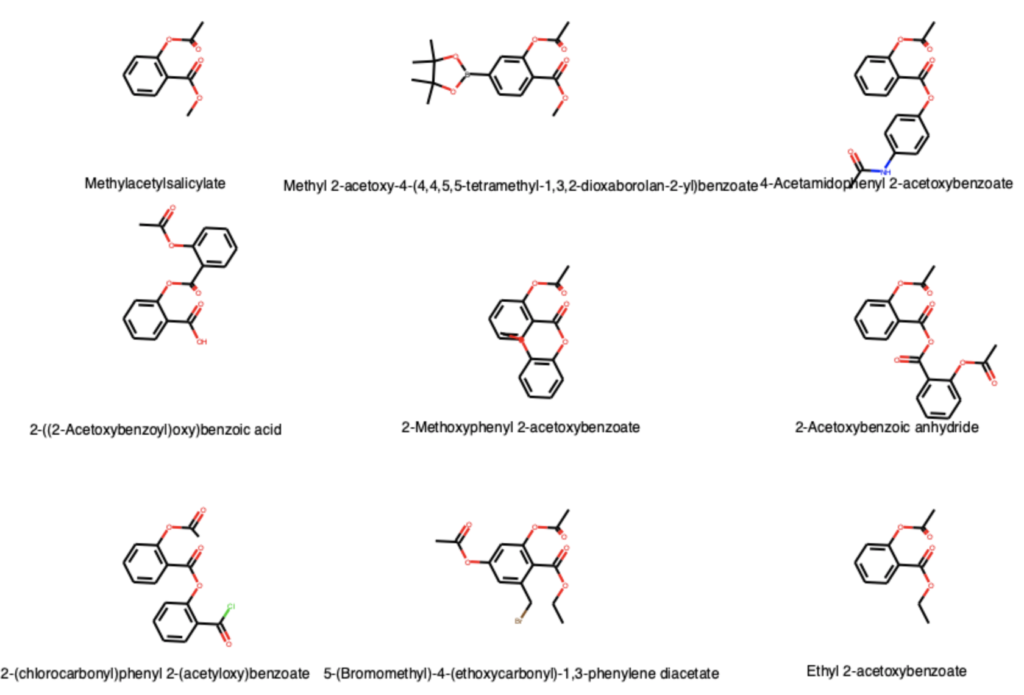
ここでは例としてアスピリン(アセチルサリチル酸)を部分構造に持つ分子の数を調べ,描画してみます.431個の分子うち,11個の分子のフェノールがアシル化されているとわかりました.
# テンプレート作成
aspirin = pcp.get_compounds('aspirin', 'name')
aspirin = aspirin[0]
aspirin_sm = aspirin.canonical_smiles
aspirin_mol = Chem.MolFromSmiles(aspirin_sm)
AllChem.Compute2DCoords(aspirin_mol)
# 部分構造を有する分子の描画
match = []
for mol in mols:
if mol.HasSubstructMatch(aspirin_mol):
match.append(mol)
print(len(match)) # 11
Draw.MolsToGridImage(match[:9], molsPerRow=3, subImgSize=(300,200),
legends=[x.GetProp('Name') for x in match])
終わりに
「RDKitでケモインフォマティクスに入門」ではRDKitにおける分子の表現方法であるMolオブジェクトの作成方法をいろいろと扱いました.
今回は「RDKitの分子Molオブジェクトを扱う」という話題について,
- 分子を構成する原子や結合に関する情報を取得する方法
- 分子内の環構造に関する情報を有するRingInfoオブジェクトの扱い方
- 分子の2次元表記について構造の描画方法や部分構造の扱い方
などを学びました.
次回からは分子を2次元から3次元へと展開していくことで,コンフォマーの生成や分子力学法を用いた構造最適化・エネルギー計算といった内容を扱います.
ここまでを学びきることができれば記述子を使ったケモインフォマティクスにおける予測モデルの作成から,Gaussianを始めとした外部の量子化学計算プログラムとの連携までいろいろな可能性が広がります.是非頑張っていきましょう.
>>>次の記事:「RDKitによる3次元構造の生成」
コメント
「Atomオブジェクト」の節 (#Atom) で (i) の黄色枠に Mol.GetAtomWithIndex(idx) としておりますが Mol.GetAtomWithIdx(idx) の誤りではないでしょうか、確認お願いいたします。
S様
ご指摘ありがとうございます.修正させて頂きました.