「RDKitによる3次元構造の生成」という記事でJupyterノートブック上で化合物の3次元構造を表示するにはpy3Dmolというライブラリを利用するのが広く使われるようになってきていると述べました.実際にpythonのケモインフォマティクス用ライブラリであるrdkitのIPythonConsoleモジュールでも立体構造の表示の際には内部でpy3Dmolをimportして使っていることも説明しました.
今回はpy3Dmolに関して,そもそもどういったライブラリなのかの説明から始めて,詳しい使い方を解説していきたいと思います.是非とも使い方をマスターして美しい立体構造を堪能してください.
3Dmol.jsはバイオインフォマティクス用のjavascriptライブラリ
3Dmol.jsとはピッツバーグ大学のDavid Koesのグループが作成しているjavascriptのライブラリです.Bioinformaticsの研究グループですので,特にタンパク質の立体構造の描画を意識して設計されたライブラリになりますが,低分子の描画にも十分使えます.ライセンスはBSDです.
例としてグルタチオンを3Dmol.jsを使って表示すると下のボックスのようになります.(表示されていない場合はページをリロードしてみてください.)
py3DmolはJupyter上で3Dmol.jsを使うためのpythonライブラリ
今回取り上げるpy3Dmolは,Jupyter上で3Dmol.jsを動かすウィジェットになります.全ての機能が使えるわけではありませんが,APIの多くは共通していますので,3Dmol.jsの公式サイトを読み込むのが勉強には一番よいと思います.
py3Dmolの特長として,一度分子を読み込んでしまえば,IPythonカーネルが動いていなくても描画が可能なことがあげられます.そのためノートブックをipynbファイルとして人と共有しても,相手側で構造を動かすことが可能になります.
py3Dmolのインストール
インストールはcondaまたはpipを使って可能です.
conda install -c conda-forge py3dmol
または
pip install py3Dmol
もしもノートブックではなく,JupyterLabで使いたい場合にはextensionのインストールが必要になります.
jupyter labextension install jupyterlab_3dmol
py3Dmolの基本的な使い方
py3Dmolは以下の4段階で構造を描画します.
- py3Dmol.viewオブジェクトの作成
- viewオブジェクトに描画したい分子を追加
- 描画書式の設定
- 表示
「RDKitでの構造式描画を詳しく解説」という記事で扱った2次元構造式を描画するrdChemDraw2Dモジュールに似ています.
描画領域の作成
まずは描画領域を作成する必要があります.ここで作成されるviewオブジェクトは3Dmol.jsの$3Dmol.GLViewerオブジェクトになりますので,公式のAPIを読むことでどんな機能が実装されているかを知ることができます.
デフォルトでは横640 x 縦480の描画領域が作成されます.またviewergridオプションを与えることでグリッドを作成することが可能です.その他のオプションについては下にまとめます.
| オプション | 説明 |
|---|---|
| width | 描画領域の横幅.デフォルトは640 |
| height | 描画領域の縦幅.デフォルトは480 |
| viewergrid | (m,n)で縦m,横nのグリッドを作成 |
| linked | グリッド間で分子の動きをシンクロさせるか否か.デフォルトはTrue |
| query | データベースから化合物をダウンロードして描画領域に設定 |
データベースからの化合物取得
query=’cid:pubchem-id‘
queryオプションを用いるとPDBとPubChemから化合物情報を取得できます.CCDCも試してみましたが対応していないようです.通常ダウンロードでもメールアドレスの入力が必要ですし,難しそうですね.
描画する分子の追加
作成したviewオブジェクトに分子を追加するにはaddModelを用います.入力形式としては
- pdb
- sdf (mol)
- xyz
- mol2
と代表的な形式はサポートされています.RDKitを用いる場合にはMolオブジェクトをChem.MolToMolBlockメソッドを用いてモルブロック形式へと変更してあげる必要があります.
その他のオプションもいくつか取ることができますが,もっとも使うのはkeepHだと思います.文字通り,水素原子を保持するか否かです.デフォルトはTrueになっています.なおpy3Dmolでのオプション設定はほぼ全て辞書型で指定する必要があります.今回の例であれば,
view.addModel(Chem.MolToMolBlock(mol), 'sdf', {'keepH': False})
のように設定します.
背景色の設定
描画領域の色をHex(16進数)コード,または色の名前で指定します.デフォルトでは白に設定されています.また2つ目引数に透過度を取ることができます.
指定原子へのズームイン
分子の中心へカメラがズームインします.本来はどの原子へと近づくかなど,細かく設定が可能ですがプレゼンテーションで使うわけではない限り必要ないと思います.
描画スタイルの設定
指定した部位の描画形式のスタイルを設定します.
- 針金(line)モデル
- 棒(stick)モデル
- 空間充填(CPK)モデル
といった一般的なものから,タンパク質の描画に便利なマンガ(cartoon)モデルなどが選択可能です.珍しいものとしてはクロス(cross)モデルという星条のオブジェクトで原子を表現するモデルが選択可能です.CPKと同様に分子の大きさを把握するのに適しています.
| style | 説明 |
|---|---|
| line | 針金モデル |
| cross | クロスモデル |
| stick | 棒モデル |
| sphere | 空間充填モデル |
| cartoon | 2次構造をマンガで表示 |
スタイルの詳しい設定方法は後ほど具体例で見ていきましょう.
モデルの表示
view.render()
どちらもviewオブジェクトの中身をJupyter上に表示します.showの方がよく使われているように見受けられます.
py3Dmolを用いた描画例
これから具体例を通していくつかのオプション設定を説明していきます.
例1:構造データのダウンロードと作成したグリッドへの描画
この例では
- PubChemからアラニンのデータを取得
- 横3 x 縦1のグリッドにアラニンを表示
- グリッド毎に描画スタイルを別々に設定
- グリッド毎に背景の色を設定
- 分子の表示
- 描画したviewオブジェクトをpng形式で固定
という一連の作業を行っていきます.
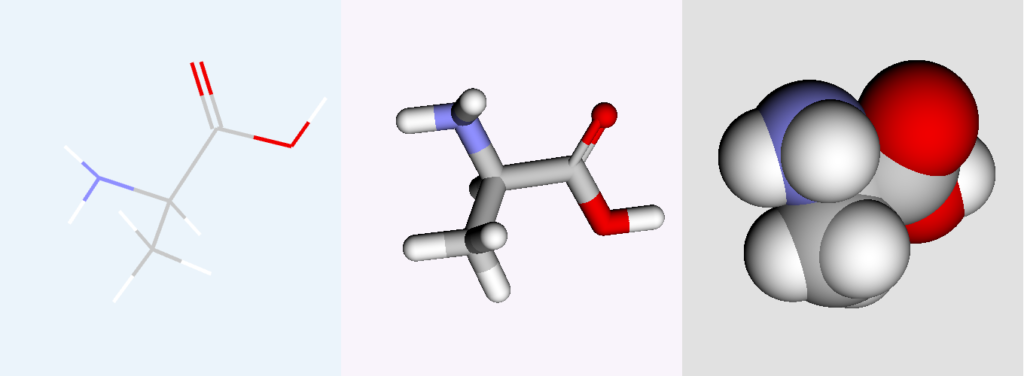
PubChemから構造をダウンロードし,グリッドを作成
アラニンの化合物ID(cid)は5950ですので,query=’cid:5950′を設定します.するとPubChemから立体構造が取得され,自動的に作成したviewオブジェクトに読み込まれます.
さらにグリッドをviewergrid=(1,3)で設定しています.queryで取り込んだ構造が全てのグリッドに反映されます.
これからviewオブジェクトに対して行う分子の追加・描画スタイル・背景の設定などは全てviewer=(m,n)によってどのグリッドに対して設定を行うかを明示することになります.指定しない場合は全てのグリッドに反映されます.
針金モデルのスタイル設定
針金モデルでは以下の設定が可能です.今回はlinewidthを用いて線の太さを太めに設定しています.
| 設定 | 説明 |
|---|---|
| hidden | モデルを非表示にするかどうか.デフォルトはFalse |
| linewidth | 線の太さ |
| colorscheme | ColorschemeSpec型による要素毎のカラー設定 |
| color | ColorSpec型による単一カラー設定.colorschemeと重なった場合は上書き |
棒モデルのスタイル設定
棒モデルでは以下の設定が可能です.今回のように何も設定しない場合にも空の辞書型を渡す必要があります.
| 設定 | 説明 |
|---|---|
| hidden | モデルを非表示にするかどうか.デフォルトはFalse |
| radius | 原子の半径 |
| singleBonds | 全ての結合を単結合として表記するか否か.デフォルトはFalse |
| colorscheme | ColorschemeSpec型による要素毎のカラー設定 |
| color | ColorSpec型による単一カラー設定.colorschemeと重なった場合は上書き |
空間充填モデルのスタイル設定
空間充填モデルでは以下の設定が可能です.今回のように何も設定しない場合にも空の辞書型を渡す必要があります.
| 設定 | 説明 |
|---|---|
| hidden | モデルを非表示にするかどうか.デフォルトはFalse |
| radius | ファンデルワールス半径の設定を書き換える |
| scale | ファンデルワールス半径を設定比率で拡大・縮小 |
| colorscheme | ColorschemeSpec型による要素毎のカラー設定 |
| color | ColorSpec型による単一カラー設定.colorschemeと重なった場合は上書き |
png画像として出力
残念ながらpy3Dmolにはmatplotlibのsavefigメソッドのように,描画した内容をpythonで扱えるメソッドは存在しません.しかし全ての設定を終えてノートブック上に表示したviewオブジェクトのスナップショットを撮るメソッドが実装されています.
まずは分子を好みの大きさ,角度で表示し,view.png()メソッドを呼び出すと,ノートブック上に静止画としてpngファイルが表示されます.それを右クリックで保存することで形式的にpy3Dmolの描画内容を画像として書き出すことができます.
コード例
以上の内容を反映したコードは下記の通りです.
view = py3Dmol.view(width=680, height=250, query='cid:5950', viewergrid=(1,3), linked=False)
view.setStyle({'line': {'linewidth': 5}}, viewer=(0,0))
view.setStyle({'stick': {}}, viewer=(0,1))
view.setStyle({'sphere': {}}, viewer=(0,2))
view.setBackgroundColor('#ebf4fb', viewer=(0,0))
view.setBackgroundColor('#f9f4fb', viewer=(0,1))
view.setBackgroundColor('#e1e1e1', viewer=(0,2))
view.show()
view.png()
例2:分子の部分選択と色の設定
この例では分子の一部を選択し,部分ごとに色を設定していきます.具体的には
- PDBからインスリンのデータを取得し,横2 x 縦2のグリッドに配置
- 分子の部分毎に描画スタイルや色を変更
- 各グリッドにsurfaceオブジェクトを追加
- 分子の表示
という作業を行います.
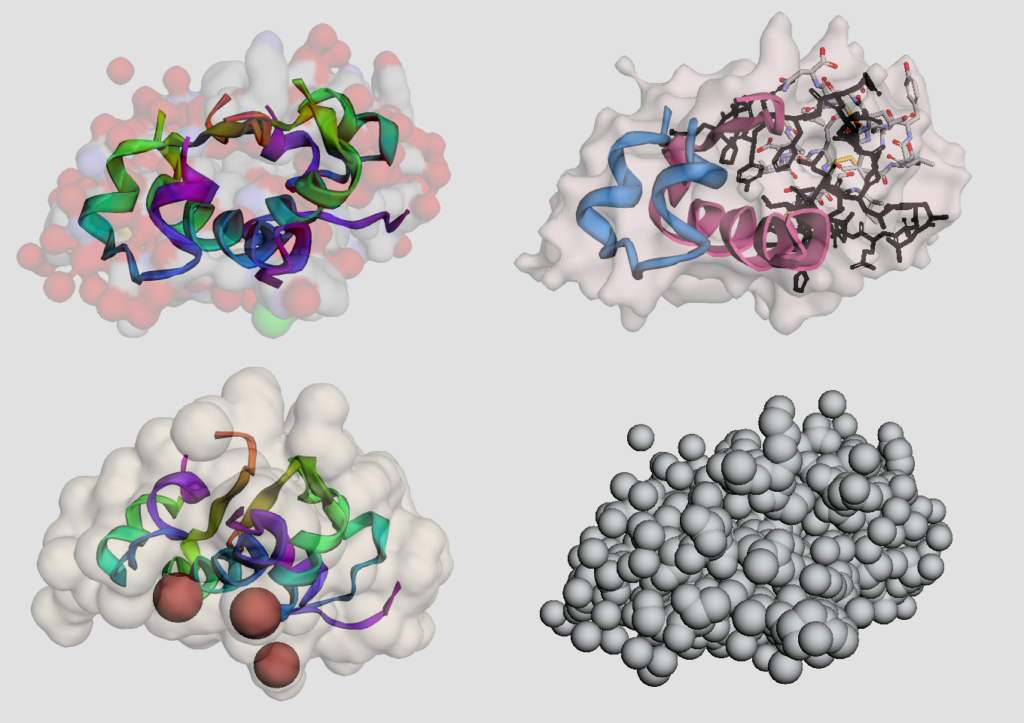
分子の一部の選択
py3Dmolでは色々な方法によって部分構造を選択可能です.ここでは一部だけを取り上げますので,詳細な情報公式APIの「AtomSelectionSpec」を参照してください.
今回の例では,chainを用いてA鎖やB鎖などのチェーン番号を,またelemを用いて亜鉛を元素記号で選択しています.選択した部分構造に対して,それぞれ別の描画設定を施しています.
| 設定 | 説明 |
|---|---|
| resn | 残基の名前Parent residue name |
| elem | 元素記号(例:’H’,’Ca’) |
| chain | PDBに設定されているチェーン番号(例:’A’) |
| resi | 残基番号 |
マンガ(cartoon)モデルのスタイル設定
マンガ(cartoon)モデルでは以下の設定が可能です.
| 設定 | 説明 |
|---|---|
| color | ColorSpec型によるストランドのカラー設定.’spectrum’は残基番号を基準としたグラディエント |
| style | スタイル設定.trace, oval, rectangle(デフォルト), parabola, edgedの5種類 |
| ribbon | リボンを幅一定に描画するか否か |
| arrows | βシートの方向を示す矢印を表示するか否か |
| tubes | αヘリックスを円柱で描画するか否か |
| thickness | ストランドの太さ.デフォルトは0.4 |
| opacity | 透過度 |
分子表面(サーフェス)の追加
溶媒接触表面(SAS)などの分子表面(サーフェス)を追加します.サーフェスの種類は$3Dmol.SurfaceTypeで指定し,
- VDW(ファンデルワールス表面)
- MS
- SAS
- SES(溶媒排除表面・Connolly表面)
の4種類が選択可能です.今回はVDW,SES,SASを描画しています.
サーフェスのスタイルは透過度や色が指定できます.
| 設定 | 説明 |
|---|---|
| opacity | 透過度 |
| colorscheme | ColorschemeSpec型による要素毎のカラー設定 |
| color | ColorSpec型による単一カラー設定.colorschemeと重なった場合は上書き |
コード例
以上の内容を反映したコードは下記の通りです.
view = py3Dmol.view(width=680, height=480, query='pdb:1zni', viewergrid=(2,2), linked=False)
view.setStyle({'cartoon': {'color': 'spectrum'}}, viewer=(0,0))
view.setStyle({'chain': 'A'}, {'cartoon': {'color': '#4aa6ff', 'style': 'oval'}}, viewer=(0,1))
view.setStyle({'chain': 'B'}, {'cartoon': {'color': '#f65fab', 'style': 'edged'}}, viewer=(0,1))
view.setStyle({'chain': 'C'}, {'stick': {}}, viewer=(0,1))
view.setStyle({'chain': 'D'}, {'stick': {'color': 'black'}}, viewer=(0,1))
view.setStyle({'cartoon': {'color': 'spectrum'}}, viewer=(1,0))
view.setStyle({'elem': 'Zn'}, {'sphere': {'scale': 2.0}}, viewer=(1,0))
view.setStyle({'sphere': {'color': '#CCD1D1'}}, viewer=(1,1))
view.addSurface(py3Dmol.VDW, {'opacity': 0.5}, viewer=(0,0))
view.addSurface(py3Dmol.SES, {'opacity': 0.6, 'color': '#FADBD8'}, viewer=(0,1))
view.addSurface(py3Dmol.SAS, {'opacity': 0.6, 'color': '#FDEBD0'}, viewer=(1,0))
view.setBackgroundColor('#e1e1e1')
view.show()
例3:グリッド毎に異なるカラースキームの設定
この例では低分子をグリッドに表示していき,それぞれのグリッドで描画に用いるカラーパレットを変更していきます.具体的には
- 表示する分子の立体構造を準備
- グリッドを作成し,そこに分子を追加
- グリッドに異なるカラーパレットを適用
- 分子の表示
という作業を行います.描画する分子の2次元構造はこちらです.
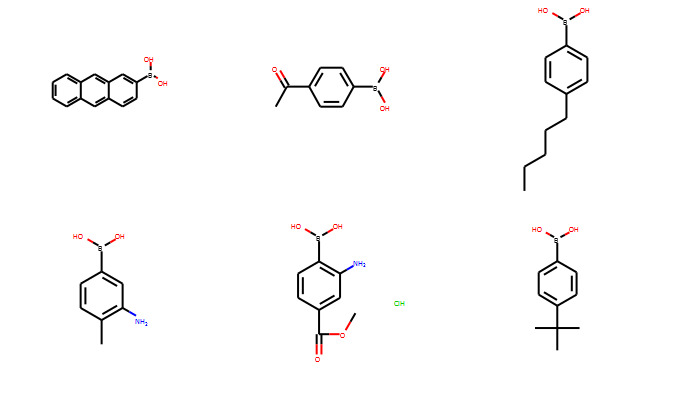
これを以下のように立体表記していきます.
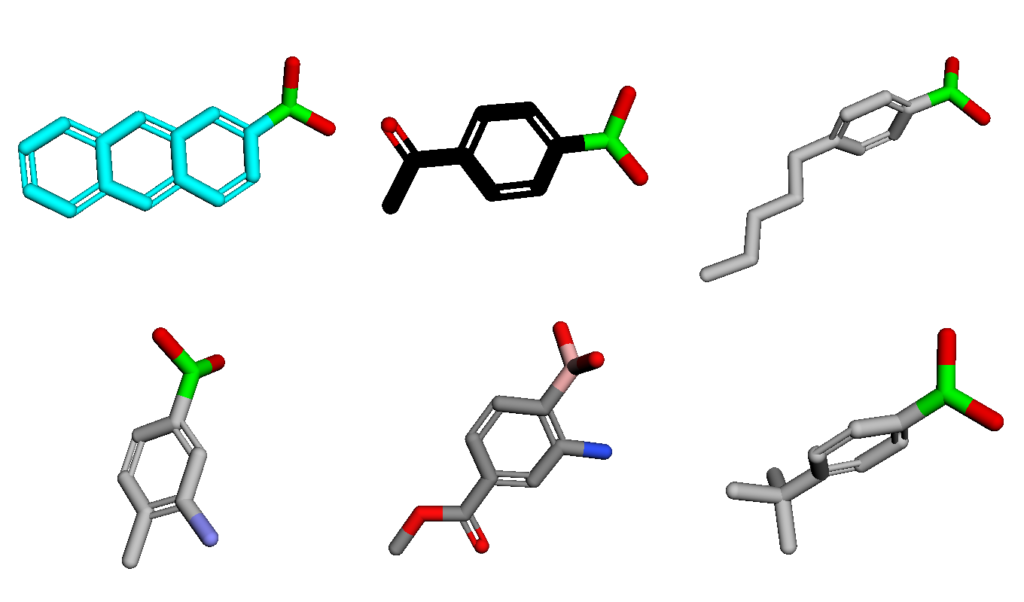
py3Dmolのカラーパレット
py3DmolではColorschemeSpec型というクラスにカラーパレットが定義されています.今回は以下のパレットから設定しています.
| カラーパレット | 説明 |
|---|---|
| ‘color’Carbon | 炭素の色をcolorで設定.炭素以外の色はdefaultパレットから使用 |
| ssPyMOL | PyMolの第2カラーパレット |
| ssJmol | Jmolの第2カラーパレット |
| Jmol | Jmolの第1カラーパレット |
| default | デフォルトのカラーパレット |
コード例
以上の内容を反映したコードは下記の通りです.
add_mols = []
for m in mols[:6]:
m_h = Chem.AddHs(m)
AllChem.EmbedMolecule(m_h, AllChem.ETKDG())
add_mols.append(m_h)
color_scheme = ['cyanCarbon', 'blackCarbon', 'ssPyMOL', 'ssJmol', 'Jmol', 'default']
view = py3Dmol.view(width=680, height=400, viewergrid=(2,3), linked=False)
for (n, (i,j)), c in zip(enumerate([(a,b) for a in range(2) for b in range(3)]), color_scheme):
mb = Chem.MolToMolBlock(add_mols[n])
view.addModel(mb, 'sdf', {'keepH': False}, viewer=(i,j))
view.setStyle({'stick': {'colorscheme': c}}, viewer=(i,j))
view.zoomTo()
view.show()
例4:分子を重ね合わせてモデル毎に異なる色で描画
この例では同一のグリッドに複数の分子を重ね合わせて表示し,それぞれを異なる色で描画してみます.今回は異なる色(color)を設定していますが,当然カラースキーム(colorscheme)の適用も可能です.
- 表示する分子の立体構造を準備し,鋳型を基準に重ね合わせを行う
- 分子をviewerに追加
- それぞれの分子に異なる色を適用
- 分子の表示
という作業を行います.描画する分子の2次元構造はこちらです.
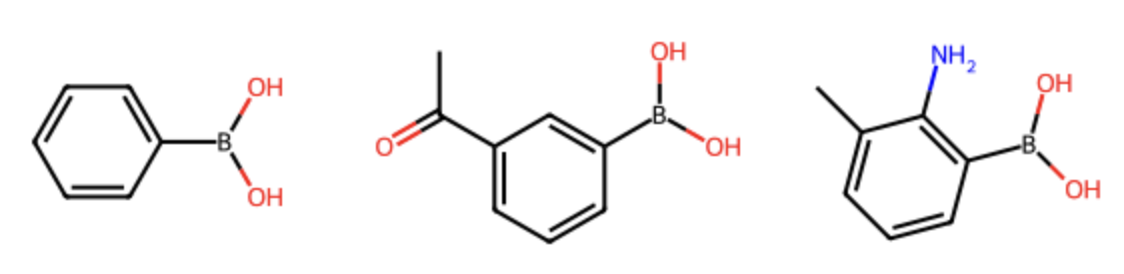
これら分子のフェニルボロン酸部位を鋳型として重ね合わせた上で,灰色,シアン,ピンクと異なる色で描画します.
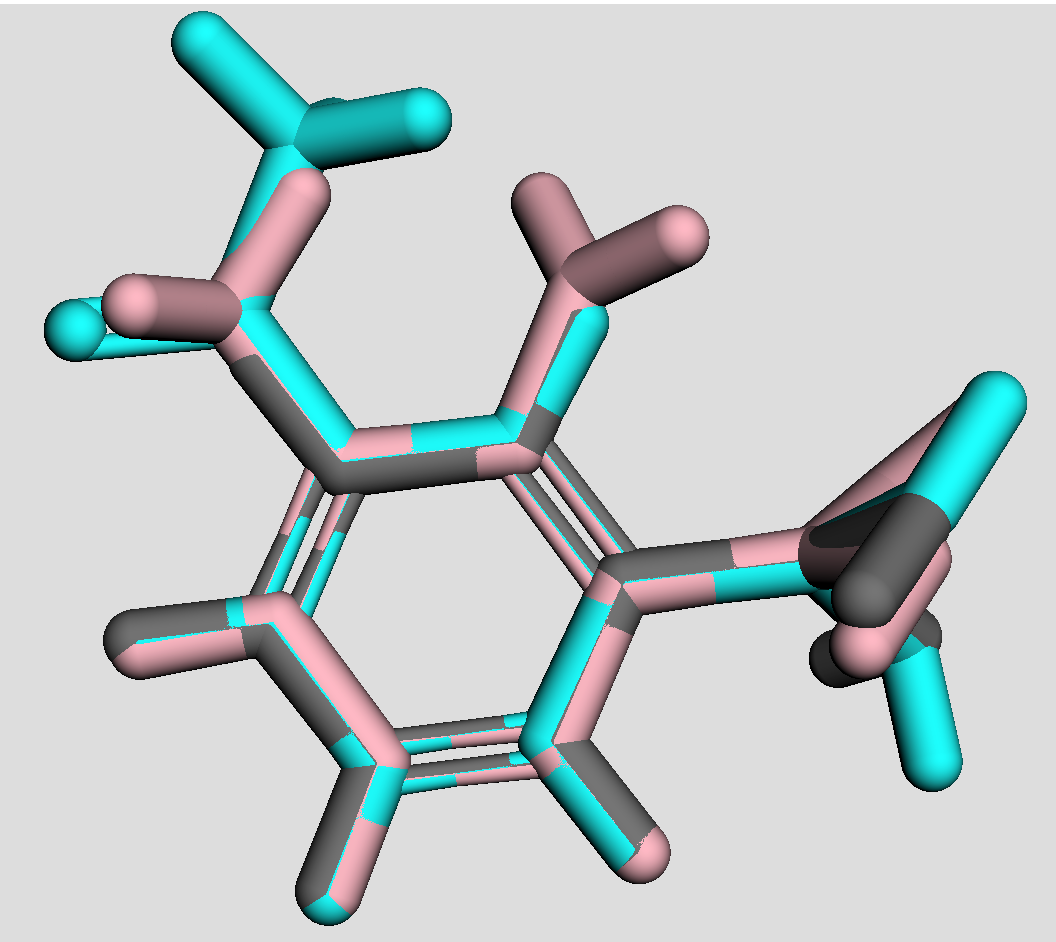
モデルの選択
前述の分子の部分選択では「chain」などを指定することで,分子の部分構造毎に描画設定を変えてました.viewerに複数のモデルを追加した場合,「model」で追加した順番を指定することで,モデル毎に設定を変えることが可能となります.
コード例
以上の内容を反映したコードは下記の通りです.
## 分子の準備
m1 = Chem.MolFromSmiles('c1ccccc1B(O)O')
m2 = Chem.MolFromSmiles('c1ccc(C(=O)C)cc1B(O)O')
m3 = Chem.MolFromSmiles('c1ccc(C)c(N)c1B(O)O')
## 分子の立体構造を準備
m1 = Chem.AddHs(m1)
AllChem.EmbedMolecule(m1, AllChem.ETKDGv3())
m2 = Chem.AddHs(m2)
AllChem.EmbedMolecule(m2, AllChem.ETKDGv3())
m3 = Chem.AddHs(m3)
AllChem.EmbedMolecule(m3, AllChem.ETKDGv3())
## 分子をフェニルボロン酸を鋳型として重ね合わせる
template = Chem.MolFromSmiles('c1ccccc1B')
refMatch = m1.GetSubstructMatch(template)
for mol in [m2, m3]:
prbMatch = mol.GetSubstructMatch(template)
AllChem.AlignMol(prbMol=mol,
refMol=m1,
atomMap=list(zip(prbMatch, refMatch)))
## py3Dmolで表示
view = py3Dmol.view(width=600, height=600)
colors = ['gray', 'cyan', 'pink']
for i, (mol, color) in enumerate(zip([m1, m2, m3], colors)):
mb = Chem.MolToMolBlock(mol)
view.addModel(mb, 'sdf')
### modelの選択と色の指定
view.setStyle({'model': i}, {'stick': {'color': color}})
view.setBackgroundColor('#e1e1e1')
view.show()
終わりに
今回はJupyter上での化学構造描画ライブラリとして人気の高いpy3Dmolの使い方を学んできました.今回紹介しなかった内容として,描画領域のアニメーションが挙げられます.これらの魅せるための機能はプレゼンテーションなどで使うのでなければあまり必要がないと思いますので,興味がある方は公式ドキュメントを読んでみてください.
3次元構造を自在に表現できるようになったので,コンフォマーが扱いやすくなりました.次はコンフォマーの生成とその分析を分子力学法を扱いながら見ていきましょう.
>>次の記事:「RDKitによるコンフォマーの生成」

コメント
細かいことだし、読む人はみんな理解できると思うんですが
描画する分子…のところで、addModelがaddMoldelになってます。
細かい指摘で申し訳ないです。
コメントありがとうございます.修正させて頂きました.
少し気になったのですが、重ねたときに片方は色を変えることはできるのですか?
コメントありがとうございます.具体例4として追記させて頂きました.