1060以上と言われる広大なケミカルスペースの中から,なんらかの基準を用いることでより医薬品になりやすそうな化合物を選択し,優先的に評価を行っていくことが創薬研究においては大切です.
例えば「RDKitにおける記述子の扱い方をリピンスキーの法則を通して学ぶ」という記事で扱った「ルール・オブ・ファイブ」は薬らしさを評価する基準として最も有名なものと言えます.リピンスキールールはもともと経口医薬品のためのガイドラインとしての位置づけでしたが,現実的には,ルールに当てはまらない化合物を除外するフィルターとして使われることが多いです.
とはいえ,例えばリピトールのような大成功をおさめた薬すら,リピンスキールールの全てには合致しません.従ってルール・オブ・ファイブをフィルターとして化合物を「ゼロ」と「イチ」の2つのクラスに分類するような運用方法は好ましくないと言えるでしょう.
今回取り上げるQED(Quantitative Estimate of Drug-likeness)は薬らしさを0から1の間の数値として定量化する試みになります.そのため,リピンスキールールで見られたようなカットオフの問題が生じません.
薬らしさ(ドラッグライクスネス; drug-likness)
リピンスキーが示したように,承認されている薬の物理化学的パラメーターを見てみると,ある狭い範囲に化合物が分布していることがわかります.このケミカルスペースの中に入る化合物を「薬らしい(drug-like)」といいます.
つまり薬らしさとは化学構造上の特徴ではなく,いくつかの物性パラメーターの組み合わせによって定まる指標になります.いくつかの指標が薬らしさを評価する際に提唱されていますが,もっとも影響力のあるものがリピンスキーらによるルール・オブ・ファイブになります.QEDも複数の分子記述子を組み合わせることで薬らしさを評価する方法の1つです.
薬らしさの定量化:QEDとは
QEDは771個の経口医薬品をデータセットに用いて,次の8つの記述子を用いてモデル化されました.
- 分子量(MW)
- logP(ALOGP)
- 水素結合ドナーの数(HBDs)
- 水素結合アクセプターの数(HBAs)
- 極性表面積(PSA)
- 回転可能結合数(ROTBs)
- 芳香環の数(AROMs)
- 忌避構造の数(ALERTS)
採用されている記述子のリストから,ルール・オブ・ファイブに強く影響を受けていることが伺えます.またいずれもよく使われる記述子で,本サイトでもいくつかは取り上げています.
- LogPについては「ケモインフォマティクスとLogP計算:CLogPのCは”calculated”ではありません」
- 極性表面積については「トポロジカル極性表面積は計算コストの低い推定PSA」
- AROMについては「RDKitで化合物の芳香族度合を示す分子記述子を計算する」
などの記事で解説しています.参照してみてください.
3つのQED
実際にQEDの定義を決定する際には,各記述子の重み付けを決定する必要があります.シャノンのエントロピー(QEDで説明できる情報量)を最大化するように係数を決めることとし,次の3つの場合を考えています.
- QEDw, max: 最大のエントロピーを与える係数
- QEDw,mo: 上位1000のエントロピーを与える係数の平均
- QEDw,u: 全ての記述子を等価に考慮(係数を1.0)
興味深い点として,QEDw,maxの場合には「PSAとHBAの係数がゼロ」であることが挙げられます.すなわち,これらに含まれる情報は他の記述子によってカバーされていると示唆されます.これは論文中で行われている主成分分析によっても支持されました.
RDKitにおけるQEDの実装
RDKitではQEDはChem.QEDモジュールに実装されています.APIの利用方法は公式ドキュメントにまとめられています.
RDKitでのQEDの計算方法
Chem.QED.weights_mean(mol)
Chem.QED.weights_none(mol)
Chem.QED.qed(mol)
論文中の3つの重み付けパターンはQED.weights_xxxメソッドに対応しています.またQED.qedメソッドはデフォルトではweights_meanと同じ重み付けで計算を行いますが,独自の重み付けパターンで計算させることも可能です(使う機会はほとんどないでしょう).
下のコードでは論文中のFigure2cに類似した分析として,QED値の分布がルール・オブ・ファイブフィルターの可否でどうかわるかを見ていきたいと思います.
具体的には,
- 必要なライブラリのimport
- SDFファイルをpandasのデータフレームとして読み込む
- pandasのmap関数を用いてQEDを計算
- ルール・オブ・ファイブの可否を判定する関数の定義
- pandasのmap関数を用いてルール・オブ・ファイブ可否を計算
- バイオリンプロットを描画
という手順を行っていきます.
# 1. 必要なライブラリのimport
from rdkit import rdBase, Chem
from rdkit.Chem import PandasTools, QED, Descriptors, rdMolDescriptors
print(rdBase.rdkitVersion) # 2018.09.1
# 2. SDFファイルをpandasのデータフレームとして読み込む
df = PandasTools.LoadSDF('enzyme-in-vivo.sdf')
len(df) ### 653
# 3. QED計算
df['QED'] = df.ROMol.map(QED.qed)
# 4. 関数の定義
def rule_of_five(m):
mw = Descriptors.MolWt(m)
logp = Descriptors.MolLogP(m)
hbd = rdMolDescriptors.CalcNumLipinskiHBD(m)
hba = rdMolDescriptors.CalcNumLipinskiHBA(m)
psa = Descriptors.TPSA(m)
if (mw <= 500 and logp <= 5 and hbd <= 5 and hba <= 10):
return 1
else:
return 0
# 5. ルール・オブ・ファイブ可否を計算
df['Lipinski'] = df.ROMol.map(rule_of_five)
# 6. バイオリンプロットを描画
import matplotlib as mpl
import seaborn as sns
with mpl.style.context('seaborn'):
sns.violinplot(x='Lipinski', y='QED', data=df)
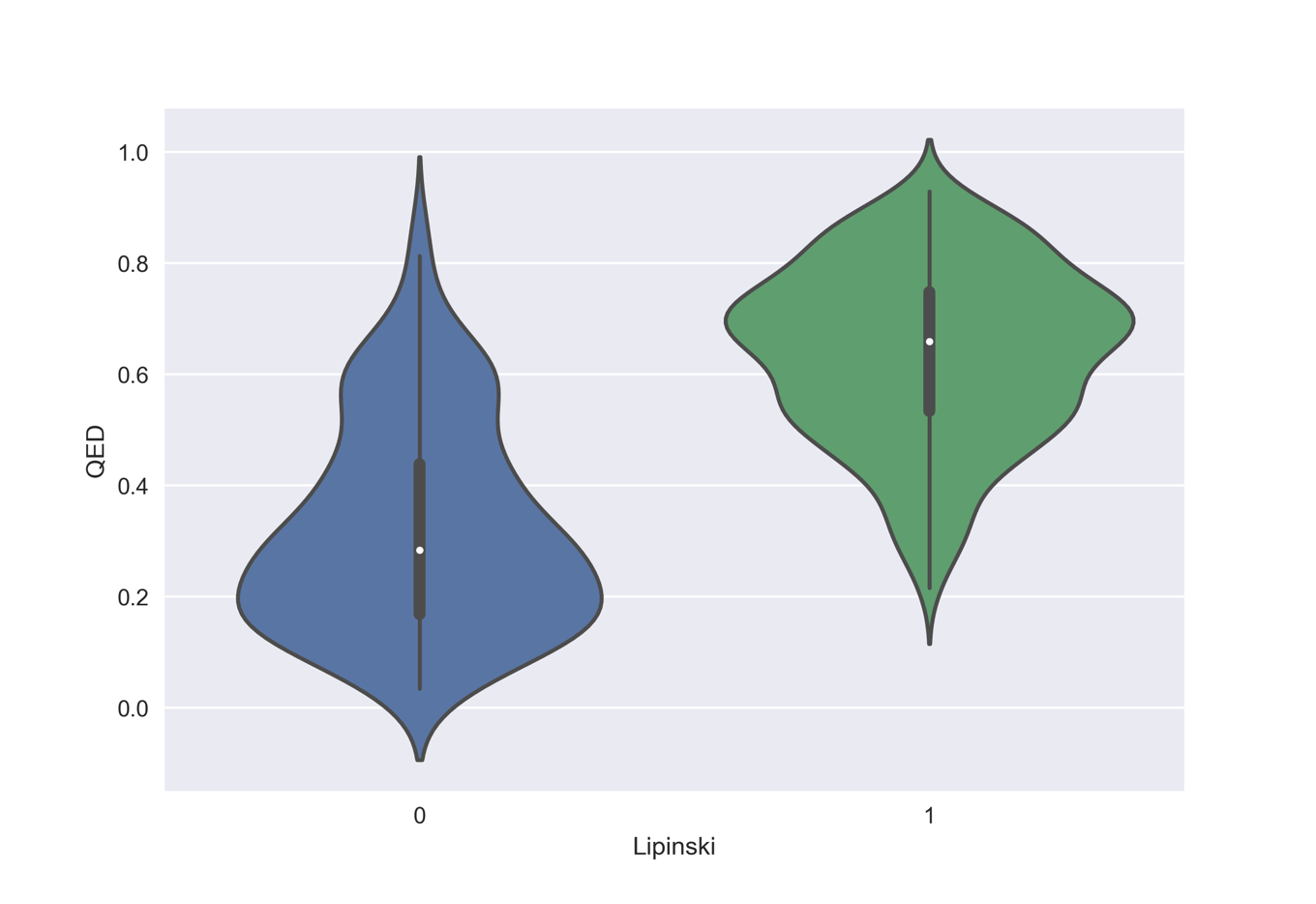
ルール・オブ・ファイブを通らない分子はQEDも低い傾向にありますが,いくつかの分子は高いQEDを有することがわかります.未来のリピトールになりうるような分子を捨ててしまうことになるかもしれません.
終わりに
今回は「RDKitで薬らしさを定量的に評価する」というお題について,
- 薬らしさと(ドラッグライクスネス)は何か
- QEDとは何か
- RDKitにおけるQEDの実装
について見てきました.特にルール・オブ・ファイブで弾かれる化合物の中にもQEDが高い化合物が散見されることを確認しました.
今回はさらりと触れるにとどめましたが,QEDでは忌避構造の有無が評価に組み込まれています.このような「好ましくない部分構造によるフィルタリング」は化合物を評価する際に大切になります.次回はそのような忌避構造リストの中でも有名な「PANISフィルター」について扱ってみようと思います.
>>次の記事:「RDKitのPAINSフィルターで化合物をスクリーニング」
コメント