ケモインフォマティクスではコンピュータで化学構造を扱います.我々が普段扱っている構造式はコンピュータに優しくないため,必然的に別のフォーマットに変換して処理させることになります.
「MOLファイル・SDFとはどんな化学情報ファイルなのか?」という記事では「コネクションテーブル」に基づいたMOLファイルとSDFを取り扱いました.また「SMILES記法は化学構造の線形表記法」という記事では「線形表記法」であるSMILES記法やInChI記法などを説明してきました.これらの形式は有用で,広く使われていますが化合物の全ての性質を表しているわけではありません.そのため,ある目的には別のフォーマットが必要なことが多々あります.
計算化学やケモインフォマティクスで用いられるファイル形式は,ソフトウェア固有の形式も含めると膨大な数にのぼります.今回はそういった様々な化学情報フォーマット間の変換を行う際にもっともよく使われる「Open Babel」というオープンソースソフト・ライブラリについて解説していきます.
Open Babelの起源:Babel
Open Babelは名前からもわかるように1992年にリリースされた「Babel」というソフトをもとにしています.Babelはもともとアリゾナ大学のDaniel Dolataのグループで,Patrick WaltersとMatt Stahlによって作成されたプログラムでした.
Matt StahlがさらにC++で書き直したOBabelを作成,彼がOpenEye社に移った後にそれをもとにGPLライセンスでOELibというソフトを開発しました.一方でPatrick WaltersはVertex Pharmaceuticalsに行きました(2016年にRelay Therapeuticsというスタートアップに移ったようです).
その後,OpenEye社は2001年にOELibを商用ソフトとして書き直すことを決断します.こうして生まれたのが現在のOEChemライブラリというソフトウェアになります.Open BabelはOELibの既存コードをもとにはじまったオープンソースプロジェクトです.
」という記事でさらに詳しく書いていますので,興味のある方は参照してみてください.
Open Babelの特徴
計算化学・ケモインフォマティクスの分野では,用途に応じて色々な種類の化学情報フォーマットが存在します.そのファイル形式間の相互変換を容易にしてくれるのがOpen Babelになります.最も大きな特徴は対応フォーマットの数です.2018年10月現在,なんと100を軽く超える種類のフォーマットに対応しています.
さきに上げたMOLやSMILES,InChIなどの主要な形式に対応しているのは当然として,さまざまな量子化学・分子動力学計算用の個別ソフトの入力・出力ファイルの読み書きができる点が非常に便利です.
またオープンソースであるという特徴から,多くのソフトウェアやウェブサイトに使われています.例えば分子モデリングソフトの「Avogadoro」にもOpen Babelが組み込まれています.そのため利用者は変換作業の裏側を意識することなくファイル形式の変換が可能です.
さきほどの「Open Babel: An open chemical toolbox」という論文の引用が既にに2000回を超えていることからも,このプロジェクトの影響力の大きさがうかがい知れると思います.
実はOpen Babelは単なるファイル変換ツールではなく,分子力学法による構造計算を含んださまざまな機能を内包しています.今回の記事ではファイル変換に特化して解説していきます.その他の機能について興味のある方は「Open Babel: The Open Source Chemistry Toolbox」を参照してみてください.
Openbabelのインストール
Windows
インストーラーが公式サイト「Category:Installation」にありますので,それをダウンロードしてインストールしてください.個人的には使ったことがありませんが,GUIが起動するようです.
Mac
Macの場合は,パッケージマネージャーのhomebrewを使ってインストールするのが簡単です.「open-babel」と間にハイフンが入っていることに注意してください.
brew install open-babel
インストール後に「babel」と「obabel」という2つのコマンドが使えるようになっていれば無事にインストールできています.
Mac用のGUIにはChris Swain作の「iBabel」を使うのがよいと思います.OpenBabelをインストール後に,「Macs in Chemistry:iBabel 3.6」からダウンロードします.
Linux
UbuntuなどのDebian系ディストリビューションを使っている方はパッケージマネージャーで簡単にインストール可能です.
apt-get install openbabel
この場合も,インストール後に「babel」と「obabel」という2つのコマンドが使えるようになっていれば無事にインストールできています.CentOSなどの場合は調べてませんが,おそらくyumを使って同じようにインストール可能なのではないかと思います.
GUIでOpen Babelを使う
手元にWindowsがないため,MacでiBabelを使ってみます.写真を見る限り,WindowsのGUIは3カラムになっていますが,操作感は似ているのではないでしょうか.
下のようなN-メチルグリシン(サルコシン)を描画したChemDrawファイルを用意します.
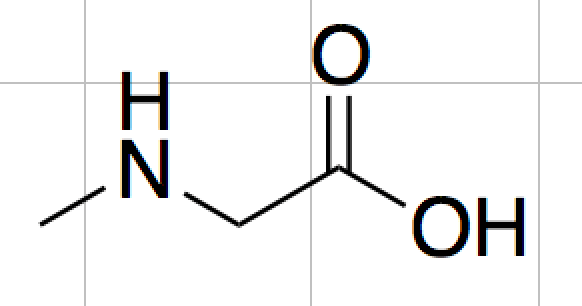
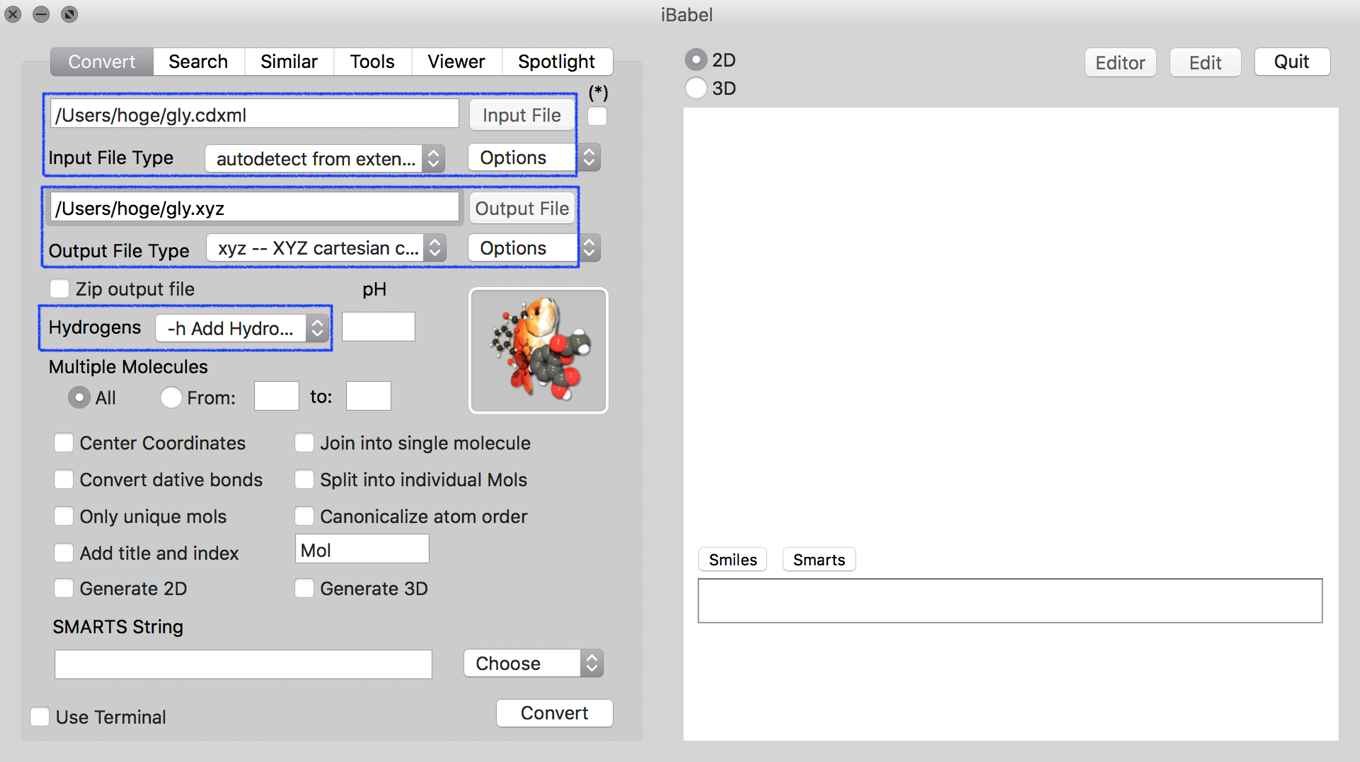
この「cdxml」ファイルを,量子計算などでよく使われる座標情報の入った「xyz」形式に変換してみましょう.ここでは水素原子をきちんと書き込むことにします.InputファイルとOutputファイルの形式やファイル名を設定し,右下の「Convert」を押します.
得られたxyzファイルの中身は以下のようになっています.追加した水素原子も含めて2次元ですが,座標が入っています.
13 3 C 33.50000 32.00000 0.00000 C 45.97000 24.80000 0.00000 N 21.07000 24.82000 0.00000 O 58.40000 31.98000 0.00000 O 45.97000 10.45000 0.00000 C 8.65000 32.00000 0.00000 H 33.49990 33.07000 0.00000 H 32.57327 32.53486 0.00000 H 21.06982 23.80000 0.00000 H 59.21157 31.51167 0.00000 H 8.65060 33.07000 0.00000 H 7.72305 31.46552 0.00000 H 7.72365 32.53552 0.00000
対応フォーマットについては「Input File Type」や「Output File Type」を選択することで,長いプルダウンメニューが現れますので,そこから選択してください.

コマンドラインでOpen Babelを使う
小数のファイルを変換するだけならばGUIの利用でも問題ありませんが,大量のファイルを処理したい場合にはコマンドラインの利用が必須になります.「babel」と「obabel」という2つのコマンドは似ていますが,細かい部分が異なります.obabelの方がオプションの設定を細かくできるようですが,ファイルフォーマットの変換を行いたいだけならそこまで違いはないと思います.
babel/obabelを使ったファイル変換
obabel option -iformat1 filename1 -oformat2 -O filename2
ファイルの変換は-iと-oを接頭辞にファイル形式,続いてファイル名を与えるだけで指定のフォーマットに変換可能です.もしもファイル形式を省略してファイル名だけを与えた場合には,Open Babelが拡張子からファイル名を推定して変換をおこないます.
オプションも色々ありますが,最もよく使うのは水素を付加する「h」だと思います.詳しくはmanを読むか,「Tutorial:Basic Usage」を参照してください.
下の例では先ほどのサルコシンを描画したChemDrawファイルを,
- babelを使ってpdb
- obabelを使ってSDF
へと変換してみます.どちらも水素を付加しています.
babel -h -icdxml ./gly.cdxml -opdb ./gly.pdb obabel -h -icdxml ./gly.cdxml -osdf -O ./gly.sdf
1 molecule converted 20 audit log messages 1 molecule converted
簡単に変換が行えました.
pythonからOpen Babelを使う
Open BabelはC++で書かれたプログラムになります.他のプログラミング言語から使うためラッパーとしてはRuby,Perl,Java用のものも存在しますが,ここではpythonから使う方法を紹介します.
pythonからOpen Babelを使うには2つの方法が用意されています.
- openbabel
- pybel
というライブラリ−・モジュールを用いる方法です.openbabelはSWIGを用いて作成されたpythonライブラリでOpen Babelの全ての機能をカバーしています.openbabelのうちよく使う機能を”pythonic”に記述するために開発されたpythonモジュールがpybelになります.
これらのうち,どちらか一方だけを選ばなければいけないというわけでなく,両者は共存可能です.
python用モジュールのインストール
「openbabel」のインストールはcondaを使うのがよいでしょう.公式チャンネルから「eigen」を一緒にインストールすることでpybelも使えるようになります.
conda install -c openbabel openbabel eigen
importを試みてエラーが出なければ,無事にインストールできています.
import openbabel as ob from openbabel import pybel
Pybelモジュールを使ったファイル形式の変換
ここではOpen Babelの根幹機能である「ファイルフォーマットの変換」をpybelを使ってどのように行うかを解説していきます.具体的には,
- pybelを使ってファイルを読み込む
- pybelの分子オブジェクトpybel.Molecule,またはそのリストを作成
- 他の形式に書き出す
という一連の作業を行ってみたいと思います.なおSDFやSMILES形式の読み込みですと,RDKitなどの他のケモインフォマティクス用ライブラリでも可能ですので,ここではOpen Babelでしかできないフォーマット(ChemDraw形式など)を使ってみたいと思います.
入出力の対応フォーマット
pybel.outformats
入力・出力に対応したフォーマットはpybel.informatsとpybel.outformatsに辞書形式で保存されています.2018年10月現在では読み込みに対応しているのが142フォーマット,書き込みに対応しているのが132フォーマットと圧倒的な種類をカバーしています.とは言っても,この中には拡張子が違うだけで実質的な中身は同じフォーマットもありますので,実際の数はもう少し少なくなります.
print(len(pybel.informats), len(pybel.outformats)) ### 142 132
単一分子の読み込み
pybel.readfile(format, file)
SMILESなどの線形表記法はreadstringを使って読み込みます.ファイルに保存された情報を読み出す場合にはreadfileメソッドを使います.SDFなどのファイルから最初の分子にアクセスするにはnext()を使う必要があります.
さきほどのサルコシンを描画したChemDrawファイルを読み込んでみましょう.
m = [x for x in pybel.readfile('cdxml', './gly.cdxml')]
m = m[0]
print(type(m))
### pybel.Molecule
無事にサルコシンがpybel.Moleculeオブジェクトとして作成されました.Moleculeオブジェクトは例えば分子式などのレコードを持っていますし,分子に対する簡単な計算操作なども行えます.また他のファイル形式に書き出すwriteメソッドを使って,他のフォーマットへの変換が行えます.
Molecule.write(format, file)
SMILES形式などの線形表記ではファイル名を指定する必要がありません.ファイルに保存したい場合は,フォーマットとファイル名の両方を指定してください.ここでは先ほどのサルコシンを量子化学計算用プログラムGAMESSのインプットファイル形式に書き出してみます.
m.write('inp', './gly.inp')
今回の場合は座標が2次元しか設定されていませんが,フォーマット自体は整っていますので基本的にはあとは計算手法などを適宜設定することで計算を開始することができそうです.
$CONTRL COORD=CART UNITS=ANGS $END $DATA 3 C1 C 6.0 33.5000000000 32.0000000000 0.0000000000 C 6.0 45.9700000000 24.8000000000 0.0000000000 N 7.0 21.0700000000 24.8200000000 0.0000000000 O 8.0 58.4000000000 31.9800000000 0.0000000000 O 8.0 45.9700000000 10.4500000000 0.0000000000 C 6.0 8.6500000000 32.0000000000 0.0000000000 $END
複数分子の読み込み
複数分子の読み込みの場合は基本的には同じです.ここでは下のようにいくつかのボロン酸誘導体を描画したChemDrawファイルを入力ファイルとして読み込んでみましょう.
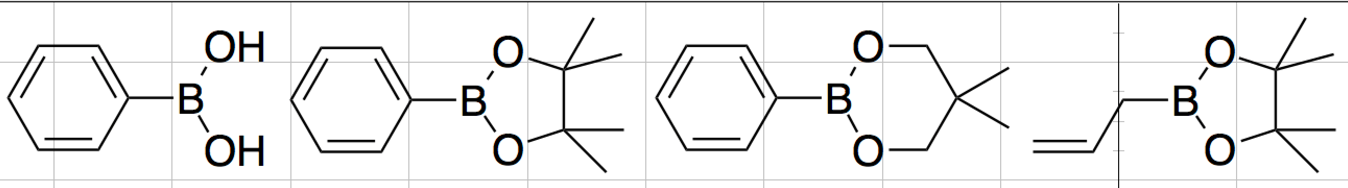
1つの分子の情報を書き出したい場合にはwriteメソッドを使えば問題ありません.それでは複数の分子の情報を1つのファイルに書き込みたい場合はどうすればよいでしょうか?
pybelではこのような目的のためにOutputfileを用意しています.この方法ではファイルオブジェクトのように扱えるpybel.Outputfileオブジェクトを作成します.このオブジェクトに対して各々の分子の情報を追記していくことで複数分子の情報を1つのファイルにまとめることが可能になります.ファイルオブジェクトになりますので,最後にcloseを忘れないようにしてください.Outputfileがwithブロック内で扱えれば便利ですが,どうやら対応していないようです.
mols = [x for x in pybel.readfile('cdxml', './boronic_acids.cdxml')]
output = pybel.Outputfile('sdf', './boronic_acids.sdf')
for m in mols:
output.write(m)
output.close()
終わりに
今回はさまざまな化学情報フォーマット間の変換方法としてOpen Babelの使い方を説明しました.特に大量の化合物を扱う場合にはコマンドライン・プログラミング言語の利用が不可欠になりますので,是非とも習熟しておきたいところです.
ここまで数回に渡って基本的な化学情報フォーマットと,その扱い方を学んできました.望みの化合物情報を得たら,次はコンピュータに読み込ませて,実際にコンピュータ上で分子をどのように扱うかを学んでいきましょう.
このサイトではpython用のケモインフォマティクス用ライブラリであるRDKitの使い方を初歩から解説しています.今回はOpen Babelを用いて簡単な分子の扱い方を見てきましたが,RDKitではもっと複雑な操作が可能になります.
>>次の記事:「RDKitでケモインフォマティクスに入門」

コメント