これまでに非常に多くの化合物が単離または合成されており,その化学的性質に関する膨大な知見が蓄積されています.これら化合物の情報を一カ所に集めたものが化学データベースになります.今回とりあげるChemSpiderもそういったデータベースのうちの1つです.
この記事では英国化学会(RSC)が管理する化学データベースであるChemSpiderにpythonを使ってアクセスすることで化合物データを取得する方法を見ていきます.ChemSpiderは2018年になってAPIを刷新しましたが,今回紹介する方法は最新版に対応したものになっています.
化学データベースの種類
化学物質に特化して作られたデータベースとして最も有名なものはアメリカ化学会が管理するCASだと思いますが,他にも化合物の性質・用途に応じた様々なデータベースが存在しています.下のテーブルに代表的なものをいくつかまとめます.
| データベース | 説明 |
|---|---|
| CAS | アメリカ化学会が運営.非常に巨大 |
| PubChem | アメリカNIH傘下のNCBIが運営するデータベース |
| ChemSpider | 英国化学会(RSC)が所有する化合物データベース |
| ZINC15 | 主にバーチャルスクリーニング用途に市販化合物を集めたデータベース |
| ChEMBL | 文献情報から化学構造と生物活性データを集めたデータベース |
| PDB | 主に生体高分子の固相構造を集めたデータベース |
| CCDC | 主に低分子の固相構造を集めたデータベース |
| SDBS | 有機化学者にお馴染みのスペクトルデータを集めた国産のデータベース |
なかでもPubChemとChemSpiderは収載項目も多く,APIによる自動取得が可能なことから使いやすいです.

ChemSpiderとは
ChemSpiderは2009年にChemZoo社から英国化学会(RSC)が買収した化学データベースです.数多くのデータベースから化合物を収載しており,2021年1月現在で1億を超える化合物が登録されています.PubChemよりやや少ないですが,十分大きなデータベースだと言えます.
基本的な名称や構造データが登録されているのはもちろんのこと,物性値やスペクトルデータなどが参照可能な他,購入可能なサプライヤー情報なども登録されているのが特徴です.構造式からSMILESやInChIなどにも変換可能です.

ChemSpiPyはpythonでChemSpiderにアクセスするライブラリ
RSCはWEB APIを公開しており,ユーザー登録を行うことで利用が可能です.このAPIをpython経由で用いることで色々な作業を自動化することが可能になります.そのためのライブラリがChemSpiPyになります.
なおRSCは2018年に新しいAPIへと移行し,旧API群は利用できなくなりました.これを受けてこれまでChemSpiPyを用いてpythonからクエリーを送っていた方法が変更になります.2018年9月に新しいAPIに対応したChemSpiPyのversion 2.0が発表されましたので,今回はその使い方を見ていきたいと思います.
APIの利用申請
利用に際して,ChemSpiderのウェブサイトにユーザー登録とAPIの利用申請を行う必要があります.Developers Portalからアカウントを作成してください.
利用申請後に出てくる画面で「My Keys」タブを選択します.
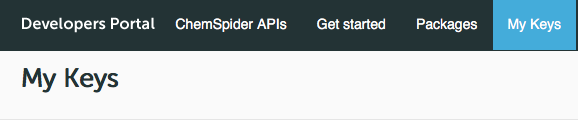
「Keys」というタブで表示されるテーブルの「Consumer Key」という項目の右側に出ている文字列があなたのAPI利用キーになります.大切に保存してください.

なおこのページからAPIキーの利用統計を見ることも可能なので,不正アクセスの有無なども確認できます.
インストール方法
condaを用いた方法(推奨)
公式のドキュメントではversion 2よりcondaの利用が推奨されています.
conda config --add channels conda-forge conda install chemspipy
pipを用いた方法
pipでもインストールが可能です.以前のversionではこちらの方が推奨されていました.
pip install chemspipy
chemspipyの基本的な利用法
先ほどメモしたAPIキーを用いてChemSpiderクラスのインスタンスを作成します.具体的な検索などの操作はこのインスタンスを使っていくことになります.なおAPIキーはユーザー登録の際に入力した個人情報と紐付いているため,他人との共有は控えてください.コードを公開するときにも,自分のAPIキーが露わになっていないかに細心の注意を払ってください.
from chemspipy import ChemSpider
cs = ChemSpider('YOUR-API-KEY')
セキュリティの観点からのおすすめ法
先ほど述べたようにセキュリティの観点からは自分のAPIキーをコードの中に記述しない方が安全です.公式ドキュメントでのおすすめ法は自分のAPIキーを新しい環境変数に設定して格納することのようです.システムの環境変数はos.environによって呼び出すことが出来ます.
os.environではシステムの環境変数を次の例のように辞書型で取得可能です..bashrcなどの設定ファイルに環境変数(この場合はCSAPIKEY)を宣言しておけばコード内にAPIキーを書く必要がなくなるので安心です.
import os api_key = os.environ['CSAPIKEY'] cs = ChemSpider(api_key)
ChemSpider IDでの化合物取得
ChemSpider.get_compounds(list-of-csid)
最も簡単な方法は検索したい化合物のCSID(ChemSpider ID)がわかっている場合になります.この場合は,
- get_compoundメソッドでChemSpiPyの分子に相当するCompoundオブジェクトの取得
- get_compoundsメソッドでは複数分子の取得
が可能です.
ChemSpiPyでの分子,Compoundオブジェクト
ChemSpiPyの分子に相当するCompoundオブジェクトを一度取得すると,それを用いて分子の色々な情報を取り出すことができます.ここではCompoundオブジェクトからアクセスできるレコードについて主要なものをまとめます.詳細については公式ドキュメントの「Compound Properties」の項を参照してください.
なおChemSpiPyでは不要なデータベースへのアクセスを防ぐために,実際にCompoundオブジェクトを作成して特定の情報を取得しようとする段階まではAPIを用いたデータベースへのアクセスは行われません.
| レコード | 説明 |
|---|---|
| Compound.record_id | 化合物のChemSpider ID |
| Compound.image_url | 化合物の2D構造式のPNG画像のURL |
| Compound.molecular_formula | 化合物の分子式 |
| Compound.inchi | 化合物のInChI |
| Compound.inchikey | 化合物のInChIKey |
| Compound.molecular_weight | 化合物の分子量 |
| Compound.common_name | 化合物の一般名 |
| Compound.mol_2d | 化合物の2次元構造のMOLブロック |
| Compound.mol_3d | 化合物の2次元構造のMOLブロック |
| Compound.image | 化合物の2D構造式の画像 |
| Compound.external_references | 化合物に関する外部リソースのリスト |
3次元構造(mol_3d)をそのまま取得できると,他のライブラリとの連携が捗りそうですね.
分子の情報を一気に取得する
ChemSpider.get_details_batch(list-of-csid)
先ほど述べたようにChemSpiPyでは特定の情報にアクセスする段階まではデータベースにアクセスしません.場合によっては化合物を取得した段階で様々な情報を同時に得たい場合もあると思います.
そのような場合に利用するのがget_detailsメソッドになります.複数分子について行いたい場合はget_details_batchを使います.得られる結果は辞書型になります.
ChemSpiPyを使った化合物の検索
実際に化合物の情報が欲しい場合に,化合物のChemSpiderIDが事前にわかっている場合は多くありません.そのため通常の利用状況においては
- 化合物名
- 分子式
- 分子量
- SMILES
- InChI
といった情報から化合物データを取得することになります.当然ChemSpiPyにもこれらの要望に応えるメソッドが実装されています.
最も汎用性のあるメソッドがsearchで,これはChemSpiderウェブサイトの検索ボックスから検索を行うことに相当します.「benzene」と入力すれば化合物名を「C6H6」と入力すれば分子式と判断して検索結果を返してくれます.
得られる結果は下の例のようにCompoundオブジェクトの入ったリスト,Resultオブジェクトになります.
for result in cs.search('alanine'):
print(result.record_id)
582 5735 64234
ChemSpiPyを使った化合物の条件指定検索
ChemSpider.filter_results(query_id)
ChemSpider.filter_results_sdf(query_id)
上で示したsearchメソッドでも多くの場合は目的の化合物に辿り着くことができます.しかし場合によっては分子式で入力したつもりがSMILES形式に解釈されてしまって望みの結果が得られないこともあると思います.そういった場合には検索に用いる条件を指定した上で,検索を実行するのがよいです.
ChemSpiPyにもfilter_’xxxx’というメソッドがいくつかのメソッドが実装されています.これらは‘xxxx’を条件として検索することになります.実際の’xxxx’の値としては
- element
- formula
- inchi
- inchikey
- mass
- name
- smiles
などが実装されています.
filter_’xxxx’メソッドの戻り値は検索結果ではなく,クエリーIDになります.このIDを使ってfilter_resultsメソッドを使うと,ようやく検索結果としてChemSpider IDのリストが得られます.searchメソッドの時のようにCompoundオブジェクトのリストではないことに注意してください.このようにして得たCSIDを使って再度検索を行うことで目的の化合物が得られます.
下の例では分子式「C6H12」を有する化合物を検索するために
- 「formula」でフィルターした検索を行う
- 得られたクエリーIDを「filter_results」によってCSIDのリストに変換
という作業を行っています.この場合は41個の化合物が見つかったことがわかります.
q_id = cs.filter_formula('C6H12')
x = cs.filter_results(q_id)
len(x) # 41
他の方法としては,filter_results_sdfメソッドを用いることでクエリーIDに対する検索結果をSDFとして取得可能です.多くの場合はこちらの方が望ましいかもしれません.
検索結果に対する操作
検索結果はCompoudオブジェクトを内包するリストであるだけでなく,Resultオブジェクトに対する操作も行えます.ここでは得られた結果を全て含むSDFを出力するsdfメソッドを見てみましょう.
SDFは標準のファイルオブジェクトとして書き込むことが可能です.注意点としてはbytesオブジェクトとして出力されますので,オプションに’b’を付加する必要があります.
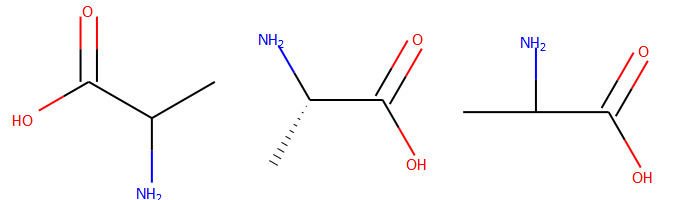
下の例では’alanine’で検索した結果をSDFとして保存しています.
results = cs.search('alanine')
with open('alanine_sample.sdf', 'bw') as f:
f.write(results.sdf)
出力されたSDFには以下のような構造3つが含まれていました.
フォーマット形式の変換
入力したフォーマット形式を,指定したフォーマットに変換します.対応フォーマットは
- SMILES
- InChI
- InChIKey
- Mol
の4種類になります.ウェブ上で変換する場合には結構重宝するこの変換機能ですが,正直python経由で行う場合にはRDKitを使った方が速いと思うのであまり使い道がないかもしれません.
終わりに
実験化学者にとって大切なことは,日常的に行っている作業のうち一部だけでも自動化することでプログラミングの考え方・恩恵を実感することだと思います.一見すると日々の業務とはかけ離れた内容かも知れませんが,メリットが見えることで,勉強のモチベーションがわいてくるのではないでしょうか.
今回のエントリーではpythonを使って化学情報を扱う例として,ChemSpiPyを用いたChemSpiderからの化合物情報取得の自動化を行いました.実際には今回のような情報の取得だけで作業が完結することはありません.
次回以降ではそもそもどのように化合物をコンピュータ上で表現するかから始めます.pythonのケモインフォマティクス用ライブラリRDKitを用いて分析を行っていきましょう.
>>次の記事:「RDKitでケモインフォマティクスに入門」
コメント