我々が化合物を扱う際に用いている構造式は,人間の目にはわかりやすいですがコンピュータにはわかりにくいものです.そのためコンピュータにわかりやすい形式で化合物を表現してあげることが重要となりますが,用途によって望ましい形式が変わってきます.
例えばケモインフォマティクスのように分子をグラフ構造として扱う場合には,原子間の結合情報が重要となります.一方で第一原理(ab initio)計算では,原子の位置が重要であり結合情報は不要です.そのため計算化学からケモインフォマティクス分野へと化合物情報を渡す際には,原子間の結合情報を推定する必要があります.
今回はRDKitの2022.09のアップデートから追加された「xyz2mol」について見ていきたいと思います.その名の通り,XYZ形式をRDKitのMOLオブジェクトへと変換するメソッドになります.RDKitでは2019.09のアップデートでMOLオブジェクトからXYZ形式への書き出しに対応しましたから,今回のアップデートで双方向の変換が可能となりました.
XYZファイルとは
XYZファイルとは拡張子が「.xyz」となるファイル形式で,各原子の3次元空間における座標をオングストローム単位で記述することで,分子の構造を表現したものになります.直交座標系のXYZ軸に沿った原子の位置を示すことから,デカルト座標系(Cartesian coordinate)とも呼びます.
特に計算化学の分野でよく使われる入力形式ですが,標準化された規格がないため,ソフトウェアによってそのフォーマットがやや異なるのが難点になります.
原子の座標を与えれば,原子間の距離がわかりますので,どの原子間に「結合」が存在するかを推定することが可能です.そのためXYZ形式のみで分子構造を完全に表現できると言えます.
通常,分子軌道法やDFT計算のインプットファイルにはXYZ形式のみの指定で十分です.一方で分子力学法計算では,各原子に力場パラメータを当てはめる際に結合種類の情報が必要になりますので,結合リストの情報が必要になります.
基本フォーマット
XYZ形式で一般的に広く用いられるフォーマットは下記のようになります.今回扱うRDKitでの入出力もこのフォーマットに対応しています.
分子内の原子数 コメント行 元素記号 x座標 y座標 z座標 元素記号 x座標 y座標 z座標 ・・・
中心をなすのが以下の
という部分です.
xyz2molとは
もともとはコペンハーゲン大学のJan H. Jensenの研究グループにより開発されたpythonライブラリで,xyzファイルをもとに原子間の結合情報を推定し分子グラフを構築します.
こちらの論文に書かれているアルゴリズムを実装したもになります.
Universal Structure Conversion Method for Organic Molecules: From Atomic Connectivity to Three-Dimensional Geometry
Yeonjoon Kim, Woo Youn Kim Bull. Korean Chem. Soc. 2015, 36, 1769-1777.
分子のグラフ構造が得られれば,それをもとにsmilesへと変換したり,RDKitのMOLオブジェクトへは容易に変換可能です.
この度,Google Summer of Code 2022の成果を受けて,2022.09バージョンからRDKitに統合されました.

準備
今回は以下の環境でコードを実行しています.計算に用いるCPUやメモリは各自の環境に合わせて適宜変更してください.
import sys
import numpy as np
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, rdDetermineBonds
from rdkit.Chem.Draw import IPythonConsole
import psi4
import py3Dmol
print(f'python version:\n{sys.version}')
print(f'numpy version: {np.__version__}')
print(f'rdkit version: {rdBase.rdkitVersion}')
print(f'psi4 version: {psi4.__version__}')
psi4.set_num_threads(8)
psi4.set_memory('16GB')
# python version:
# 3.10.6 | packaged by conda-forge | (main, Aug 22 2022, 20:38:29) [Clang 13.0.1 ]
# numpy version: 1.23.4
# rdkit version: 2022.09.2
# psi4 version: 1.7
xyz2molの使い方
Chem.MolFromXYZBlock(xyz-blocck)
Cheem.rdDetermineBonds.DetermineBonds(mol)
既に有志の方々による2022.09のリリースノート翻訳に簡単な使い方の説明が記されています.基本的な使い方としては,
- MolFromXYZFile/Blockで原子位置を保持したMOLオブジェクトを作成
- DetermineBondsで原子間の結合を推定
という2段階工程になります.
今回はエタノールアミンを例としてみましょう.
まず
- smilesからRDKitのMOLオブジェクトを作成
- 水素原子の付加
- 立体構造の生成
を行い,xyz形式にて出力します.
mol = Chem.MolFromSmiles('NCCO')
mol_h = Chem.AddHs(mol)
AllChem.EmbedMolecule(mol_h)
xyz = Chem.MolToXYZBlock(mol_h)
print(xyz)
この段階でのXYZ形式は以下の通りでした.
11 N 1.535699 -0.024779 -0.379784 C 0.355985 0.237892 0.404670 C -0.875027 -0.439099 -0.136693 O -1.981254 -0.150007 0.659909 H 2.114964 0.851773 -0.481723 H 2.182283 -0.723676 0.082840 H 0.549831 -0.206596 1.420569 H 0.180192 1.315725 0.589495 H -1.123522 -0.146303 -1.159337 H -0.748728 -1.541147 -0.105222 H -2.190422 0.826217 0.656805
続いて作成したXYZブロックをもとに,RDKitのMOLオブジェクト作成を試みます.
mol2 = Chem.MolFromXYZBlock(xyz) print(Chem.MolToSmiles(mol2)) # C.C.N.O.[HH].[HH].[HH].[HH].[HH].[HH].[HH]
SMILESからもわかるように,この段階では結合情報がありません.
結合情報がないことはMOLブロックからもわかります.
RDKit 3D
11 0 0 0 0 0 0 0 0 0999 V2000
1.5357 -0.0248 -0.3798 N 0 0 0 0 0 0 0 0 0 0 0 0
0.3560 0.2379 0.4047 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8750 -0.4391 -0.1367 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9813 -0.1500 0.6599 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1150 0.8518 -0.4817 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1823 -0.7237 0.0828 H 0 0 0 0 0 0 0 0 0 0 0 0
0.5498 -0.2066 1.4206 H 0 0 0 0 0 0 0 0 0 0 0 0
0.1802 1.3157 0.5895 H 0 0 0 0 0 0 0 0 0 0 0 0
-1.1235 -0.1463 -1.1593 H 0 0 0 0 0 0 0 0 0 0 0 0
-0.7487 -1.5411 -0.1052 H 0 0 0 0 0 0 0 0 0 0 0 0
-2.1904 0.8262 0.6568 H 0 0 0 0 0 0 0 0 0 0 0 0
M END
続いてDetermineBondsメソッドを用いることで原子間の結合情報を推定します.
rdDetermineBonds.DetermineBonds(mol2) print(Chem.MolToMolBlock(mol2))
MOLブロックに結合情報が賦与されました.
RDKit 3D
11 10 0 0 0 0 0 0 0 0999 V2000
1.5357 -0.0248 -0.3798 N 0 0 0 0 0 0 0 0 0 0 0 0
0.3560 0.2379 0.4047 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8750 -0.4391 -0.1367 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.9813 -0.1500 0.6599 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1150 0.8518 -0.4817 H 0 0 0 0 0 0 0 0 0 0 0 0
2.1823 -0.7237 0.0828 H 0 0 0 0 0 0 0 0 0 0 0 0
0.5498 -0.2066 1.4206 H 0 0 0 0 0 0 0 0 0 0 0 0
0.1802 1.3157 0.5895 H 0 0 0 0 0 0 0 0 0 0 0 0
-1.1235 -0.1463 -1.1593 H 0 0 0 0 0 0 0 0 0 0 0 0
-0.7487 -1.5411 -0.1052 H 0 0 0 0 0 0 0 0 0 0 0 0
-2.1904 0.8262 0.6568 H 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0
1 5 1 0
1 6 1 0
2 3 1 0
2 7 1 1
2 8 1 0
3 4 1 0
3 9 1 6
3 10 1 0
4 11 1 0
M END
具体例:RDKitとpsi4の連携
XYZ形式は計算化学の分野で汎用されるフォーマットですので,今度はpythonの量子化学計算用ライブラリであるpsi4を用いて構造最適化を行い,その構造を基にRDKitのMOLオブジェクトを作成してみます.
分子としては以下のようにビフェニルを母骨格としてオルト位が塩素原子で置換された化合物を用い,塩素原子の置換程度による二面角の変化を調べてみます.
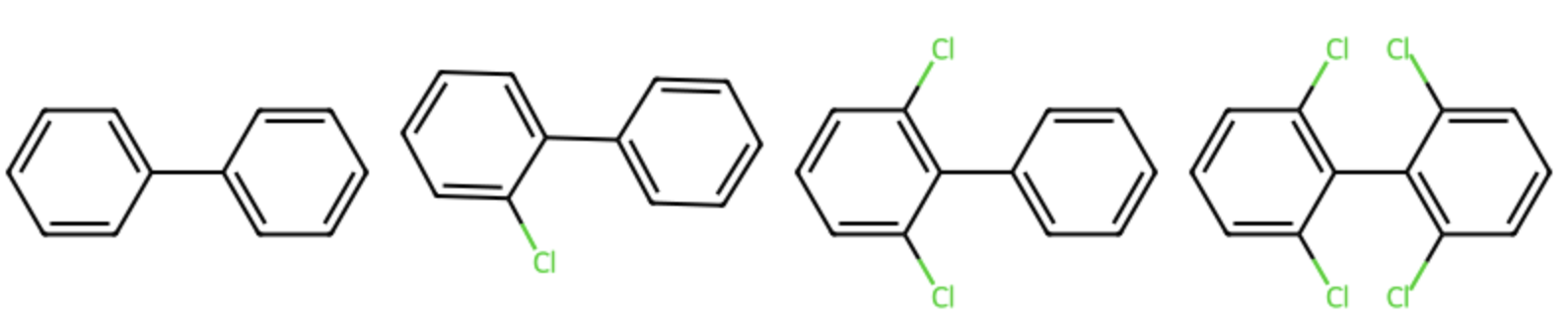
以下のコードでは
- psi4を用いて各分子をHF/STO-3Gレベルで構造最適化,振動数計算を行い安定構造であることを確認
- 最適化構造をxyz形式で保存
- 保存したxyzファイルからRDKitを用いてMOLオブジェクトを作成
- 芳香環を鋳型として各分子の座標を合わせる
- py3Dmolを用いて立体構造を描画
の順番で処理をしていきます.
psi4を用いた構造最適化・振動数計算
psi4.optimize(level-of-theory, molecule)
psi4.frequency(level-of-theory, molecule, return_wfn=bool)
psi4.Molecule.save_string_xyz_file()
本ブログでは「計算化学にpythonとpsi4で入門」という記事からはじめて,psi4の使い方を取り上げています.構造最適化,振動数計算については以下の記事でそれぞれ解説していますので,詳細を知りたい方は参照してみてください.
- 構造最適化:「計算化学の構造最適化の基本をpsi4で学ぶ」
- 振動数計算:「psi4における振動数計算:IRスペクトルや異性体間のエネルギー差を計算」
以下のコードでは
- psi4のMoleculeオブジェクトを作成
- HF/STO-3Gレベルで構造最適化
- 最適化構造を用いてHF/STO-3Gレベルで振動数計算
- 負の振動数がないことを確認
- 最適化構造をxyzファイルに保存
という順番で処理を行っています.
biphenyl = psi4.geometry('''
0 1
C -1.60677167 0.57674840 0.00000000
C -0.20435967 0.57674840 0.00000000
C 0.49918233 1.77936240 0.00000000
C -0.18689967 2.99365840 -0.00276100
C -1.58155267 3.00175740 -0.00410600
C -2.28951367 1.80169940 -0.00129900
C -2.34720267 -0.68369560 0.00125500
C -1.91583367 -1.76170960 -0.78523500
C -2.62398167 -2.96161460 -0.78348600
C -3.76696067 -3.10067760 0.00359000
C -4.20140767 -2.03351860 0.78943600
C -3.49817067 -0.83068560 0.78885600
H 0.34069133 -0.37912460 0.00768300
H 1.59903833 1.76928540 0.00327200
H 0.37006933 3.94170340 -0.00385900
H -2.12584767 3.95754040 -0.00849600
H -3.38981967 1.81245940 -0.00794900
H -1.01781667 -1.65416360 -1.41200100
H -2.27983967 -3.80040660 -1.40621400
H -4.32385867 -4.04876460 0.00450100
H -5.10115267 -2.14040360 1.41300000
H -3.84113667 0.00700840 1.41453700
''')
Cl_biphenyl = psi4.geometry('''
0 1
C -1.60677167 0.57674840 0.00000000
C -0.20435967 0.57674840 0.00000000
C 0.49918233 1.77936240 0.00000000
C -0.18689967 2.99365840 -0.00276100
C -1.58155267 3.00175740 -0.00410600
C -2.28951367 1.80169940 -0.00129900
C -2.34720267 -0.68369560 0.00125500
C -1.91583367 -1.76170960 -0.78523500
C -2.62398167 -2.96161460 -0.78348600
C -3.76696067 -3.10067760 0.00359000
C -4.20140767 -2.03351860 0.78943600
C -3.49817067 -0.83068560 0.78885600
H 0.34069133 -0.37912460 0.00768300
H 1.59903833 1.76928540 0.00327200
H 0.37006933 3.94170340 -0.00385900
H -2.12584767 3.95754040 -0.00849600
H -3.38981967 1.81245940 -0.00794900
H -1.01781667 -1.65416360 -1.41200100
H -2.27983967 -3.80040660 -1.40621400
H -4.32385867 -4.04876460 0.00450100
H -5.10115267 -2.14040360 1.41300000
Cl -4.04672734 0.50916334 1.78960093
''')
Cl2_biphenyl = psi4.geometry('''
0 1
C -1.60677167 0.57674840 0.00000000
C -0.20435967 0.57674840 0.00000000
C 0.49918233 1.77936240 0.00000000
C -0.18689967 2.99365840 -0.00276100
C -1.58155267 3.00175740 -0.00410600
C -2.28951367 1.80169940 -0.00129900
C -2.34720267 -0.68369560 0.00125500
C -1.91583367 -1.76170960 -0.78523500
C -2.62398167 -2.96161460 -0.78348600
C -3.76696067 -3.10067760 0.00359000
C -4.20140767 -2.03351860 0.78943600
C -3.49817067 -0.83068560 0.78885600
H 0.34069133 -0.37912460 0.00768300
H 1.59903833 1.76928540 0.00327200
H 0.37006933 3.94170340 -0.00385900
H -2.12584767 3.95754040 -0.00849600
H -3.38981967 1.81245940 -0.00794900
H -2.27983967 -3.80040660 -1.40621400
H -4.32385867 -4.04876460 0.00450100
H -5.10115267 -2.14040360 1.41300000
Cl -4.04672734 0.50916334 1.78960093
Cl -0.47950275 -1.58969543 -1.78771423
''')
Cl4_biphenyl = psi4.geometry('''
0 1
C -1.60677167 0.57674840 0.00000000
C -0.20435967 0.57674840 0.00000000
C 0.49918233 1.77936240 0.00000000
C -0.18689967 2.99365840 -0.00276100
C -1.58155267 3.00175740 -0.00410600
C -2.28951367 1.80169940 -0.00129900
C -2.34720267 -0.68369560 0.00125500
C -1.91583367 -1.76170960 -0.78523500
C -2.62398167 -2.96161460 -0.78348600
C -3.76696067 -3.10067760 0.00359000
C -4.20140767 -2.03351860 0.78943600
C -3.49817067 -0.83068560 0.78885600
H 1.59903833 1.76928540 0.00327200
H 0.37006933 3.94170340 -0.00385900
H -2.12584767 3.95754040 -0.00849600
H -2.27983967 -3.80040660 -1.40621400
H -4.32385867 -4.04876460 0.00450100
H -5.10115267 -2.14040360 1.41300000
Cl -4.04672734 0.50916334 1.78960093
Cl -0.47950275 -1.58969543 -1.78771423
Cl -4.04939739 1.81890948 -0.01193534
Cl 0.66742196 -0.95212235 0.01228857
''')
def check_img(frequencies: np.ndarray) -> bool:
if np.sum(frequencies < 0) == 0:
return True
else:
return False
mols = [biphenyl, Cl_biphenyl, Cl2_biphenyl, Cl4_biphenyl]
for i, mol in enumerate(mols):
print('----------------------------------------')
psi4.set_output_file(f'mol_{i+1}.log')
print(f'## start optimization of No.{i + 1} ##')
print(f'initial structure:\n{mol.save_string_xyz()}\n')
e, wfn = psi4.optimize('hf/sto-3g', molecule=mol, return_wfn=True)
print(f'optimized structure:\n{mol.save_string_xyz()}\n')
print('## start freq of No.{i + 1} ##')
e, wfn = psi4.frequency('hf/sto-3g', molecule=mol,
ref_gradient=wfn.gradient(),
return_wfn=True)
if not check_img(np.array(wfn.frequencies())):
print('##################################')
print('##### OPTIMIZATION FAILED !! #####')
print('##################################')
break
with open(f'optimized_{i + 1}.xyz', 'w') as file:
print(mol.save_string_xyz_file(), file=file)
これでそれぞれの分子の最適化構造を収めたxyzファイルが得られましたので,続いてRDKitによる読み込みを行ってみます.
RDKitを用いてxyzファイルの読み込みとpy3Dmolを用いた立体構造の重ね合わせ表示
今回のように共通の母骨格を有する異なる分子の重ね合わせにはRDKitのAllChem.AlignMolが便利です.AlignMolメソッドではコンフォマーの重ね合わせと同様にatomMapをオプションに与えます.異なる点は分子が異なるため,鋳型とマッチする原子の番号の対応を与える必要があります.下のコードではzipを用いています.
鋳型にする部分構造は片方の芳香環(原子番号0から5)を使い,最初の分子である無置換のビフェニルを基準に残りの分子を重ね合わせていきます.3次元構造の表示にはpy3Dmolを用います.
## XYZファイルからMOLオブジェクトの作成
import glob
files = glob.glob('*.xyz')
mols = []
for file in files:
mol = Chem.MolFromXYZFile(file)
rdDetermineBonds.DetermineBonds(mol)
mols.append(mol)
## 片方の芳香環を鋳型とした立体構造の重ね合わせ
for mol in mols[1:]:
AllChem.AlignMol(prbMol=mol,
refMol=mols[0],
atomMap=list(zip(range(5), range(5)))
)
## py3Dmolによる描画
view = py3Dmol.view(width=400, height=400)
for mol in mols:
view.addModel(Chem.MolToMolBlock(mol), 'sdf')
view.setStyle({'stick': {}})
view.zoomTo()
view.setBackgroundColor('0xeeeeee')
view.show()
重ね合わせた結果は下のようになりました.塩素原子の数が増えるにつれて二面角が直角に近づいていくのが見てとれます.
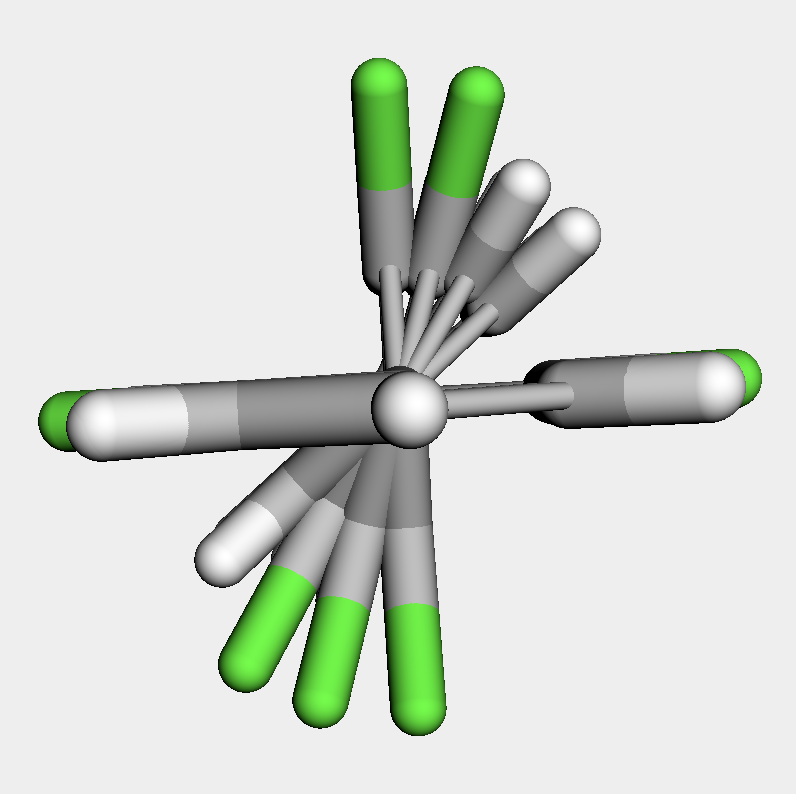
「RDKitを用いて制約付きで立体構造を生成する」という記事で説明したように,rdMolTransforms.GetDihedralDegを用いて実際に各分子の二面角を取得してみます.
from rdkit.Chem import rdMolTransforms
for i, mol in enumerate(mols):
print(f'dihedral angle of {i + 1}: {rdMolTransforms.GetDihedralDeg(mol.GetConformer(-1),1, 0, 6, 11): .1f}')
塩素原子の数が増えるにつれて二面角が小さくなることを数値の面からも確認できました.
dihedral angle of 1: 141.3 dihedral angle of 2: 125.3 dihedral angle of 3: 108.5 dihedral angle of 4: 90.1
終わりに
今回は「RDKitのxyz2molでケモインフォマティクスと計算化学の橋渡し」という話題について,2022.09のアップデートからRDKitに追加されたxyz2molに関し
- xyzファイルとはなにか
- xyz2molとはなにか
− RDKitのxyz2molの使い方 - xyz2molを用いてpsi4とrdkit間でのデータ共有例
などについて,psi4の計算方法やRDKitのMOLオブジェクトの細かい使い方にも触れながら見てきました.
化合物をコンピュータで扱う際,目的によって適切なファイル形式が異なります.一方で人間が化合物を扱う際は,今回の最後の例でも見たように実際に眼で確認することが最もわかりやすいです.py3Dmolは分子の立体構造をJupyter Notebook上で確認することが可能なライブラリーです.引き続き使い方に関し,理解を深めていくのがよいと思います.
>>次の記事:「py3Dmolを使って化学構造をJupyter上で美しく表示する」

コメント