本ブログではJupyter Notebook上で分子構造を描画するためのライブラリーとして「py3Dmolを使って化学構造をJupyter上で美しく表示する」という記事でpy3Dmolについて説明しました.一方で生命科学の分野で,分子構造を可視化する際に使われるポピュラーなソフトのうちの1つは「PyMOL」だと思います.PyMOLは現在はシュレディンガー社によって管理・配布されていますが,ソースコード自体はオープンソースです.
今回はRDKitのMOLオブジェクトをPyMOLを利用して可視化する方法として,RDKit.Chem.PyMOLモジュールを紹介します.この方法により,Jupyter Notebook上からPyMOLを操作し,分子構造を表示することができるようになります.
準備:PyMOLの起動
RDKitからPyMOLを操作するためには「XML-RPC server」を利用します.使い方は下記のようにコマンドラインからPyMOL起動時に「-R」オプションをつけるだけです.
$ pymol -R
起動したPyMOLの画面上に下のように「xml-rpc server running」と表示が出ていれば大丈夫です.
![]()
分子の準備
今回も3次元構造を有する分子を扱いますので,「RDKitによる3次元構造の生成」という記事で用いた「Platinum dataset」を用いることとします.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw, PyMol
from rdkit.Chem.Draw import IPythonConsole
print(rdBase.rdkitVersion) # 2018.09.1
suppl = Chem.SDMolSupplier('./platinum_dataset_2017_01.sdf')
mols = [x for x in suppl if x is not None]
len(mols) # 4548
RDKitからのPyMOLの利用
MolViewer.SetDisplayStyle(obj, style)
MolViewer.GetPNG(h, w, preDelay)
MolViewer.SaveFile(filename)
PyMOL関連のコードはRDKit.Chem.Pymolにあります.具体的な手順としてはPyMol.MolViewerオブジェクトをまず作成し,それを通して様々な操作を行っていくことになります.
様々なスタイルで分子を描画
下記のコードでは
- PyMolViewerオブジェクトを作成
- Viewerに分子を表示
- PyMOL上の画面をPNG画像として取得
- PyMOL上の画面を画像として保存
という順番で処理しています.分子の描画には多少の時間がかかりますので,GetPNGメソッドではpreDelayオプションをつけています.
v = PyMol.MolViewer()
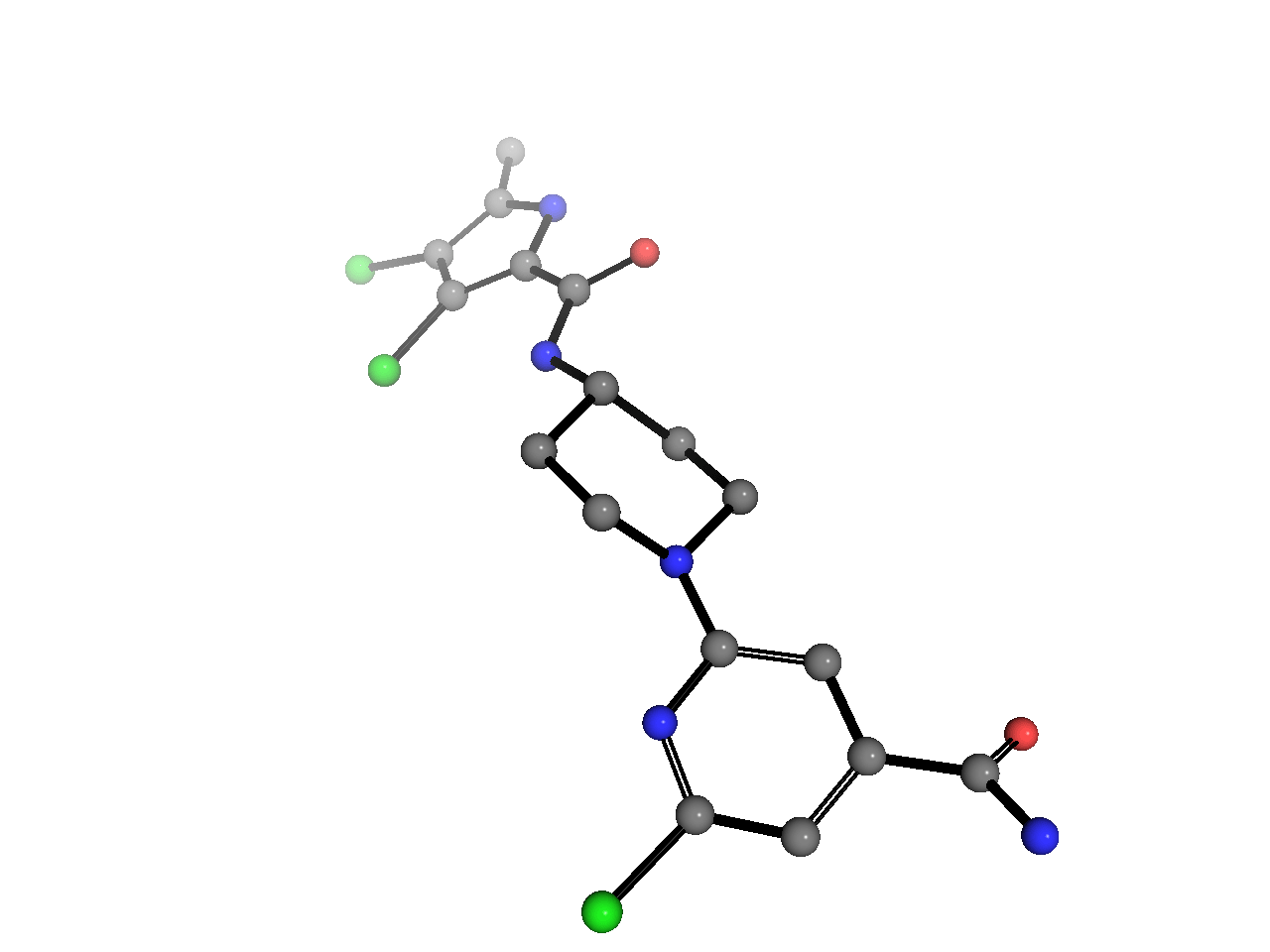
v.ShowMol(mols[0], name='mol')
v.GetPNG(h=350, preDelay=5)
v.SaveFile('./mol0_default.png')
デフォルト設定であるStickモデルで描画されました.
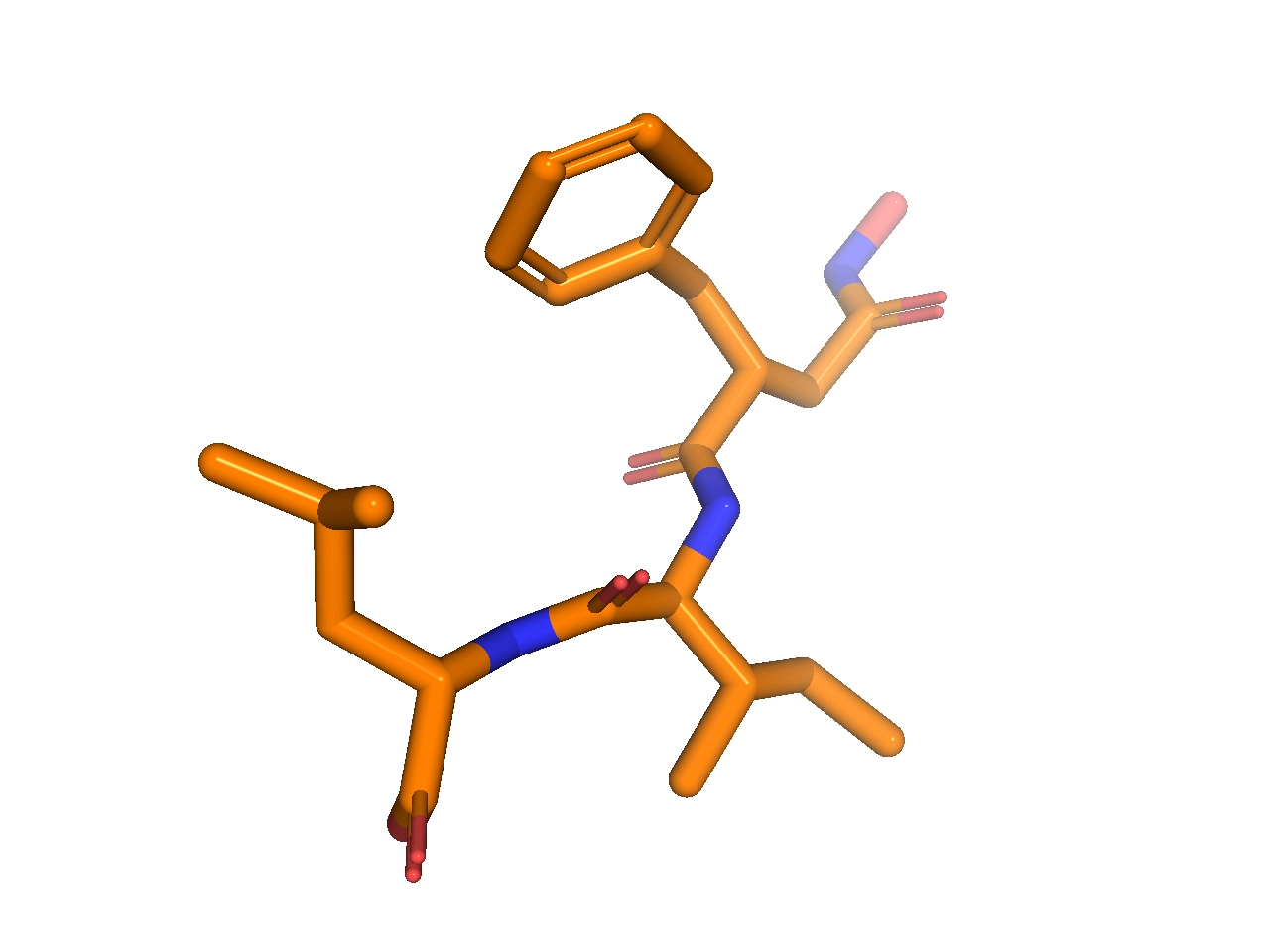
分子の描画形式はSetDisplayStyleメソッドで指定することが可能です.下のコードでは’lines‘を指定しています.
v.ShowMol(mols[0], name='mol')
v.SetDisplayStyle('mol', 'lines')
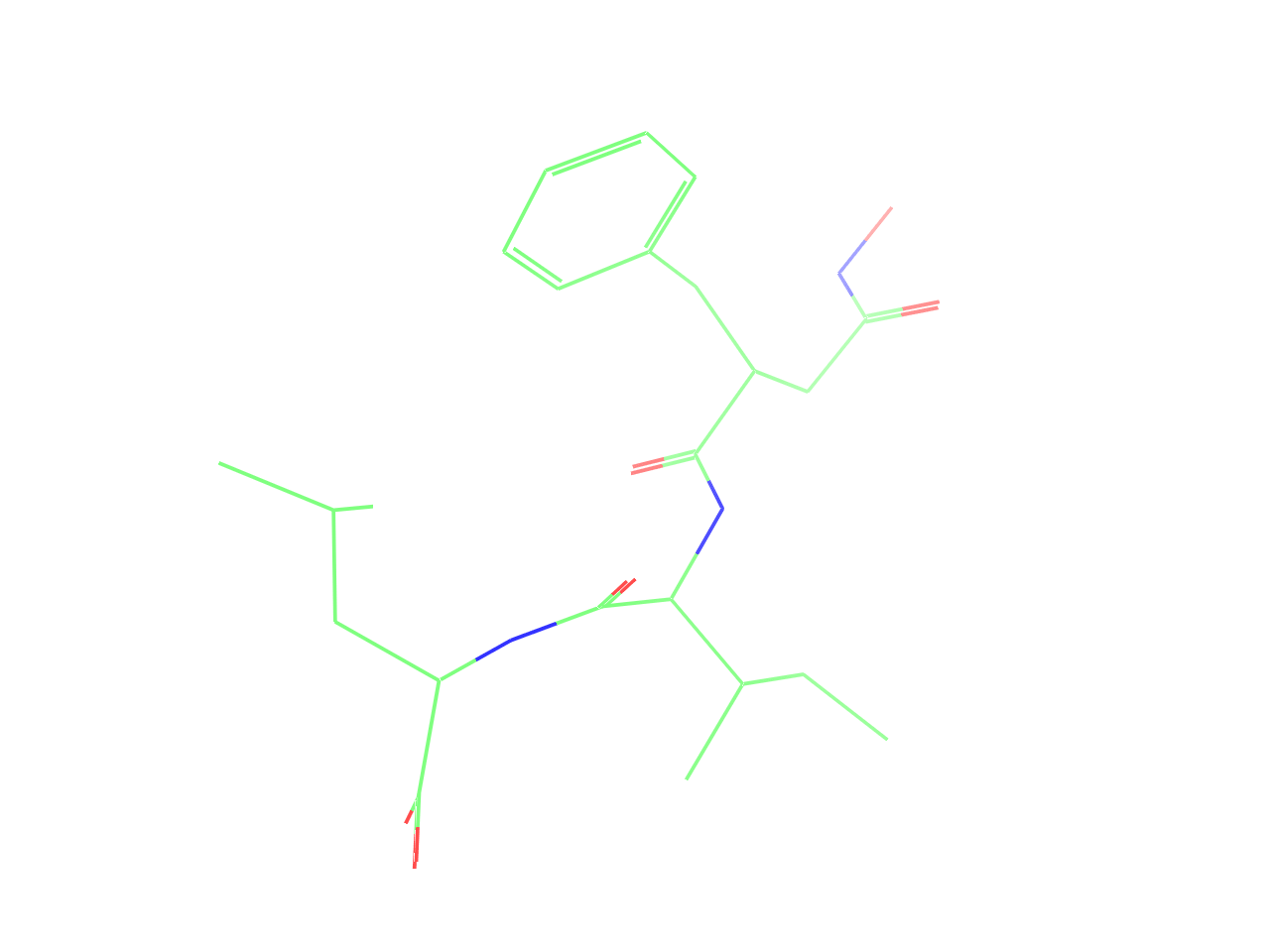
同様に ‘sticks‘(デフォルト), ‘mesh‘, ‘surface‘を指定すると以下のように見た目を変えることが可能です.
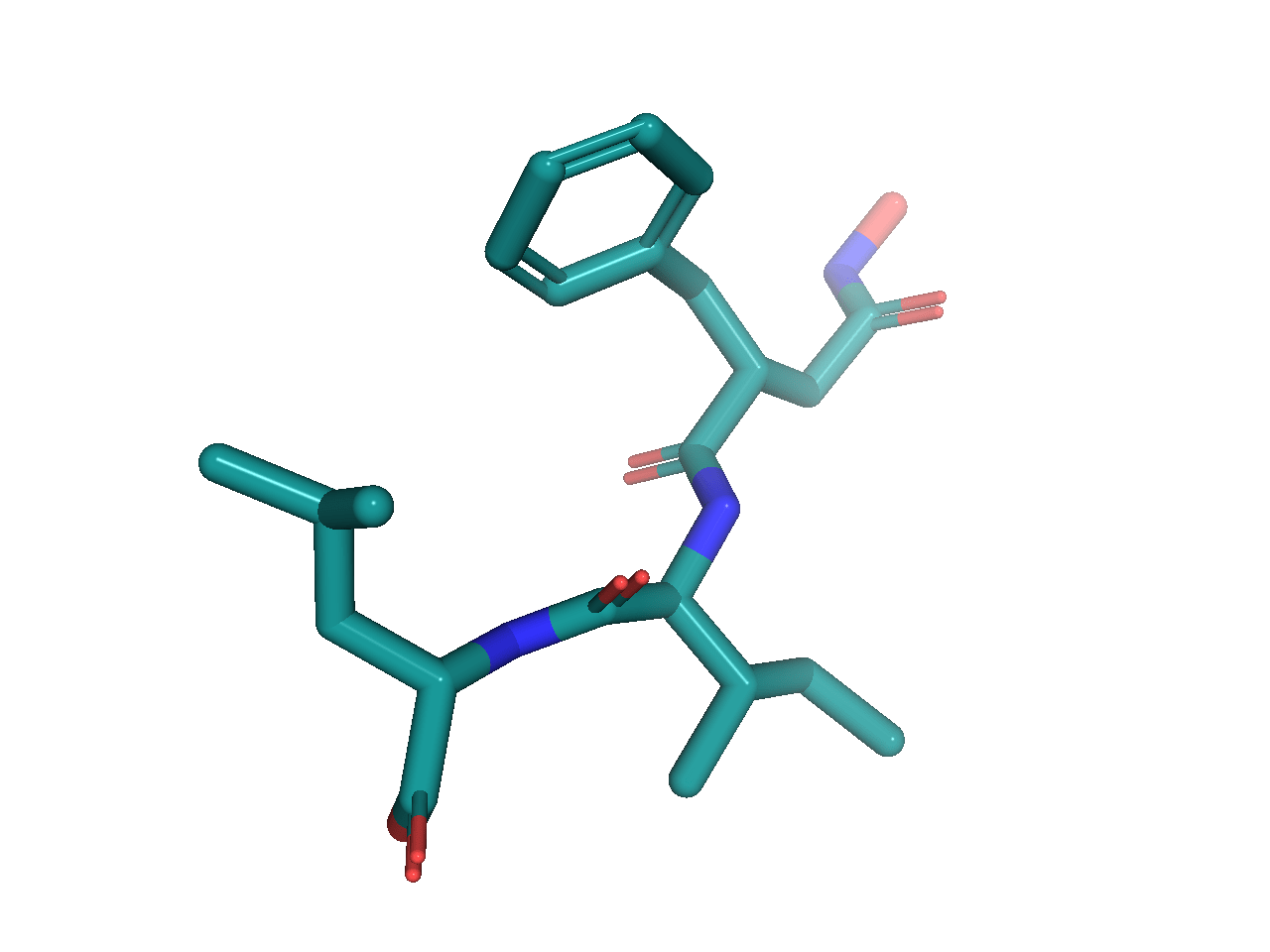
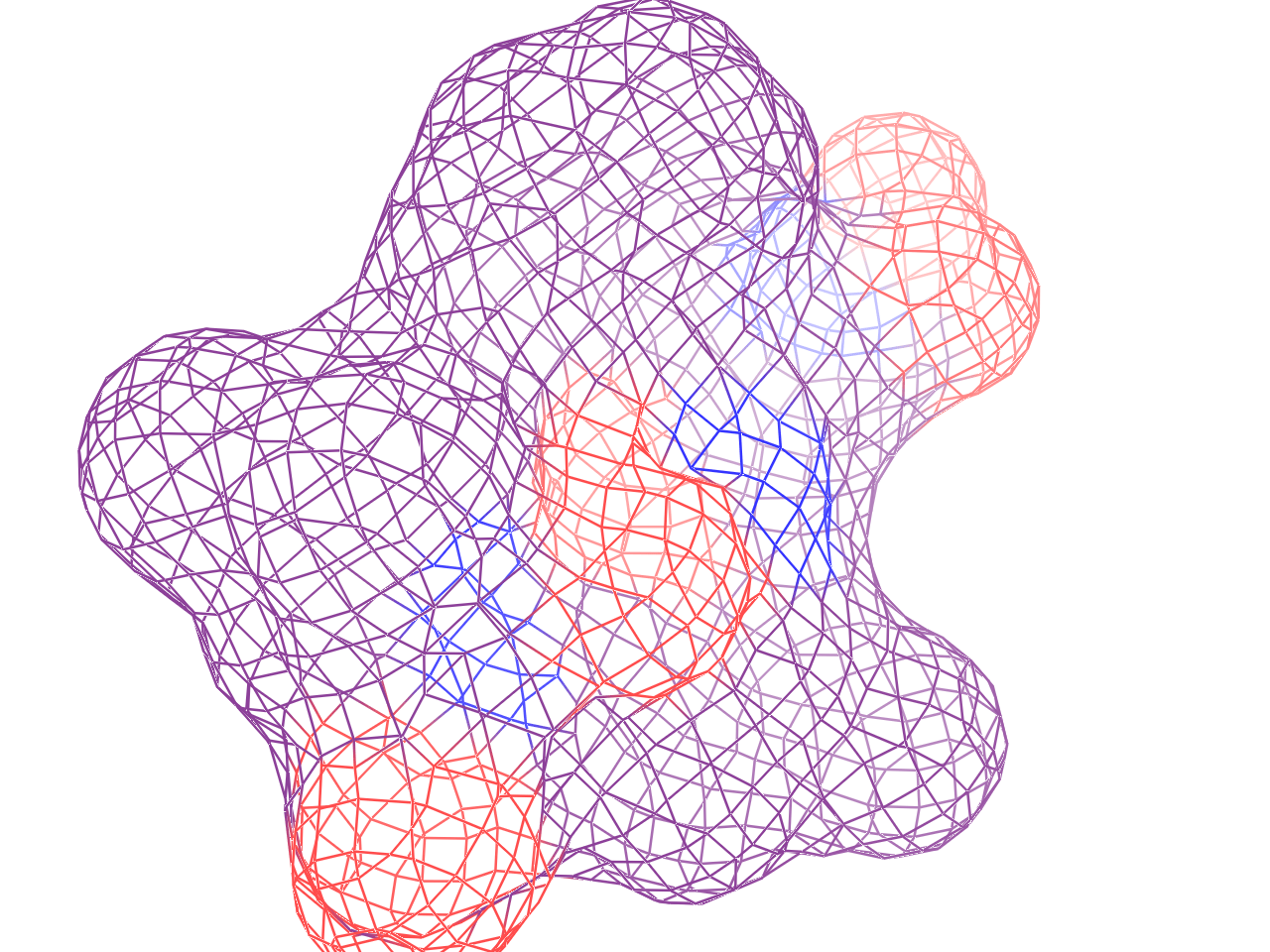
複数構造(コンフォマー)の描画
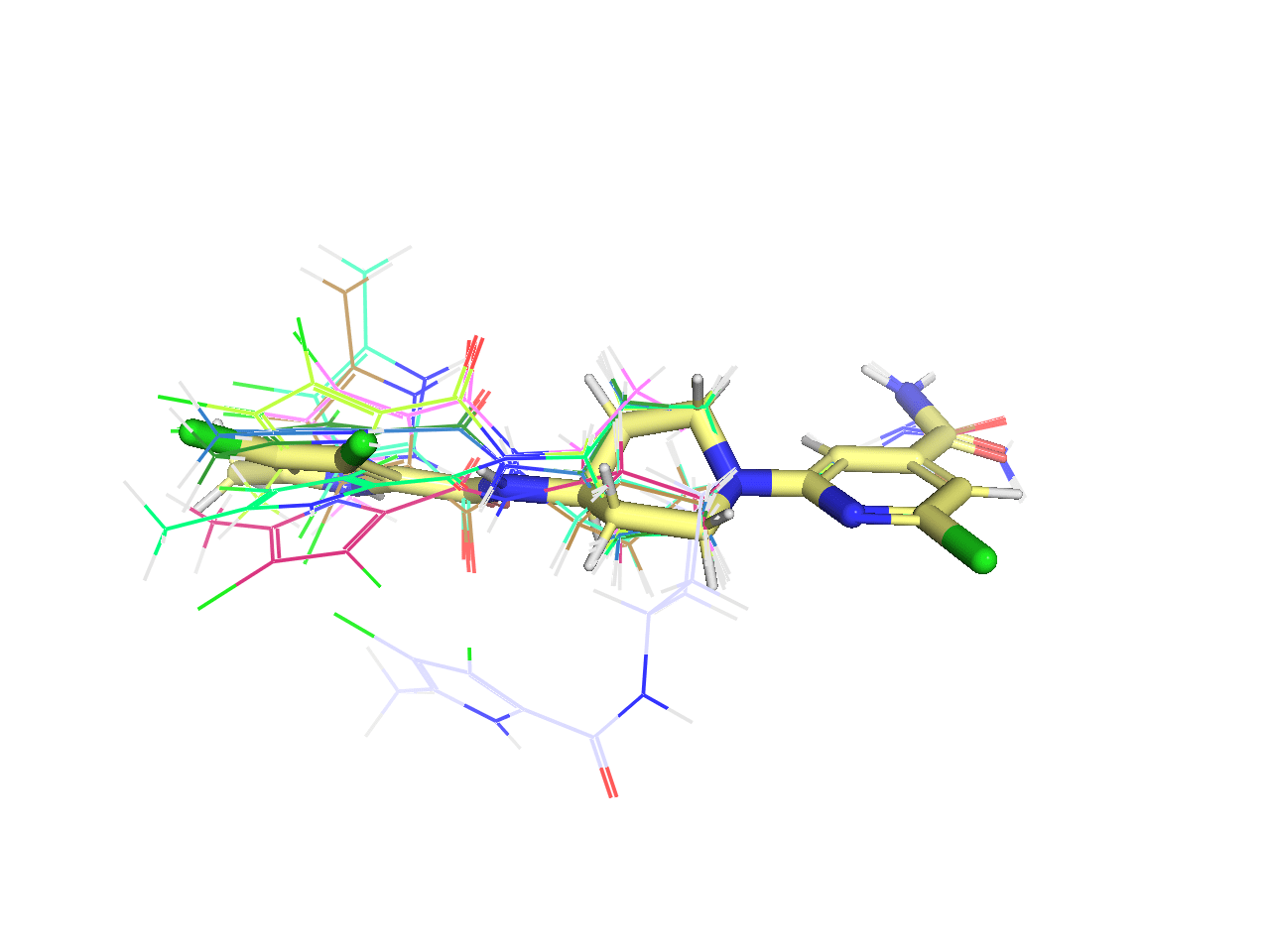
続いて分子の複数のコンフォマーを1つの画面に描画してみます.その際,ShowMolメソッドのShowOnlyオプションをFalseに設定することで構造の重ね描きが可能になります.
下記のコードでは上の構造の分子に対して,
- コンフォマーを10個生成
- 各構造をMMFFによって最適化
- 部分構造を生成し各コンフォマーを並べる
- PyMOL上にて描画
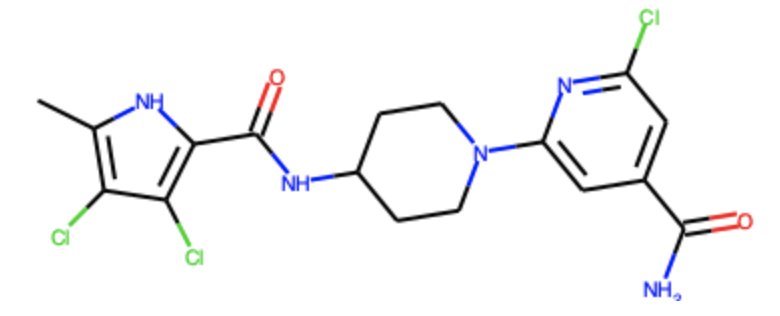
という手順を行っています.なお並べる鋳型構造としては下記のような2-アミノピリジン部位を用いました.
import copy
m = copy.deepcopy(mols[40])
m = Chem.AddHs(m)
# 1. コンフォマー発生
confs = AllChem.EmbedMultipleConfs(m, 10, pruneRmsThresh=1.0)
len(confs) # 10
# 2. MMFFによる最適化
for conf in confs:
AllChem.MMFFOptimizeMolecule(m, confId=conf)
# 3. 部分構造の生成とアライン
pat = Chem.MolFromSmiles('Nc1ncccc1')
match = m.GetSubstructMatch(pat)
match # (15, 18, 23, 22, 21, 20, 19)
AllChem.AlignMolConformers(m, atomIds=match)
# 4. PyMOL上にて描画
v.DeleteAll()
v.ShowMol(m, name='conf-0', confId=confs[0])
v.SetDisplayStyle('conf-0', 'sticks')
for i in range(1, len(confs)):
v.ShowMol(m, name='conf-'+str(i), confId=confs[i], showOnly=False)
v.SetDisplayStyle('conf-'+str(i), 'lines')
PyMOLスクリプトの利用
これまで,RDKitのPyMOLモジュールが与える機能を用いてJupyter Notebook上に分子を描画してきました.PyMOLの利点の1つとして,独自のスクリプトを利用することによる機能の拡張が挙げられます.例えば有志による様々なスクリプトが公開されており,煩雑なマウス操作をすることなく分子の操作が行えたりします.
RDKitのPyMOLモジュールでもserverコマンドを利用することで独自スクリプトなどを利用することが可能になります.以下の例では「.pymolrc」ファイルに「run ~/bin/pymolSample.py」などと書き込んでおき,「modStick」という分子を描画する関数を定義したスクリプトファイル(pymolSample.py)を事前にPyMOLに読み込んでおきます.
from pymol import cmd
def modStick(mol):
cmd.show("sticks", mol)
cmd.show("spheres", mol)
cmd.color("gray","elem C and "+mol)
cmd.set("stick_radius",0.07, mol)
cmd.set("sphere_scale",0.15, mol)
cmd.alter(mol+" and elem H", "vdw=0.75")
cmd.set("stick_color","black", mol)
cmd.set("dash_gap",0.01, mol)
cmd.hide("nonbonded", mol)
cmd.hide("lines", mol)
cmd.extend( "modStick", modStick );
上記のようにサンプルスクリプトを読み込んである状態でserver.doメソッドを用いると,自分で定義した「modStick」関数を呼び出すことが可能になります.
v.ShowMol(mols[40], name='mol')
v.server.do('modStick mol')
v.GetPNG(h=350)
分子の描画様式がデフォルトの’sticks’とは異なることがおわかり頂けると思います.
終わりに
今回は「RDKitからPyMOLを利用する」という話題について,RDKit.ChemのPyMOLモジュールの使い方を紹介してきました.
今回はPyMOLの利用の基本として,特にJupyter Notebook上で低分子を描画する方法について扱いました.この用途についてはPyMOLよりも「py3Dmolを使って化学構造をJupyter上で美しく表示する」という記事で紹介したpy3Dmolの方が使いやすいというのが個人的な印象です.
一方でPyMOLが最も利用される場面はタンパク質などの生体分子の描画です.次回はタンパク質やそのリガンド構造などを題材に,PyMOLらしい可視化について取り扱っていこうと思います.
コメント