異なる分子の立体構造を重ね合わせて眺めることで得られる知見が多くあります.これまで本ブログでは「RDKitによるコンフォマーの生成」という記事で,同じ分子のコンフォマーを重ね合わせて表示することを行いました.その際には鋳型となる原子の番号を指定することで重ね合わせの中心骨格を決めることができました.
それでは分子の立体的な性質は似ていても,構造の異なる分子の立体構造を重ね合わせるにはどうすればよいでしょうか?今回はこのような場合にRDKitで利用可能な方法を説明していきます.
異なる分子の立体構造重ね合わせ
「構造の異なる分子の立体構造の重ね合わせ」には以下のように2つの方法が考えられます.
- 共通構造を鋳型として重ね合わせる
- 性質の類似した部分構造を重ね合わせる
前者の方法はコンフォマーの重ね合わせと同じく,鋳型となる構造を指定する方法です.
- 自分で目で見て決める
- 最大共通構造(MCS)を求めて指定する
などにより鋳型を決めます.RDKitではAllChem.AlignMolで実行可能です.
一方で後者の方法では各原子が持つ何らかの性質を与えて,アルゴリズムによってどの部分構造が類似しているかを判断させる方法になります.
後者に分類されるOpen3D ALIGNは3次元構造の重ね合わせを行うためのオープンソースのプロジェクトで,原子の特徴を基にして最も類似した構造同士を一致させようする「教師なし学習」型の重ね合わせ法になります.RDKitに実装されているO3Aでは,
- MMFFの力場パラメーター
- CrippenのMolLogPとMolMRへの原子ごとの寄与
の2種類の重ね合わせ法が可能です.
分子の準備
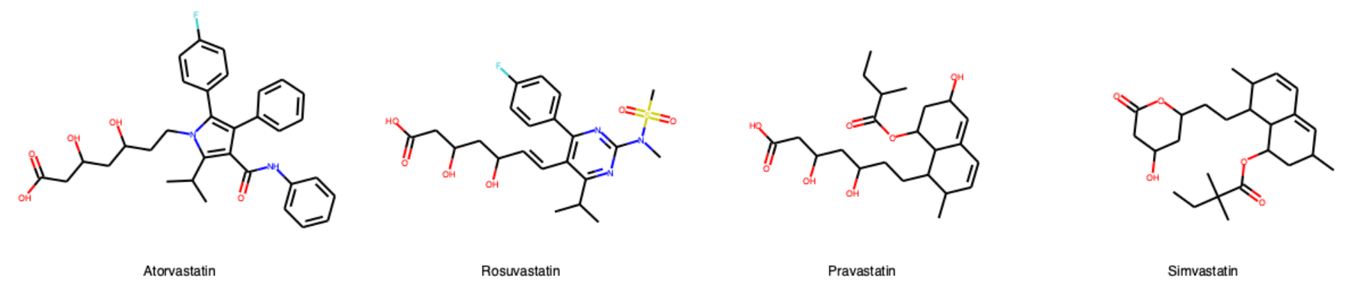
まずは必要なライブラリーのimportと分子の準備を行います.今回はいくつかのスタチン化合物を題材に3次元構造の重ね合わせを行ってみましょう.使用する分子は
の4つでそれぞれの3次元構造は下図のようにPubChemからSDFとして取得しました.
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, rdMolAlign, Draw, rdMolDescriptors
from rdkit.Chem.Draw import IPythonConsole
import py3Dmol
import copy
print(rdBase.rdkitVersion) # 2018.09.1
atorvastatin = Chem.MolFromMolFile('./Atorvastatin_3D.sdf')
rosuvastatin = Chem.MolFromMolFile('./Rosuvastatin_3D.sdf')
pravastatin = Chem.MolFromMolFile('./Pravastatin_3D.sdf')
simvastatin = Chem.MolFromMolFile('./Simvastatin_3D.sdf')
mols = [atorvastatin, rosuvastatin, pravastatin, simvastatin]
用いる分子の2次構造は下記の4つです.
鋳型を用いた重ね合わせ
AlignMolメソッドではコンフォマーの重ね合わせと同様にatomMapをオプションに与えます.異なる点は分子が異なるため,鋳型とマッチする原子の番号の対応を与える必要があります.下のコードではzipを用いています.
鋳型にする部分構造は側鎖のヒドロキシカルボン酸部位が適切だと思われます.3次元構造の表示にはpy3Dmolを用いています.
template = Chem.MolFromSmiles('O=CCC(O)CCO')
align_mols = [copy.deepcopy(m) for m in mols]
view = py3Dmol.view(width=400, height=300)
prbMatch = align_mols[1].GetSubstructMatch(template)
refMatch = align_mols[0].GetSubstructMatch(template)
AllChem.AlignMol(prbMol=align_mols[1], refMol=align_mols[0],
atomMap=list(zip(prbMatch, refMatch)))
view.addModel(Chem.MolToMolBlock(align_mols[0]), 'sdf')
view.addModel(Chem.MolToMolBlock(align_mols[1]), 'sdf')
view.setStyle({'stick': {}})
view.zoomTo()
view.setBackgroundColor('0xeeeeee')
view.show()
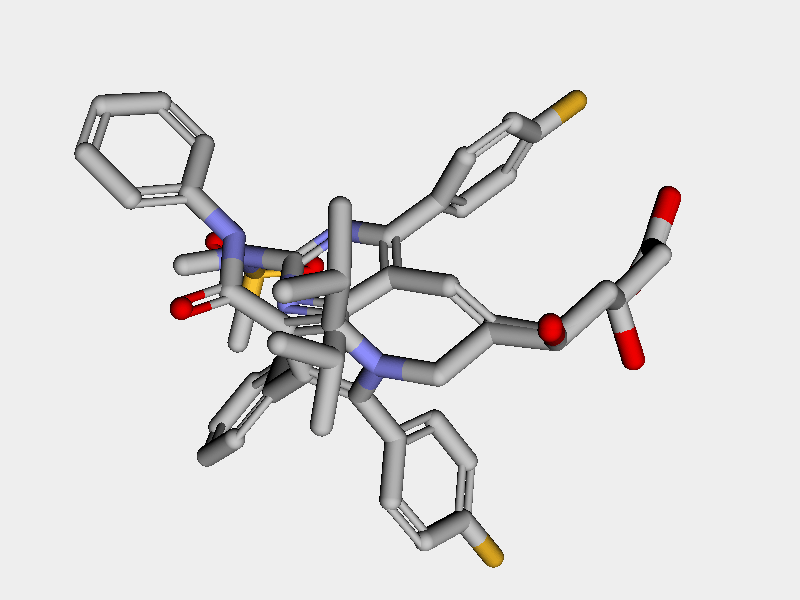
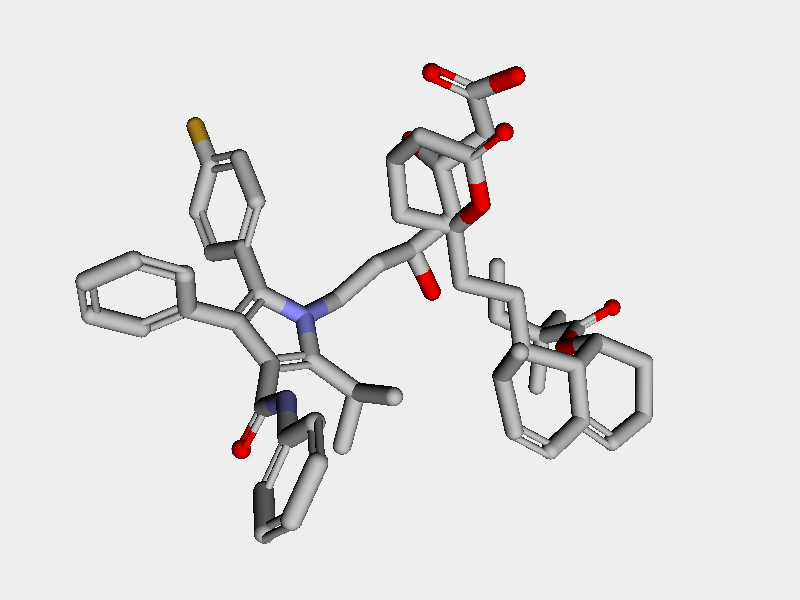
アトルバスタチンとロスバスタチンの重ね合わせの結果を下に示します.鋳型部位は完璧ですが,中心のp-F-フェニルなどがズレているのがやや残念です.
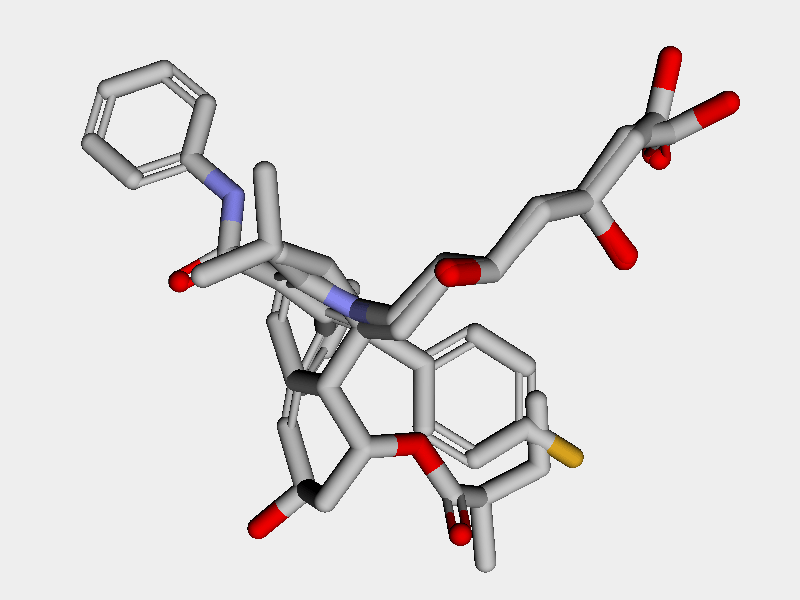
残りの2つの分子は下のようになりました.いずれもイマイチです.
O3Aを用いた重ね合わせ
O3A.Score()
RDKitのOpen3D ALIGNの実装では,まず基となる分子や参照分子などの情報からO3Aオブジェクトを作成します.作成したオブジェクトを用いて,AlignやScoreといったメソッドを利用して,分子の重ね合わせやRMS値などの取得が可能になります.
それでは先ほど述べた2つの方法によってO3Aオブジェクトを作成してみましょう.
Crippenの寄与率を用いる方法
AllChem.GetCrippenO3A(prbMol, refMol, prbCrippenContribs, refCrippenContribs)
まずはCrippenらによる原子毎の寄与度を用いる方法です.
- rdMolDescriptors._CalcCrippenContribsで寄与度を計算
- GetCrippenO3AでO3Aオブジェクトを作成
- Alignメソッドで重ね合わせ
- py3Dmolで描画
という手順で下記のコードは行っています.
crippen_mols = [copy.deepcopy(m) for m in mols]
crippen_contribs = [rdMolDescriptors._CalcCrippenContribs(m) for m in crippen_mols]
crippen_o3as = [AllChem.GetCrippenO3A(prbMol=crippen_mols[i], refMol=crippen_mols[0],
prbCrippenContribs=crippen_contribs[i],
refCrippenContribs=crippen_contribs[0])
for i in range(1, len(crippen_mols))]
view = py3Dmol.view(width=400, height=300)
view.addModel(Chem.MolToMolBlock(crippen_mols[0]), 'sdf')
crippen_o3as[0].Align()
view.addModel(Chem.MolToMolBlock(crippen_mols[1]), 'sdf')
view.setStyle({'stick': {}})
view.zoomTo()
view.setBackgroundColor('0xeeeeee')
view.show()
先ほどの場合よりも中心骨格を含めて一致しているのが見てとれます.
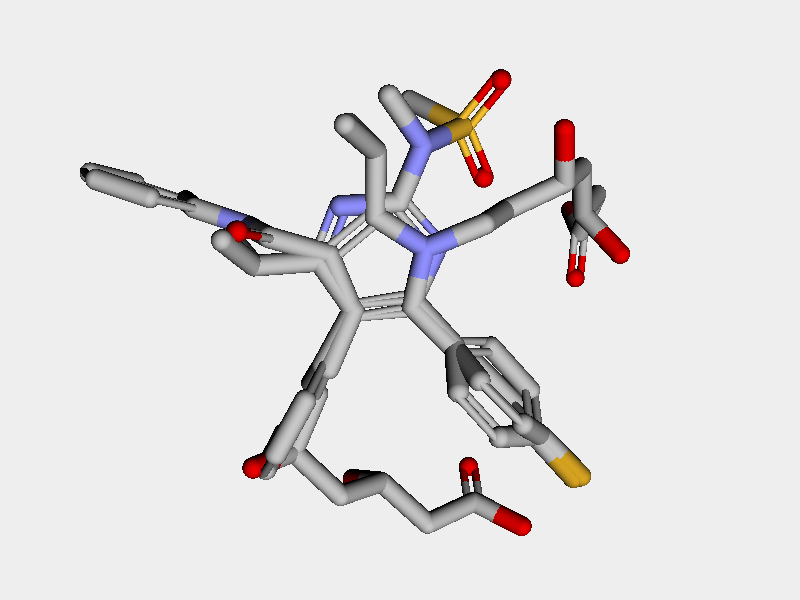
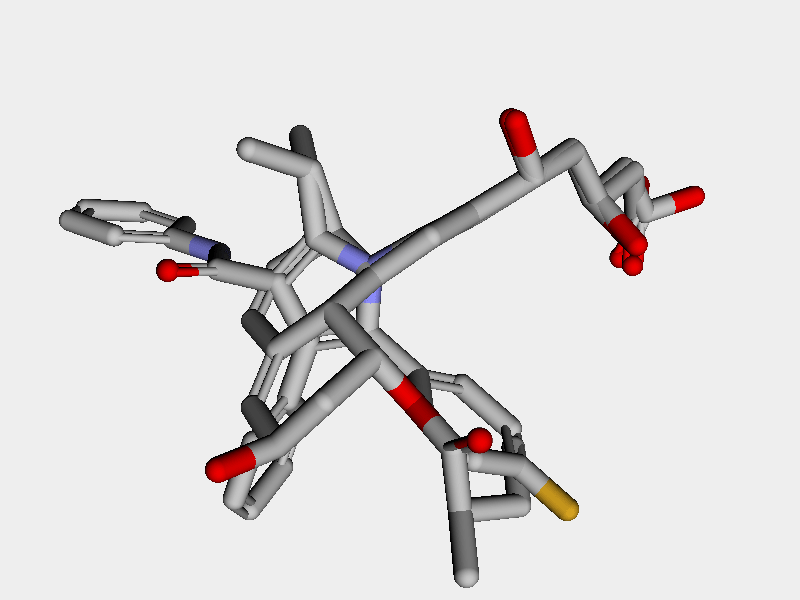
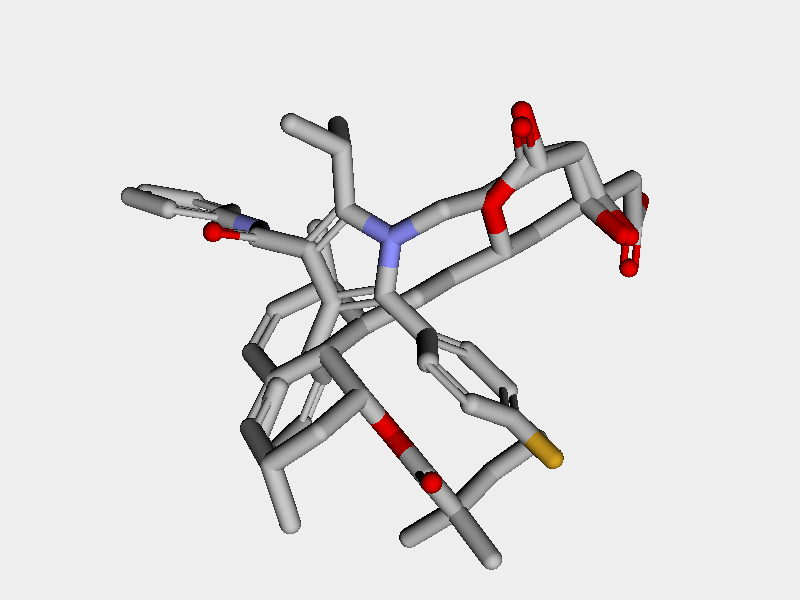
MMFFの力場パラメーターを用いる方法
MMFFのパラメーターを利用する場合もほとんど同じように実行可能です.下記のコードでは
- MMFFGetMoleculePropertiesで原子毎のパラメーターの取得
- GetO3Aメソッドを用いてO3Aオブジェクトを作成
- Alignメソッドで重ね合わせ
- py3Dmolで描画
という順番で処理を行っています.
mmff_mols = [copy.deepcopy(m) for m in mols]
mmff_params = [AllChem.MMFFGetMoleculeProperties(m) for m in mmff_mols]
mmff_o3as = [AllChem.GetO3A(prbMol=mmff_mols[i], refMol=mmff_mols[0],
prbPyMMFFMolProperties=mmff_params[i],
refPyMMFFMolProperties=mmff_params[0])
for i in range(1,len(mmff_mols))]
view = py3Dmol.view(width=400, height=300)
view.addModel(Chem.MolToMolBlock(mmff_mols[0]), 'sdf')
mmff_o3as[0].Align()
view.addModel(Chem.MolToMolBlock(mmff_mols[1]), 'sdf')
view.setStyle({'stick': {}})
view.zoomTo()
view.setBackgroundColor('0xeeeeee')
view.show()
MMFFパラメータを用いた場合もよく重なっていると言えるのではないでしょうか.
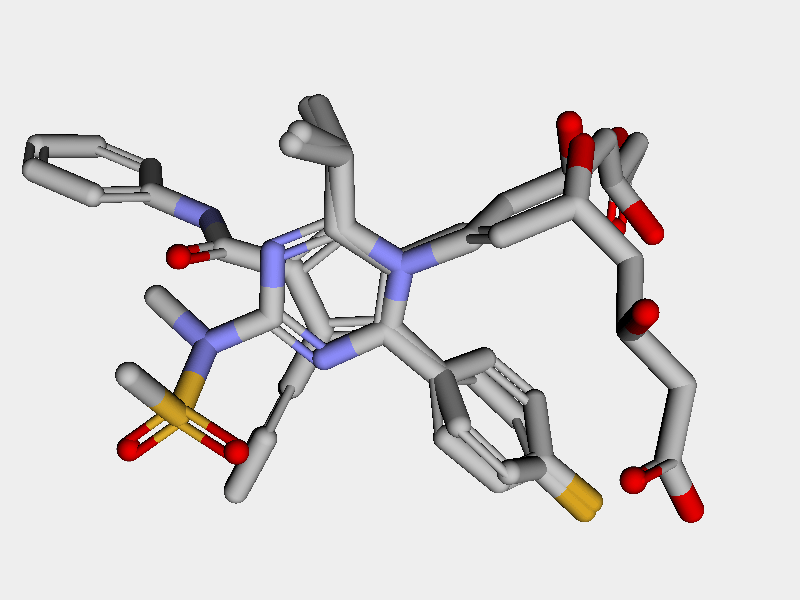


最後にCrippenとMMFFを用いた場合のRMSDスコアを計算してみましょう.
print('--- Crippen ---')
for m in crippen_o3as:
print('{:.3f}'.format(m.Score()))
print('--- MMFF ---')
for m in mmff_o3as:
print('{:.3f}'.format(m.Score()))
--- Crippen --- 108.898 100.496 60.506 --- MMFF --- 108.297 124.020 87.611
終わりに
今回は「RDKitでOpen3DALIGNを用いた立体構造の重ね合わせ」という話題について
- 鋳型構造を指定して重ね合わせを行う方法
- Open 3D ALIGN(O3A)を利用して行う方法
の2通りについて見てきました.特にO3Aではこちらで鋳型を設定することなく,重ね合わせ方法を決める「教師なし学習」型の方法であることを説明しました.
コメント
いつも勉強させて頂いております。
分子を2つ重ねあわせて、主に原子間の距離の違いや角度の違いを比較したいと考え、最近RDkitを触り始めました。こちらのブログはすごく丁寧にご説明頂いておりますので大変助かっております。
上記用途のため、2つ重ね合わせた後に、片方の分子だけを移動したり回転したりしてみたいのですが、そのようにプログラミングすることは可能でしょうか?
今までプログラミングすらやったことがなかったので、なかなかどう触れば思い通りに出来るかわかりません。ご回答頂ければ幸いです。宜しくお願い致します。
コメントありがとうございます.
ご質問に関してですか,RDKitにはそのものずばりのメソッドは実装されていないようですので,ご自分でコードを書くことになります.
「RDKitによるコンフォマーの生成」という記事で,触れましたが分子の3次元座標を取得することができます.
そこで分子の座標をnumpy配列に格納し,行列操作を行うことで座標の(平行)移動・回転を行います.その後で移動させた座標をConformer.SetAtomPositionメソッドで設定することで,移動させたコンフォマーが得られると思います.SetAtomPositionに関してブログ内で説明した記事はありませんが,座標を設定したい原子IDとその座標の組み合わせを引数にとり,
SetAtomPosition(atomId, (x, y, z))
のように用います.
座標の平行移動については全ての原子を一定方向に動かすだけですので理解しやすいと思います.回転操作については「回転行列」と呼ばれるものを使います.例えばこちらのページなどを参照してみてください.