「psi4で遷移状態最適化:計算化学における虚振動と化学構造」という記事では,python用量子化学計算ライブラリであるPsi4を用いた遷移状態構造の最適化について学び,唯一の虚振動を可視化することで望みの構造を繋ぐ遷移状態であることを確かめました.
今回はIRC計算と呼ばれる計算を実行することで,原系がどのような経路で遷移状態から生成系へと至るかを調べていきます.また得られた反応経路をPyMOLを用いて可視化していきます.

固有反応座標とは
化学反応を議論するにあたって重要となるのは原料と生成物に対応する安定構造(minima)と,遷移状態に対応する鞍点(saddle point)です.遷移状態を求めた後は,実際に原系と生成系を繋ぐ遷移状態であることを確認する必要があります.
前回行ったような振動モードの目視によっても「正しい」遷移状態であることが示唆されますが,厳密に確認するには遷移状態から対応する安定構造へと繋がるミニマムエネルギーパス(Miniumum Energy Path)を求める必要があります.固有反応座標(Intrinsic Reaction Coordinate, IRC)はミニマムエネルギーパスのmass-weightedな座標です.
イメージとしては虚振動で表される振動モードに沿って,山頂(遷移状態)から麓(安定構造)へと下っていく様子に似ています.今回は得られた遷移状態からPsi4を用いてIRCを求める方法を見ていきます.
The path of chemical reactions – the IRC approach
Acc. Chem. Res. 1981, 14, 363–368.
Intrinsic reaction coordinate: Calculation, bifurcation, and automated search
Int. J. Quant. Chem. 2015, 115, 258-269.
Psi4を用いたIRC計算
今回も以下の環境・設定で「.py」ファイルをターミナルから動かすことで計算を行っていきます.各自の環境に合わせて,メモリ・スレッド数などを調節してください.題材としては前回と同様に2−ピリドンと2−ヒドロキシピリジンの互変異性を例とします.
from __future__ import annotations
import sys
import time
import pathlib
import numpy as np
import psi4
print(f'python version:\n{sys.version}')
# python version:
# 3.9.7 (default, Sep 16 2021, 13:09:58)
# [GCC 7.5.0]
print(f'numpy version:\t{np.__version__}')
# numpy version: 1.21.4
print(f'psi4 version:\t{psi4.__version__}')
# psi4 version: 1.4.1
filename = pathlib.Path(__file__)
logfile = filename.with_suffix('.log').name
psi4.set_output_file(logfile)
psi4.set_memory('14GB')
psi4.set_num_threads(10)
IRC計算の設定
psi4.set_options({ ‘optking__irc_direction’: direction})
IRC計算を実行するにはopt_typeオプションで「irc」を指定します.遷移状態から前方(forward),後方(backward),どちらの方面に下っていくかをoptkingモジュールのirc_directionで指定します.
なおどちらを原系に捉えるかは見方によって変わりますので,psi4が前方と考える方向と我々が考える方向が一致するとは限りません.いずれにせよ,IRC計算を行う際は遷移状態が繋ぐ二つの構造が知りたいので,前方・後方どちらの計算も行っていきます.
また公式サイトではIRC計算では最適化の終了判定を厳しめ(gau_tight)を利用することが推奨されていますので,これに従って計算していきます.前回HF/STO-3Gで求めた遷移状態を初期構造として,両方向に関してIRC計算を行うコードが以下になります.なお今回は設定していませんが,曲がりくねった道は大股では歩きにくいように,場合によってはIRC計算の歩幅(irc_step_size)を小さくする必要があります.
geom = '''
0 1
C -1.073986885437 -1.170578359942 0.000002755738
C 0.953021017198 0.140745143377 -0.000003061297
C 0.196029366481 1.360797354321 -0.000001202096
C -1.155622586106 1.236381947214 0.000002880952
C -1.818434996700 -0.031125507024 0.000005467599
H -1.510894241728 -2.166796480067 0.000004269828
H 0.691726656323 2.319296967258 -0.000002742435
H -1.770734621411 2.131633416218 0.000004678262
H -2.895472126141 -0.076640107909 0.000009377672
O 2.177846935050 -0.264655641303 -0.000003962413
H 1.432324292904 -1.412270186206 -0.000002894660
N 0.288380859812 -1.071403950594 -0.000002249524
'''
level = 'hf/sto-3g'
directions = ['forward', 'backward']
for direction in directions:
psi4.set_options({'geom_maxiter': 300,
'opt_type': 'irc',
'full_hess_every': 10,
'optking__g_convergence': 'gau_tight',
'optking__irc_direction': direction})
mol: psi4.core.Molecule = psi4.geometry(geom)
psi4.frequency(level, molecule=mol)
psi4.optimize(level, molecule=mol)
print(f'{direction} structure:\n{mol.save_string_xyz()}')
IRC計算のログファイルには以下のような計算結果が記録されています(「~」(チルダ)で抽出すると便利です).これらの結果がIRCの各ステップにおける構造最適化の経過を示しています.
今回の場合は,
- 1ステップ目が3回
- 2ステップ目が3回
で最適化が終了し,エネルギーはそれぞれ-317.39070036と-317.40017152 a.u.であるとわかります.
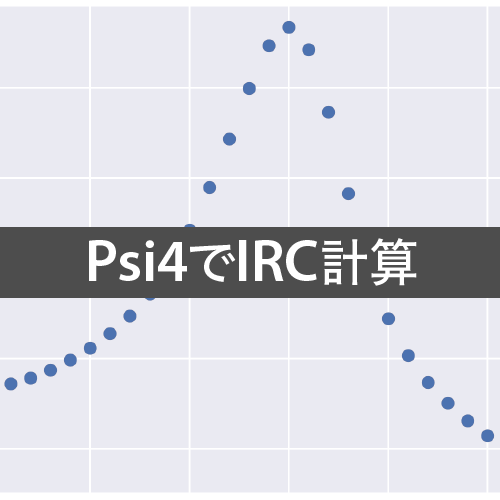
@IRC 1 1 -317.39068274 -4.07e-03 o 3.55e-03 6.59e-04 1.07e-02 1.71e-03 ~ @IRC 1 2 -317.39070800 -2.53e-05 o 1.05e-04 1.91e-05 2.50e-04 5.97e-05 ~ @IRC 1 3 -317.39070036 7.63e-06 o 2.10e-06 * 3.68e-07 * 9.07e-06 * 1.31e-06 * ~ @IRC 2 1 -317.40011444 -9.41e-03 o 6.24e-03 9.66e-04 1.63e-02 2.38e-03 ~ @IRC 2 2 -317.40018113 -6.67e-05 o 2.88e-04 4.65e-05 6.41e-04 1.09e-04 ~ @IRC 2 3 -317.40017152 9.61e-06 o 4.38e-06 * 7.90e-07 * 1.28e-05 * 2.17e-06 * ~ @IRC 3 1 -317.41124935 -1.11e-02 o 7.71e-03 1.08e-03 1.71e-02 2.37e-03 ~ ・・・以下略・・・
これらのエネルギー値を取り出してプロットすることで以下のようなIRCに沿ったエネルギー図が取得できます.
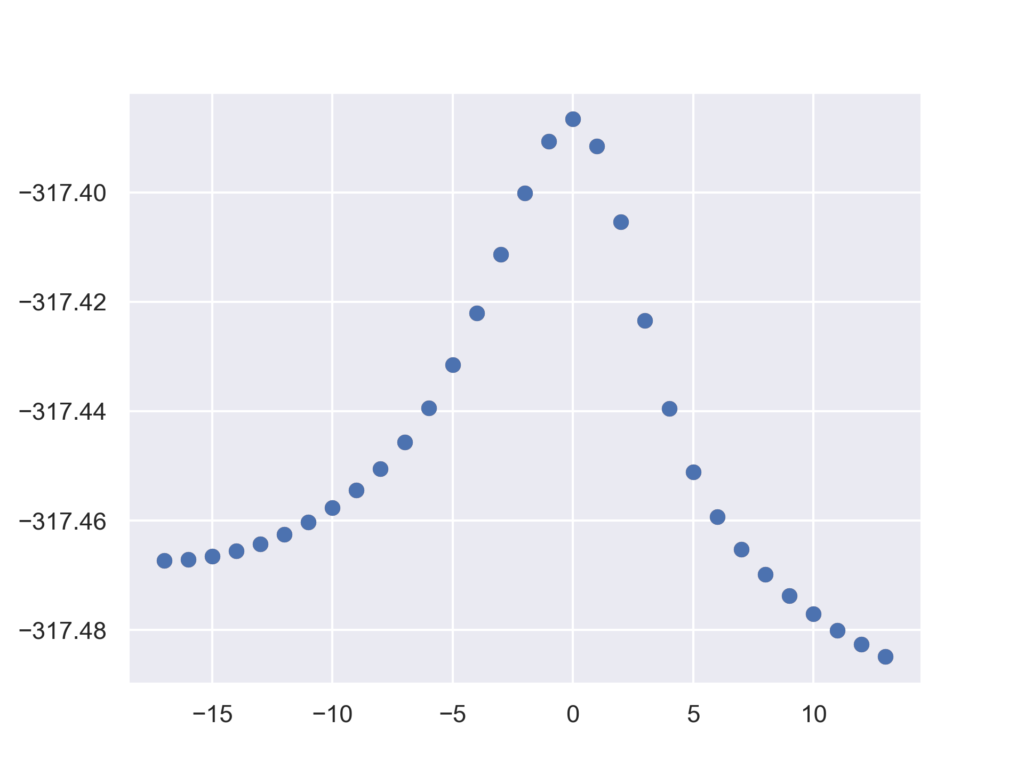
IRC計算結果の可視化
Psi4でIRC計算を行うと,irc_backward.xyzとirc_forward.xyzというIRC上の構造を含むxyz形式のファイルが生成されます.「Psi4で遷移状態最適化:計算化学における虚振動と化学構造」という記事では,振動モードに対応するXYZ形式のファイルを作成しVMDで可視化しました.今回はIRC計算で得られたXYZ形式のファイルをPyMOLで可視化してみます.
PyMOLによる固有反応座標に沿った反応経路の動画作成
irc_backward.xyzには
- 0番目(遷移状態)
- -1番目
- -2番目
- ・・・
という順番で構造が格納されています.
一方でirc_forward.xyzには
- 0番目(遷移状態)
- 1番目
- 2番目
- ・・・
という順番で構造が格納されています.そのためPyMOL上で各シーンの画像を書き出したうえで,
- backwardの場合は逆方向に動画を作成
- forwardの場合は順方向に動画を作成
- 作成した二つの動画を合体させる
という処理をすることで反応に関する動画が作成できます.今回は動画作成に関してはFFmpegというコマンドラインツールを使用することとします.
PyMOLへのxyzファイルの読み込み
PyMOLはタンパク質の可視化では素晴らしい表現力を示してくれますが,デフォルトで用意されている低分子の構造様式はあまり美しくありません.今回は「RDKitからPyMOLを利用する」という記事でも用いたように,描画方法を少しいじっています.
他にはOxford Protein Informatics Groupのブログにも低分子用の描画方法に関する記述があります.コードを見れば大体どんなことをしているかはわかると思いますので,みなさんも是非自分好みの方法にカスタマイズしてみてください.
以下のコードを「pymol_irc.py」として保存し,PyMOL中のターミナルで
run pymol_irc.py
として,pythonスクリプトを読み込みます.これで
- Viewerのサイズが「1200 x 800」に変更
- 背景色が「gray80」に変更
- 「load_xyz」という関数が使用可能
となります.
import pathlib
from pymol import cmd
cmd.viewport(1200, 800)
cmd.bg_color('gray80')
def modStick(mol):
cmd.show('sticks', mol)
cmd.show('spheres', mol)
cmd.color('gray','elem C and ' + mol)
cmd.set('stick_radius',0.07, mol)
cmd.set('sphere_scale',0.15, mol)
cmd.alter(mol + ' and elem H', 'vdw=0.75')
cmd.set('stick_color','black', mol)
cmd.set('dash_gap',0.01, mol)
cmd.hide('nonbonded', mol)
cmd.hide('lines', mol)
def load_xyz(file: str):
cmd.load(filename=file)
base_name = pathlib.Path(file).stem
if 'back' in base_name:
obj = 'back'
else:
obj = 'forward'
cmd.set_name(base_name, obj)
modStick(obj)
cmd.unbond('id 11', 'id 12')
cmd.unbond('id 11', 'id 10')
cmd.set('sphere_scale', 0.25, 'id 11')
cmd.color('pink', 'id 11')
cmd.extend('load_xyz', load_xyz)
load_xyz関数では
- irc_backward.xyzなら「back」,irc_forward.xyzなら「forward」というオブジェクトを作成して構造を読み込む
- 構造描画を改良したball-and-stickモデルで行う
- 遷移状態に関与する水素のみ,やや大きく,ピンク色に変更
という作業を行います.
スクリプトファイルを実行したら,続けてPyMOL上にて
load_xyz irc_backward.xyz load_xyz irc_forward.xyz
と入力することで以下のようにbackとforwardの二つの構造が読み込まれた状態になります.ピンク色で表現されている原子が遷移状態に関与する水素です.
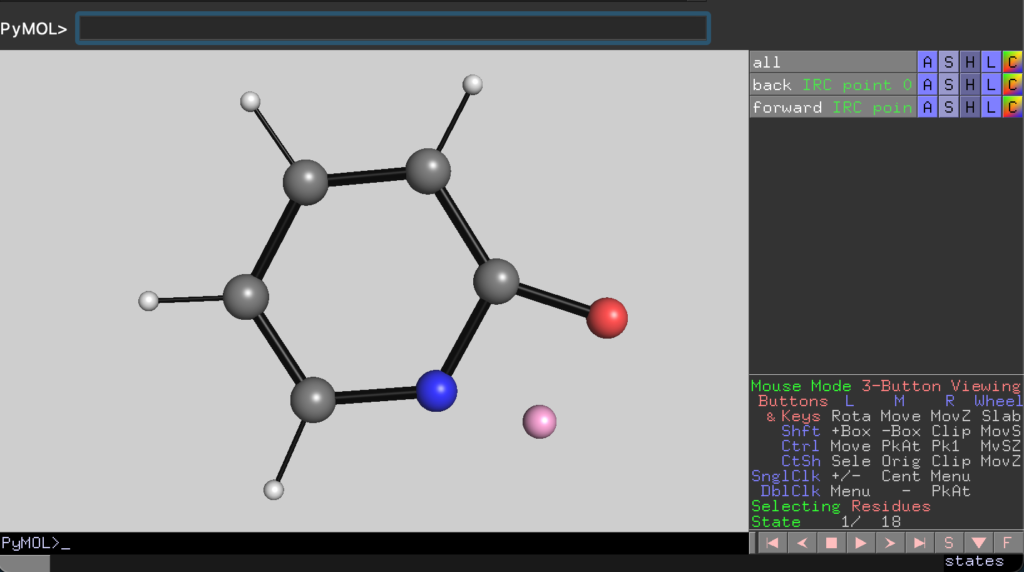
続いて「Movie > Program > State Loop > 1/3 Speed > no pause」などとすることでお好みで動画のコマ数を増やします.その後,「back」及び「forward」のみを選択(画面上に表示)した状態で,「File > Export Movie As > PNG Images」によってコマ送り用の画像を準備します.
今回は
- foward0001.png, forward0002.png, …
- back0001.png, back0002.png, …
という一連の画像を用意しました.
FFmpegによる動画作成
FFmpegはクロスプラットフォームの音声・動画ファイルの操作を行うコマンドラインツールです.
今回は
- forward0001.pngなどのファイルをmp4形式で動画化
- back0001.pngなどのファイルを逆順にmp4形式で動画化
- 作成した二つの動画を連結
- 10秒間ループし続ける動画を作成
という手順を踏んでいます.
ffmpeg -r 30 -i forward%04d.png -vcodec libx264 -pix_fmt yuv420p forward.mp4 ffmpeg -r 30 -i back%04d.png -vf reverse -vcodec libx264 -pix_fmt yuv420p back.mp4 ffmpeg -i back.mp4 -i forward.mp4 -filter_complex 'concat=n=2' irc.mp4 ffmpeg -stream_loop -1 -t 10 -i irc.mp4 irc_loop.mp4
今回の作業を通じて得られた動画は以下のようになりました.
終わりに
今回は「Psi4でIRC計算:固有反応座標で反応経路を求める」という話題について,
- IRCとは何か
- Psi4を用いたIRC計算の方法
- PyMOLを用いたXYZ形式の読み込み方法
などについて触れました.またIRCに沿った反応動画を作成するためにPyMOLとFFmpegを用いた動画作成方法について紹介しました.
今回はHF/STO-3Gという精度の低いレベルで求めた遷移状態でしたので,活性化エネルギーなどの絶対値には化学的意味はありませんが,遷移状態最適化からIRC計算という流れによってに化学反応を理解する流れはつかめたと思います.
これまで本ブログで扱ってきた量子化学計算は全て,気相中の孤立分子を扱っており溶液中の分子のモデルとしては必ずしも適していません.溶媒効果を考慮するためによく用いられるモデルとして,分極連続体モデル(Polarizable Continuum Model, PCM)があります.Psi4ではPCM Solverを通じて溶媒効果を取り入れることが可能です.次回はPCM Solverの扱い方について触れていきたいと思います.
>>次の記事:「Psi4で溶媒効果を考慮する:PCM Solverの使い方」
コメント