我々が興味のある分子のほとんどは3次元構造を有していますから,分子の立体構造の理解は大切です.
本ブログではこれまで,「RDKitによる3次元構造の生成」という記事ではRDKitを用いて立体構造をどのように発生させるかについて扱いました.その際いくつかのアルゴリズムについて学び,特にETKDGが優れていることを見てきました.
また「RDKitによるコンフォマーの生成」という記事では1つの分子から複数のコンフォマーを発生させる方法と,分子力学法を用いた構造の最適化方法を学習しました.
今回はある条件(制約)を課しながら立体構造を発生させたり最適化する方法を扱いたいと思います.地味ですがなかなか使い道のある方法だと思います.
ライブラリのインポート
いつも通りまずは必要なモジュールをimportしておきましょう.分子の可視化にはpy3Dmolを使います.
from rdkit import rdBase, Chem from rdkit.Chem import AllChem, Draw, rdMolTransforms from rdkit.Chem.Draw import IPythonConsole import py3Dmol print(rdBase.rdkitVersion) # 2019.03.3

制約つきで3次元構造を発生
AllChem.AdjustQueryProperties(mol, param)
AllChem.ConstrainedEmbed(mol, template)
ここではシクロヘキサンなどの飽和環状6員環化合物の立体配座を「イス形」で固定してみます.その際には当然「イス形」構造の座標が必要になります.
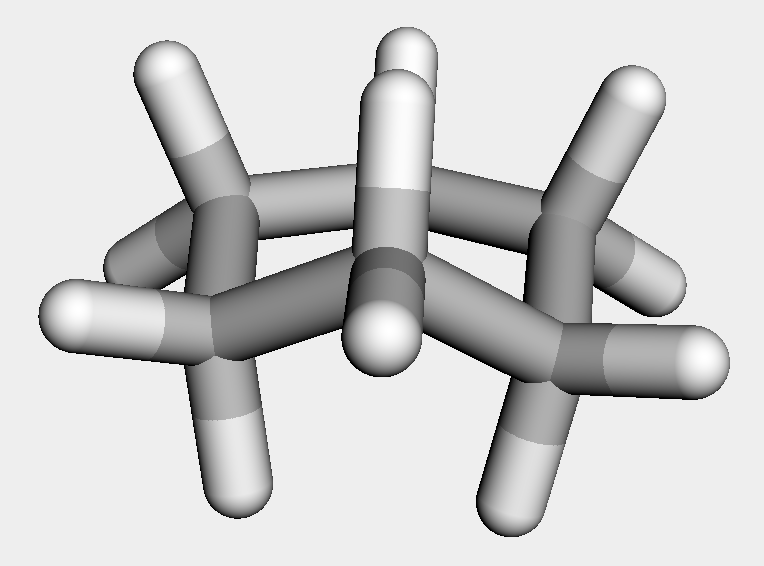
例えば適当にシクロヘキサンの3次元構造を求めた場合にはイス形になりませんでした.(ランダム要素もありますので,みなさんが試された場合にはイス形構造が得られることもあると思います.)
ch = Chem.MolFromSmiles('C1CCCCC1')
ch_h = AllChem.AddHs(ch)
AllChem.EmbedMolecule(ch_h)
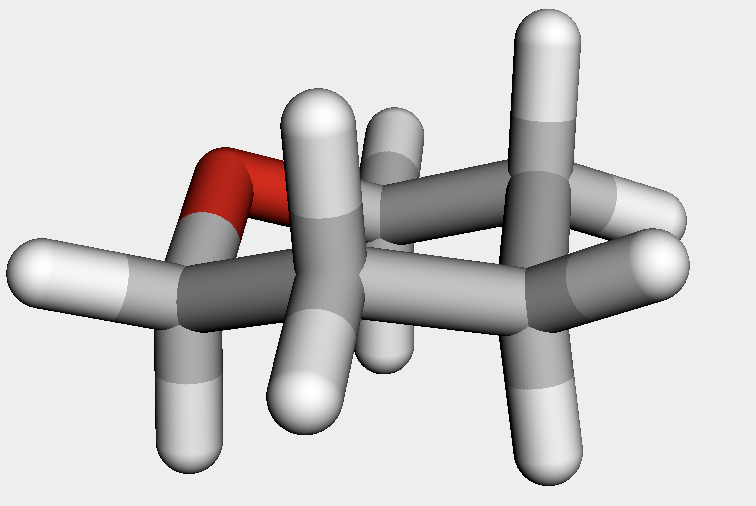
一方でテトラヒドロピランの立体配座は綺麗なイス形となりました.そこで,今回はこの構造を鋳型としていくつか6員環構造の配座を発生させてみることとします.
thp = Chem.MolFromSmiles('O1CCCCC1')
thp_h = AllChem.AddHs(thp)
AllChem.EmbedMolecule(thp_h, AllChem.ETKDGv2())
テンプレートの作成
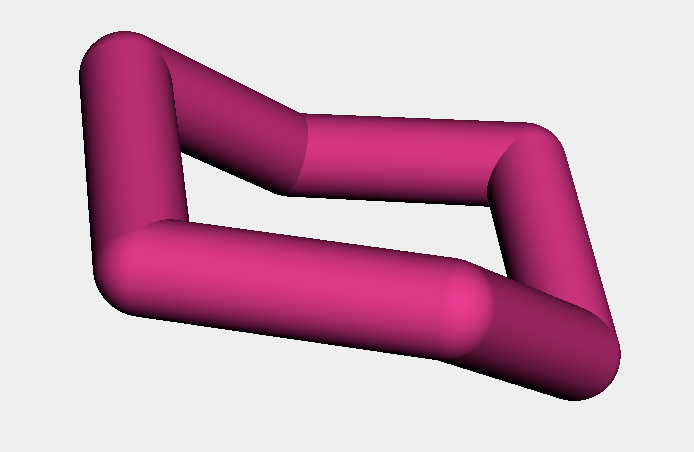
AllChem.ConstrainedEmbedに用いる鋳型の作成はAllChem.AdjustQueryPropertiesを用いて行います.その際,今回のように興味があるのが原子の種類を問わずに座標だけである場合にはmakeAtomsGenericをTrueに設定します.
## THPのイス形構造を基にしてテンプレートを作成 param = AllChem.AdjustQueryParameters() param.makeAtomsGeneric = True chair = AllChem.AdjustQueryProperties(AllChem.RemoveHs(thp_h), param)
このようにして作成した鋳型は原子の種類が「*」となっていることがMOLブロックからわかります.
RDKit 3D
6 6 0 0 0 0 0 0 0 0999 V2000
1.1092 -0.7166 0.7014 * 0 0 0 0 0 0 0 0 0 0 0 0
1.4838 0.3508 -0.1505 * 0 0 0 0 0 0 0 0 0 0 0 0
0.3228 1.3359 -0.3370 * 0 0 0 0 0 0 0 0 0 0 0 0
-0.8989 0.6047 -0.8974 * 0 0 0 0 0 0 0 0 0 0 0 0
-1.1955 -0.6470 -0.0688 * 0 0 0 0 0 0 0 0 0 0 0 0
0.0813 -1.4808 0.0972 * 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0
2 3 1 0
3 4 1 0
4 5 1 0
5 6 1 0
6 1 1 0
V 1 *
V 2 *
V 3 *
V 4 *
V 5 *
V 6 *
M END
py3Dmolでは紫色の原子で表示されています.
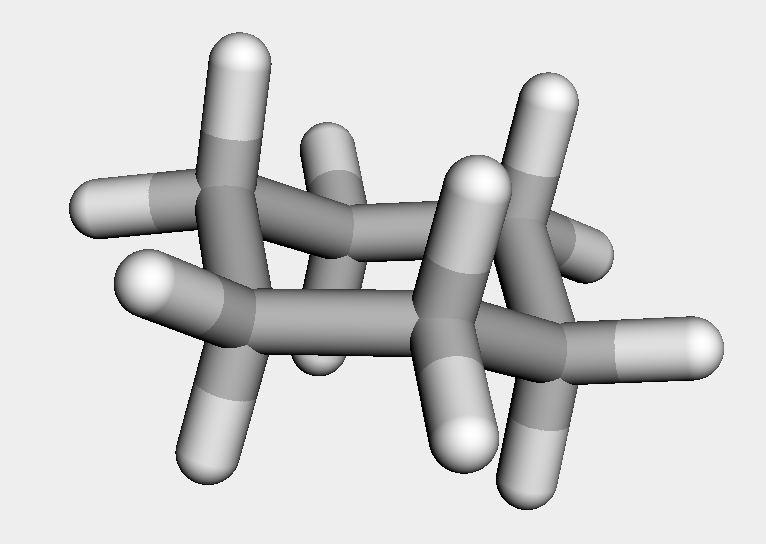
作成した鋳型を用いてConstrainedEmbedメソッドで立体構造を発生させてみます.まずはシクロヘキサンで試してみます.
chex_h = AllChem.AddHs(ch) AllChem.ConstrainedEmbed(chex_h, chair)
先ほどとは異なり,きれいなイス形配座が得られました.
続いてフェニルシクロヘキサンと1,4-ジオキサンでも行ってみます.
## Ph-シクロヘキサン
ph_chex = Chem.MolFromSmiles('c1ccccc1C1CCCCC1')
ph_chex_h = AllChem.AddHs(ph_chex)
AllChem.ConstrainedEmbed(ph_chex_h, chair)
## 1,4-ジオキサン
diox = Chem.MolFromSmiles('O1CCOCC1')
diox_h = AllChem.AddHs(diox)
AllChem.ConstrainedEmbed(diox_h, chair)
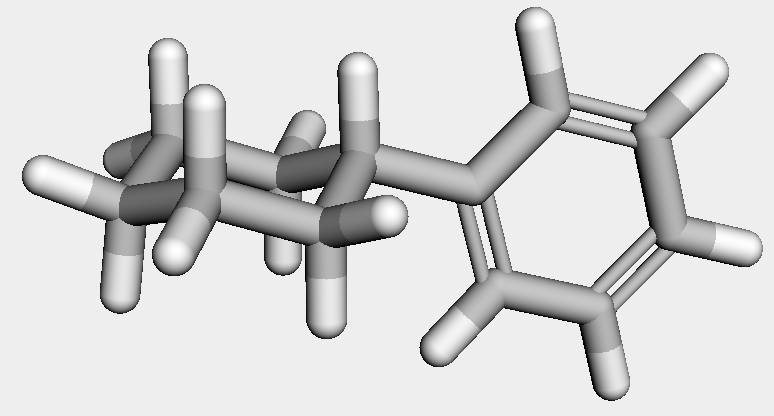
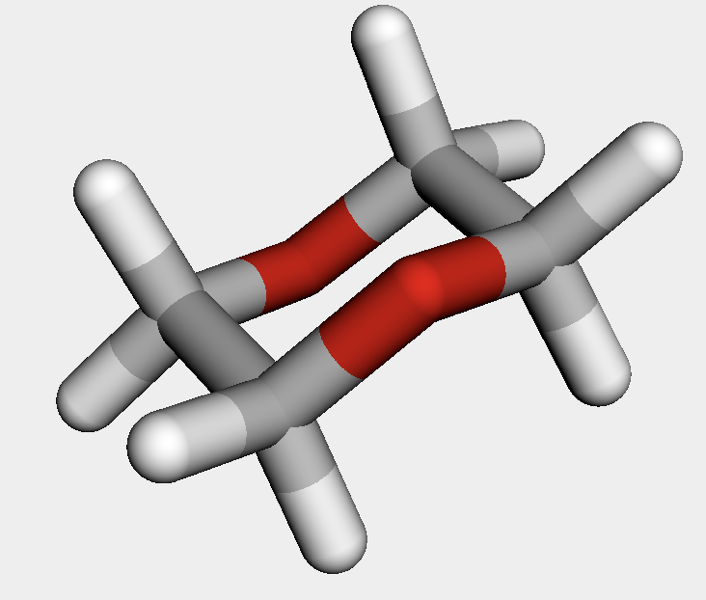
先ほど述べたように,鋳型の原子タイプを一般化したため,どのような原子で6員環が構成されていても同様に扱うことが可能です.
コンフォマーに二面角の制約を設定
rdMolTransforms.SetDihedralDeg(conf, x1,x2,x3,x4, ang)
コンフォマーオブジェクトの二面角(や結合長・角度など)に関する設定はChem.rdMolTransformsモジュールで行えます.
この例では1-クロロ-2-フルオロエタンを例として,二面角に制約を加えて立体配座を発生させてみます.
mol = Chem.MolFromSmiles('ClCCF')
mol_h = AllChem.AddHs(mol)
AllChem.EmbedMolecule(mol_h)
conf = mol_h.GetConformer(-1)
print(rdMolTransforms.GetDihedralDeg(conf, 0,1,2,3))
# -60.001693797737026
何も制約を加えないと60度でした.
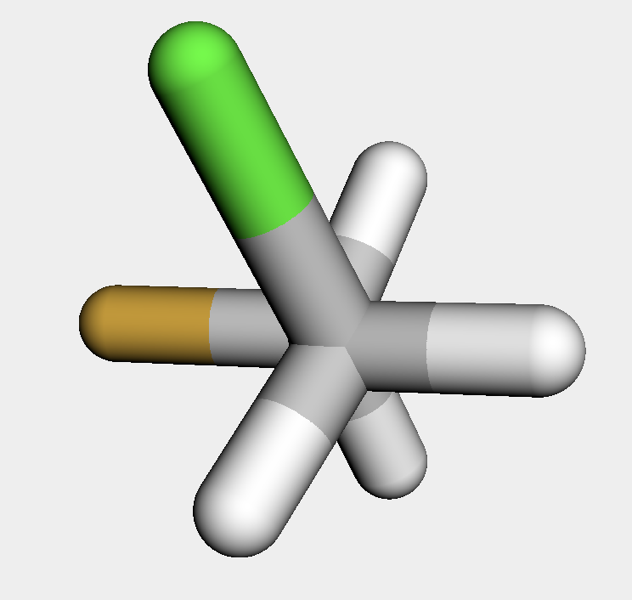
続いてこの配座の二面角をrdMolTransforms.SetDihedralDegを用いて180度に設定してみます.このメソッドはコンフォマーオブジェクトと設定する二面角の構成原子番号を引数に取ります.
atom_ids = mol_h.GetSubstructMatch(Chem.MolFromSmiles('ClCCF'))
print(atom_ids)
# (0, 1, 2, 3)
conf = mol_h.GetConformer(-1)
rdMolTransforms.SetDihedralDeg(conf, 0,1,2,3, 180)
コンフォマーの二面角が変わったことが見てとれます.
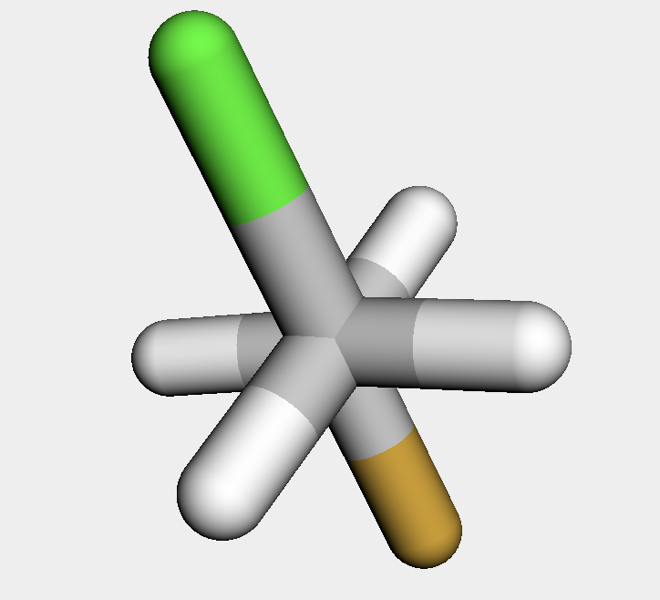
二面角に制約を課してコンフォマーを分子力学法で最適化
今度は上記の例に似ていますが,今度はMMFFで構造最適化を行う際に二面角に制約をかけてみます.このような設定は分子力場オブジェクトのAddTorsionConstraintメソッドなどで可能です.
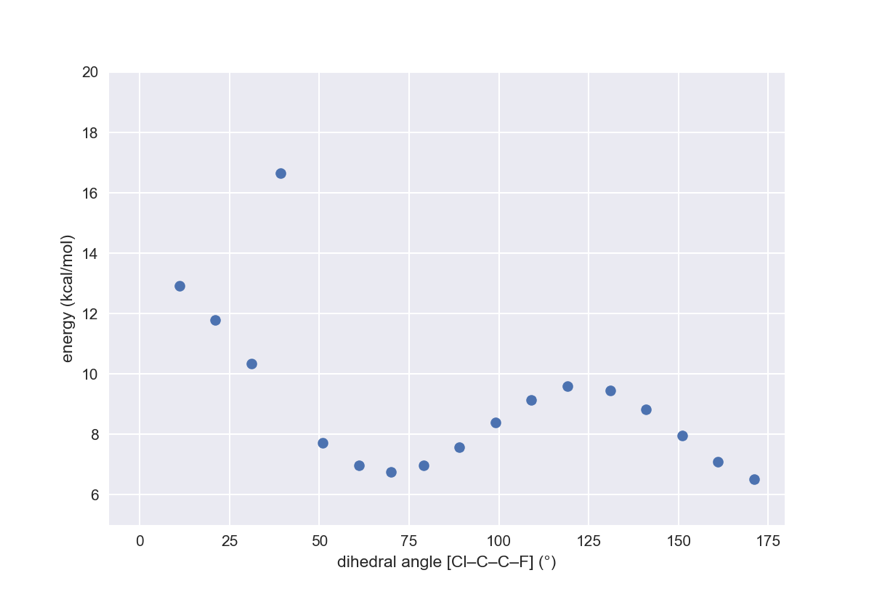
下のコードでは,
- 18個のコンフォマーを発生させる
- MMFF力場オブジェクトを作成
- 各コンフォマーの二面角を10度ずつ変えながら最適化
- 最適構造の二面角とエネルギーを取得してプロット
という順番で行っています.
# 1. コンフォマー発生
mh = AllChem.AddHs(mol)
confids = AllChem.EmbedMultipleConfs(mh, 18)
print(len(confids)) # 18
# 2,3. MMFF力場オブジェクトを作成して最適化
prop = AllChem.MMFFGetMoleculeProperties(mh)
energies = []
for confid in confids:
ff = AllChem.MMFFGetMoleculeForceField(mh, prop, confId=confid)
ang = 10*confid
ff.MMFFAddTorsionConstraint(0,1,2,3, False, ang-1., ang+1., 1e5)
ff.Minimize()
energies.append(ff.CalcEnergy())
dihedrals = [rdMolTransforms.GetDihedralDeg(conf, 0,1,2,3) for conf in mh.GetConformers()]
# 4. プロット
import matplotlib as mpl
import matplotlib.pyplot as plt
mpl.style.use('seaborn')
plt.plot(dihedrals, energies, 'o')
plt.ylim(5,20)
plt.xlabel('dihedral angle [Cl–C–C–F] (°)')
plt.ylabel('energy (kcal/mol)')
1番目と5番目のエネルギーが高くなってしまっています(無理のある構造に収束してしまった)が,あとは無難な結果になっていそうです.
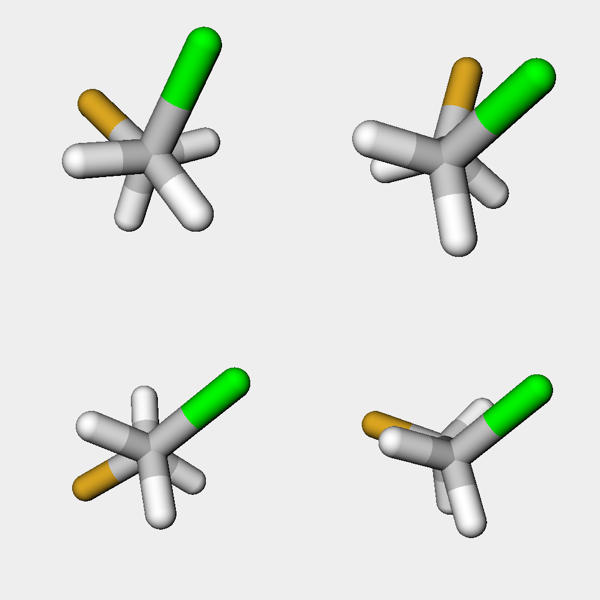
いくつか構造を取りだしてpy3Dmolを用いて表示してみましょう.
selected_confs = [3, 7, 12, 17]
v = py3Dmol.view(width=500, height=500, linked=False, viewergrid=(2,2))
for i, grid in zip(selected_confs, [(j,k) for j in range(2) for k in range(2)]):
mb = Chem.MolToMolBlock(mh, confId=i)
v.addModel(mb, 'sdf', viewer=grid)
v.setBackgroundColor('0xeeeeee')
v.setStyle({'stick': {}})
v.zoomTo()
v.show()
きちんと二面角が変化していっている様が見てとれます.
終わりに
今回は「RDKitを用いて制約付きで立体構造を生成する」という話題について,
- 鋳型を用いた立体配座の発生させる
- 立体構造の二面角を変更する
- 二面角に制約を加えながら分子力学法で構造最適化する
という3つのトピックについて簡単に見てきました.いずれの場合もシンプルな例で示してきましたが,色々と応用の利く考え方だと思います.是非ご自身の分子でも試してみてください.

コメント