数多くの化合物からライブラリーを構築する際には,
- なるべく幅広い生理活性をカバーできるように多様性に富んだ化合物で構築する
- 既に見出した生理活性化合物の近傍の化合物で構築する
といった考え方があります.前者の場合には「ケモインフォマティクスで多様なライブラリーを構築する」という記事で解説したように,化合物間の距離・類似度を基準に多様な骨格を有する化合物を選択していくことになります.
一方で,フォーカストライブラリーなどとも呼ばれる後者の場合には,リガンドベースで「活性のありそうな」部分構造を入れていくことが1つの方法になります.この際に,合成化学の観点も加味しながら,活性に影響のありそうな部分構造へと化合物を自動でフラグメント化することができれば,効率的な化合物デザインが可能になります.
今回扱うRECAP (Retrosynthetic Combinatorial Analysis Procedure)はそのような思想で設計された化合物の逆合成を提供するアルゴリズムになります.
化合物のフラグメント化とde novoデザイン
化合物をin silicoで(コンピューターを用いて)設計することを「de novoデザイン」などと呼びます.ここでは化合物のフラグメント化と分子の設計について簡単に説明します.
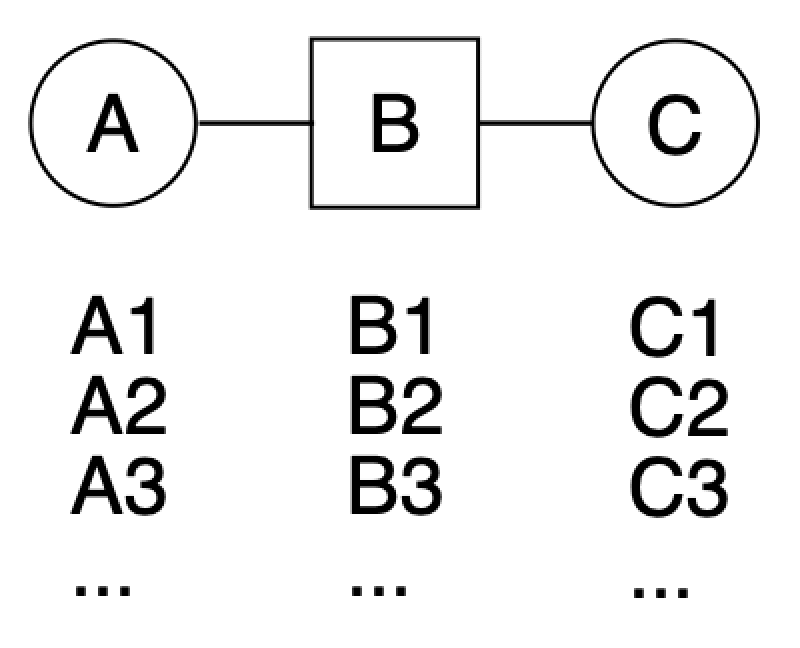
下に示すように,ある分子を「A-B-C」の部分構造へと分解したとします.それぞれのフラグメントについて,A1, A2, A3などと別の部分構造を用いることで構造的に類似した別の分子を構築することができることになります.
このこと自体は何も特別ではなく,化学者が意識せずとも行っていることになりますが,フラグメント化を化学者の眼で見て行うのではなく,あるルールに基づいて機械的に行える点にコンピュータを用いる利点があります.
RECAPとは
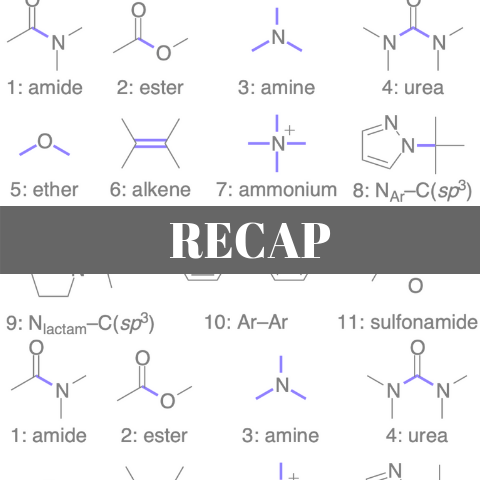
1998年に発表されたRECAPでは,生理活性物質に共通のフラグメント構造として以下に示す11の部分構造に着目して,フラグメント化を行っていきます.
いずれの部分構造においても,分子を合成する際に戦略的に逆合成を行うような結合が選ばれていることがおわかり頂けると思います.

RDKitでのRECAP実装
それではRDKitにおけるRECAPの実装について見ていきたいと思います.RECAPによるフラグメント化をRDKitで行うとツリー構造が得られます.
RECAPのツリー構造
例えばセレコキシブ(celecoxib)についてRECAPを適用してみます.下の図の青で示したように2つの切断ポイントがありますので,最初のフラグメント化では2通りのフラグメント集団が得られます.各々について1つのフラグメントはもう1度切断が可能ですので,さらに2つのフラグメントに分けられます.
RDKitでは,もととなる化合物を「parent」,フラグメントを「children」と読んでいます.また青の四角で示したようなこれ以上分解できないフラグメントを「leaf」と呼びます.Molオブジェクトに対してRECAPを施したRecap.RecapHierarchyNodeオブジェクトはこういった情報を保持しており,簡単に呼び出すことが可能です.
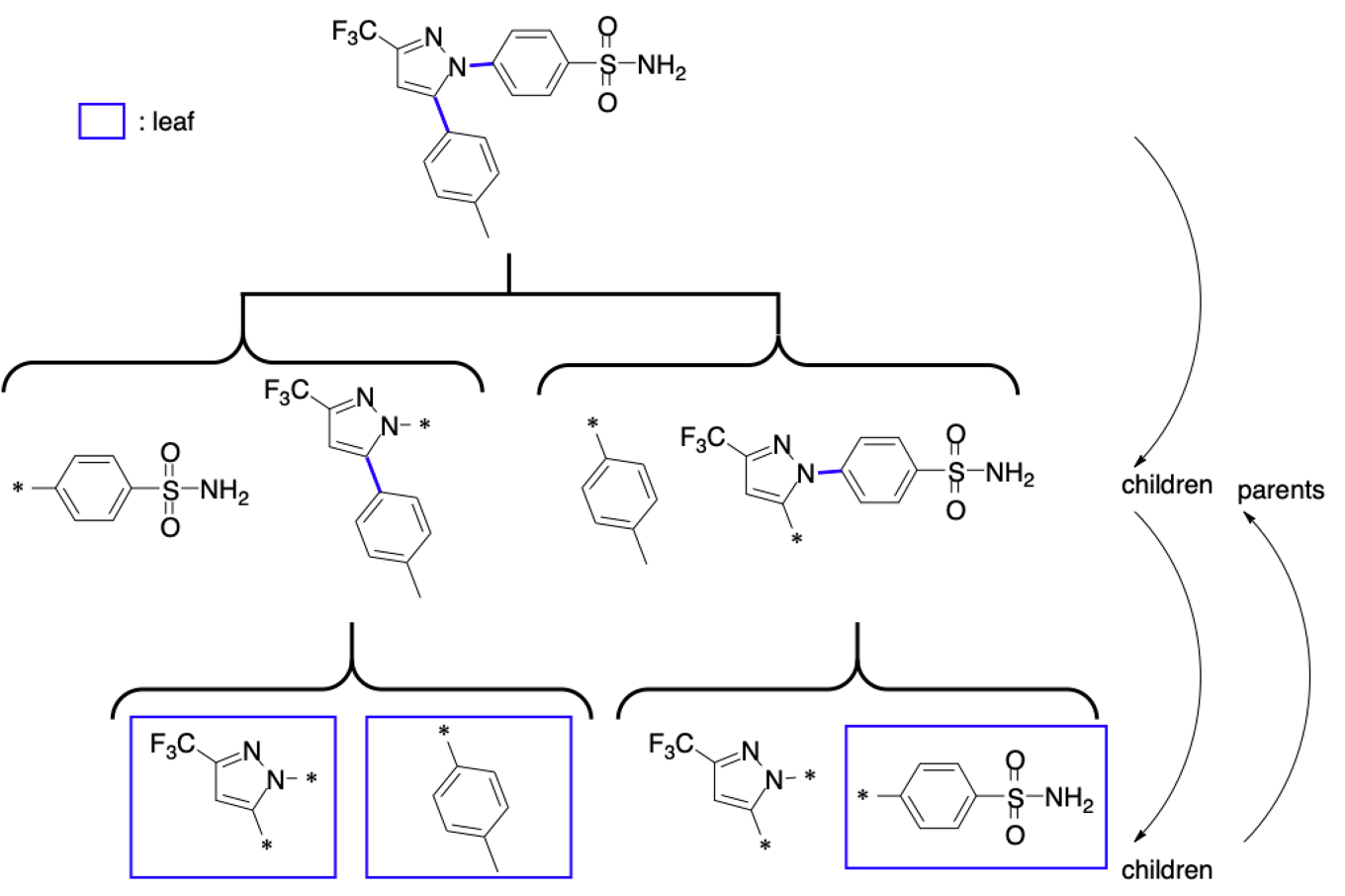
セレコキシブを用いたRECAPの具体例
それでは実際にRDKitを用いてセレコキシブのRECAPを行ってみたいと思います.以下のコードでは
- 分子情報をPubChemから取得
- SMILESからMolオブジェクトの構築
- RECAPの実行
という手順で行っています.
RECAPの実行
### ライブラリのインポート
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, Draw
import pubchempy as pcp
print(rdBase.rdkitVersion) ### 2018.09.1
### 分子の準備
celecoxib = pcp.get_compounds('celecoxib', 'name')
celecoxib = celecoxib[0]
sm = celecoxib.canonical_smiles
celecoxib = Chem.MolFromSmiles(sm)
### RECAP
decomp = Chem.Recap.RecapDecompose(celecoxib)
type(decomp)
rdkit.Chem.Recap.RecapHierarchyNode
ツリー構造の取得
RecapHierarchyNode.parents
RecapHierarchyNode.GetAllChildren()
RecapHierarchyNode.GetLeaves()
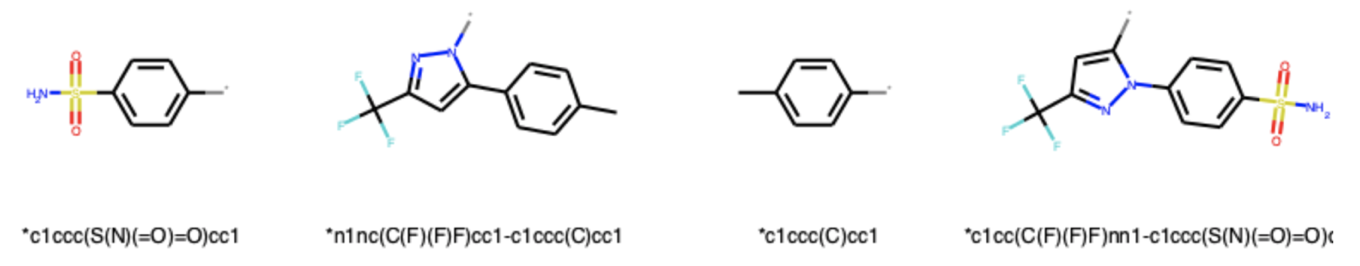
それではツリー構造から情報を取得してみます.まずは第1段階のフラグメント化で得られた分子を描画してみます.
first_gen = [node.mol for node in decomp.children.values()] Draw.MolsToGridImage(first_gen, molsPerRow=4, legends=[Chem.MolToSmiles(m) for m in first_gen])
当然ですが,上で見た図と同じフラグメントが得られています.
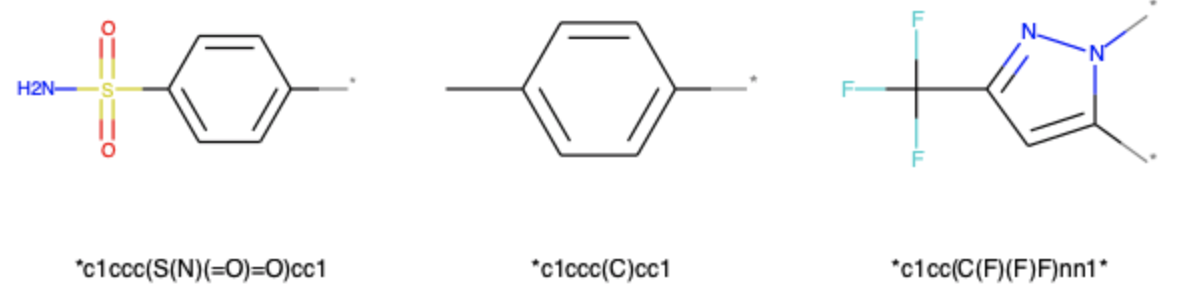
leafを得る場合にはGetLeavesメソッドを使うのが便利です.こちらも上の図で見た構造と同じものが得られます.
leaves = [leaf.mol for leaf in decomp.GetLeaves().values()] Draw.MolsToImage(leaves, legends=[Chem.MolToSmiles(m) for m in leaves])
ツリー構造の描画
最後に関数を定義することでRECAPツリーのSMILESによる描画を行ってみます.さらなるフラグメント化が不可能な場合にはRecapHierarchyNode.childrenが空の辞書を返すことを利用して,再帰的にchildrenの情報を取得することでツリーの下層へと進んでいきます.
def get_leaves(recap_decomp, n=1):
for child in recap_decomp.children.values():
print('\t'*n+child.smiles)
if child.children: ##さらなるフラグメント化のチェック
get_leaves(child, n=n+1)
def get_recap_tree(mol):
recap = Chem.Recap.RecapDecompose(mol)
print(Chem.MolToSmiles(mol))
get_leaves(recap)
get_recap_tree(celecoxib)
このようにツリー構造が表示できました.
Cc1ccc(-c2cc(C(F)(F)F)nn2-c2ccc(S(N)(=O)=O)cc2)cc1
*c1ccc(S(N)(=O)=O)cc1
*n1nc(C(F)(F)F)cc1-c1ccc(C)cc1
*c1ccc(C)cc1
*c1cc(C(F)(F)F)nn1*
*c1ccc(C)cc1
*c1cc(C(F)(F)F)nn1-c1ccc(S(N)(=O)=O)cc1
*c1cc(C(F)(F)F)nn1*
*c1ccc(S(N)(=O)=O)cc1
終わりに
今回はフォーカストライブラリー構築などに利用可能なフラグメント化のアルゴリズムとしてRECAPを扱いました.RECAPの特徴として
- 合成化学者の視点から意味のある結合でフラグメント化が行われること
- RDKitではツリー構造を形成しながらフラグメント化が行われていくこと
などを理解していただけたと思います.
次回は類似のフラグメント化のアルゴリズムとしてBRICSを扱い,今回は行わなかったフラグメント構造を用いた仮想ライブラリーの構築法なども見ていきたいと思います.
>>次の記事:「RDKitでBRICSを用いた仮想ライブラリーの構築」
コメント